Transcriptome analysis of Drosophila S2 cells after infection of Listeria monocytogenes, L. grayi or L. welshimeri
-
摘要:目的
利用果蝇作为宿主比较单增李斯特菌Listeria monocytogenes、格氏李斯特菌L. grayi以及威尔斯李斯特菌L. welshimeri侵染后触发果蝇S2细胞基因表达模式的异同,为探究宿主的先天免疫防御机制以及不同种李斯特菌的致病差异机制提供一定的研究基础。
方法单增李斯特菌、格氏李斯特菌以及威尔斯李斯特菌分别侵染果蝇S2细胞3 h后,利用转录组测序技术检测果蝇S2细胞中的mRNA表达谱,并利用生物信息学方法筛选出差异表达基因(DEGs),然后对这些基因进行GO注释和KEGG分析,最后采用qRT-PCR技术验证5个Toll和Imd通路相关的抗菌肽基因的转录水平。
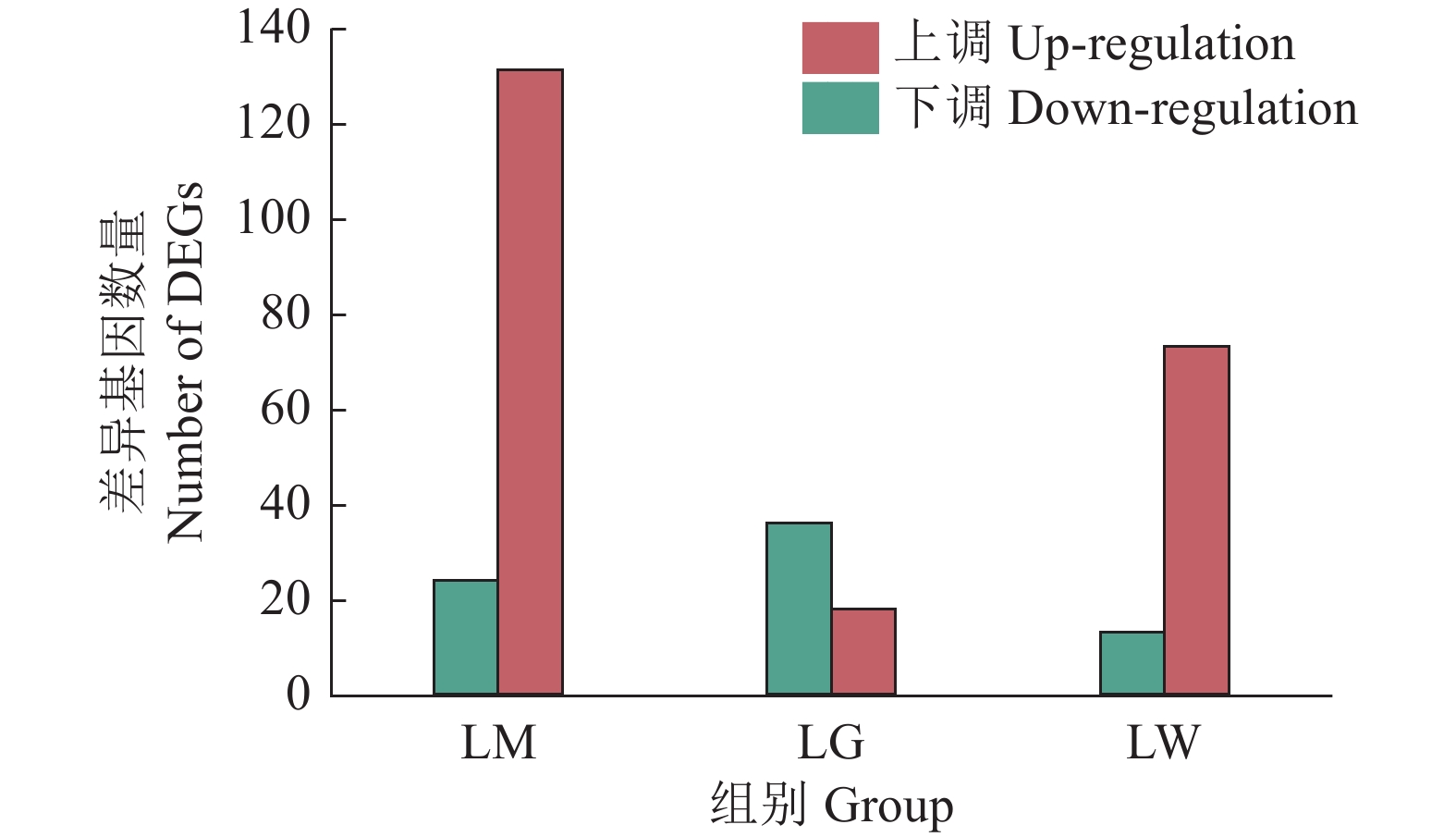
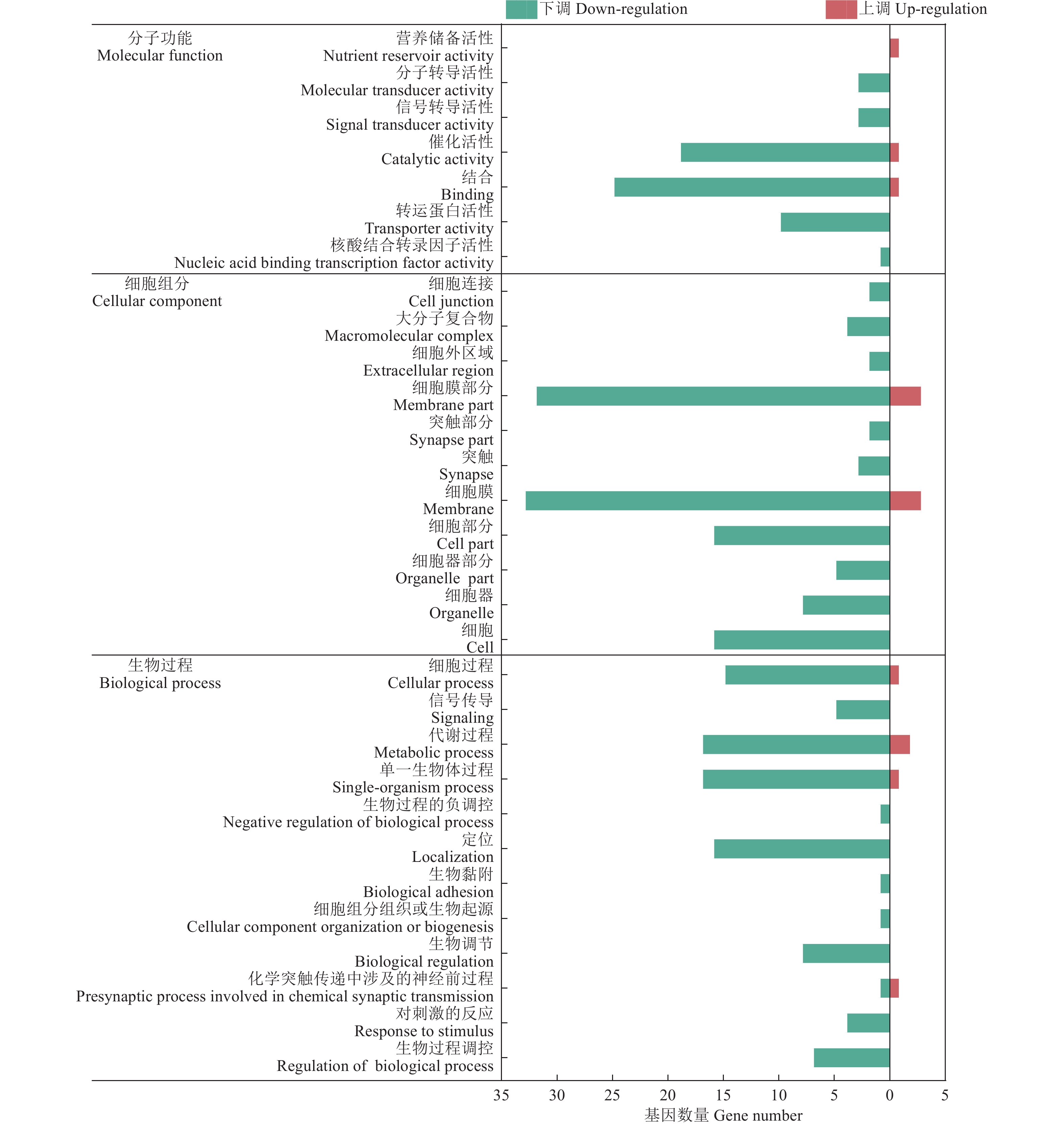
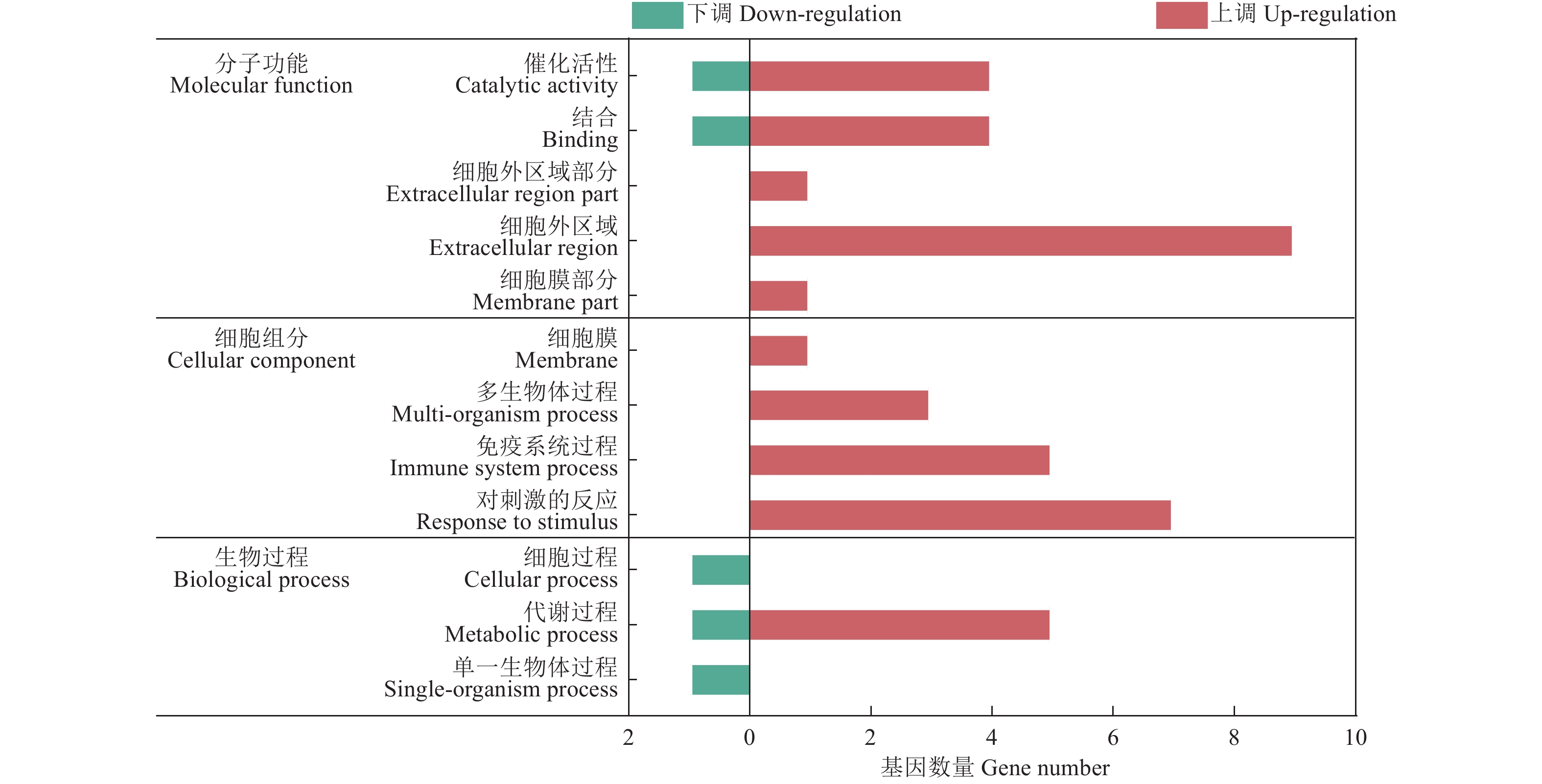
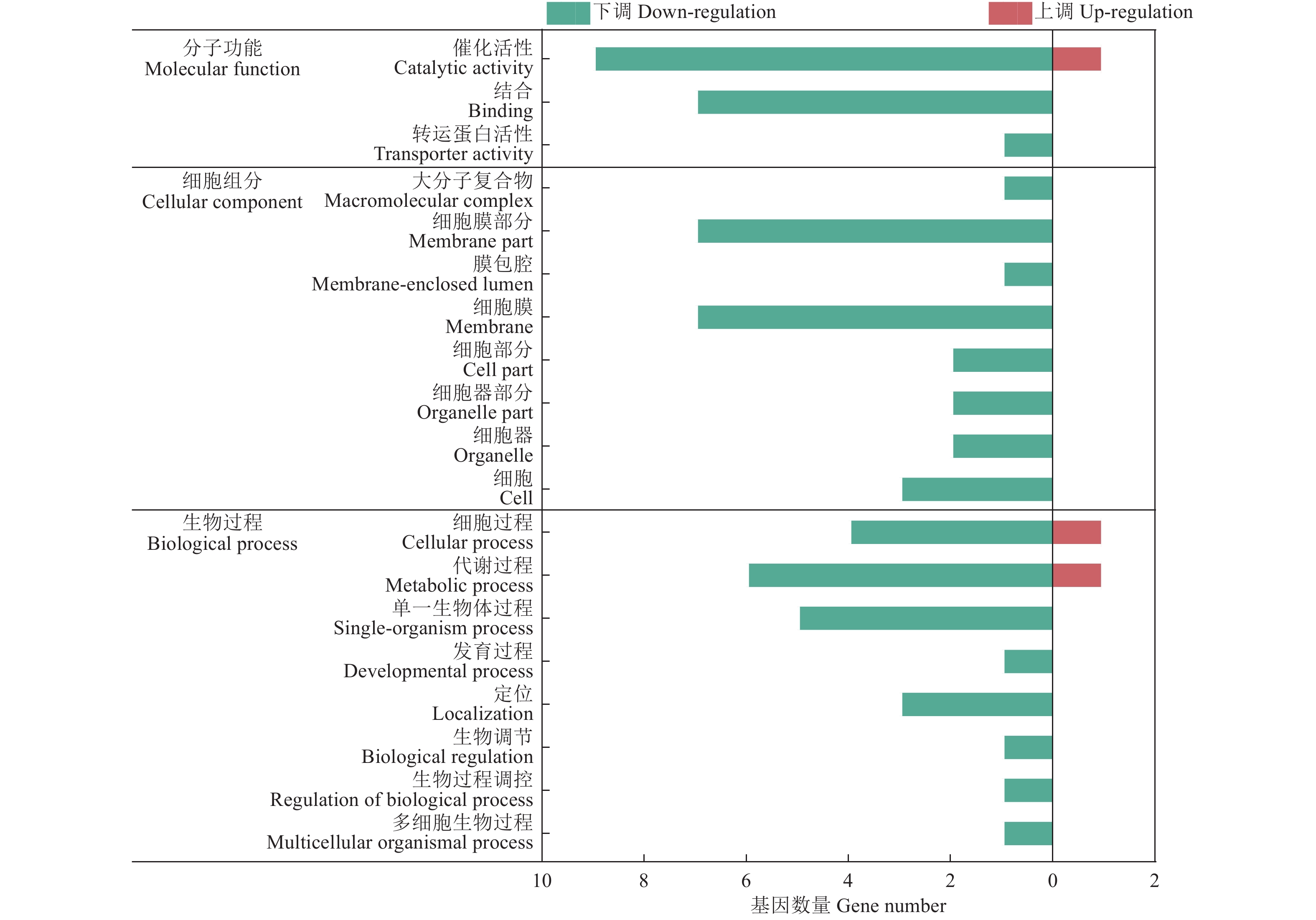
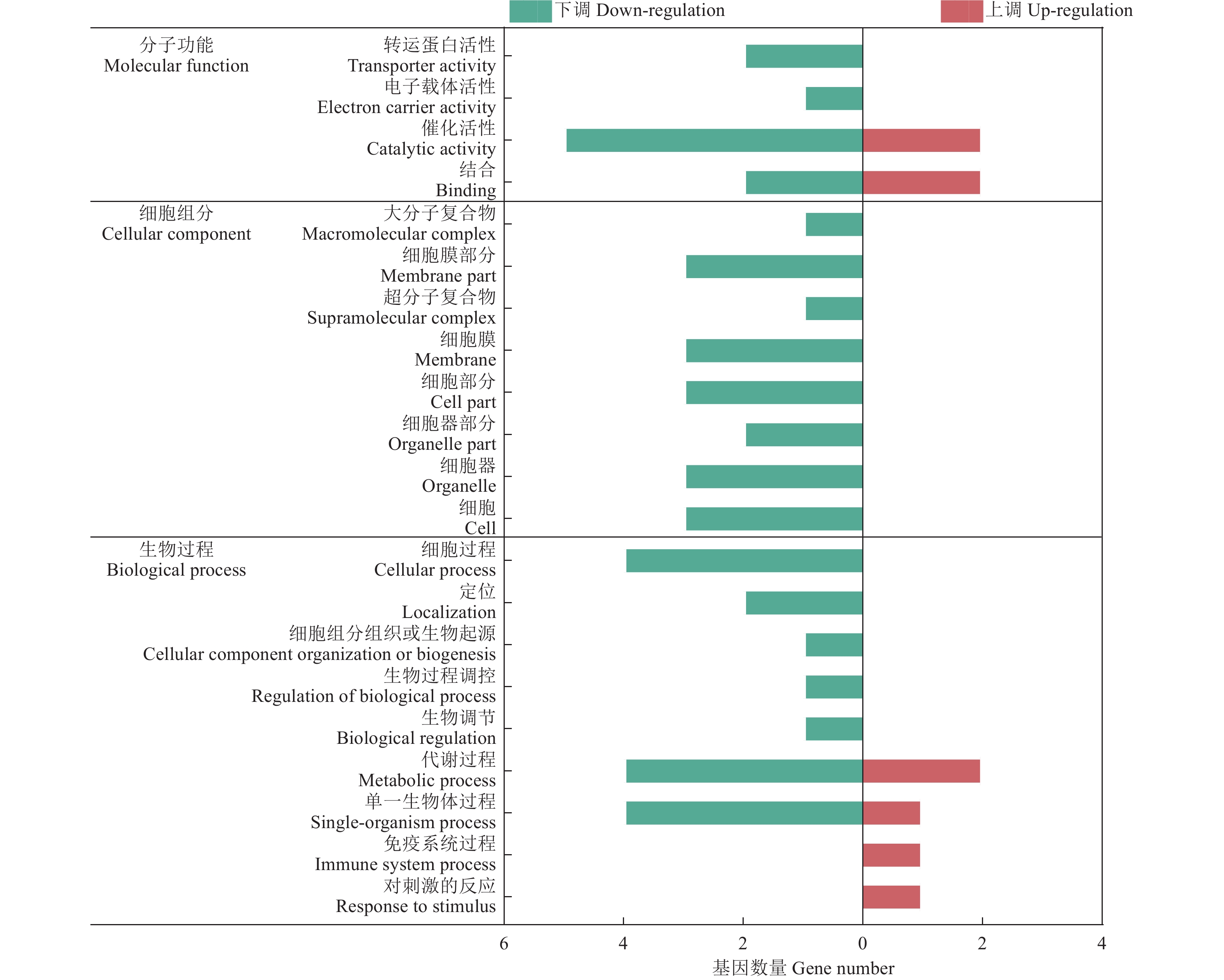
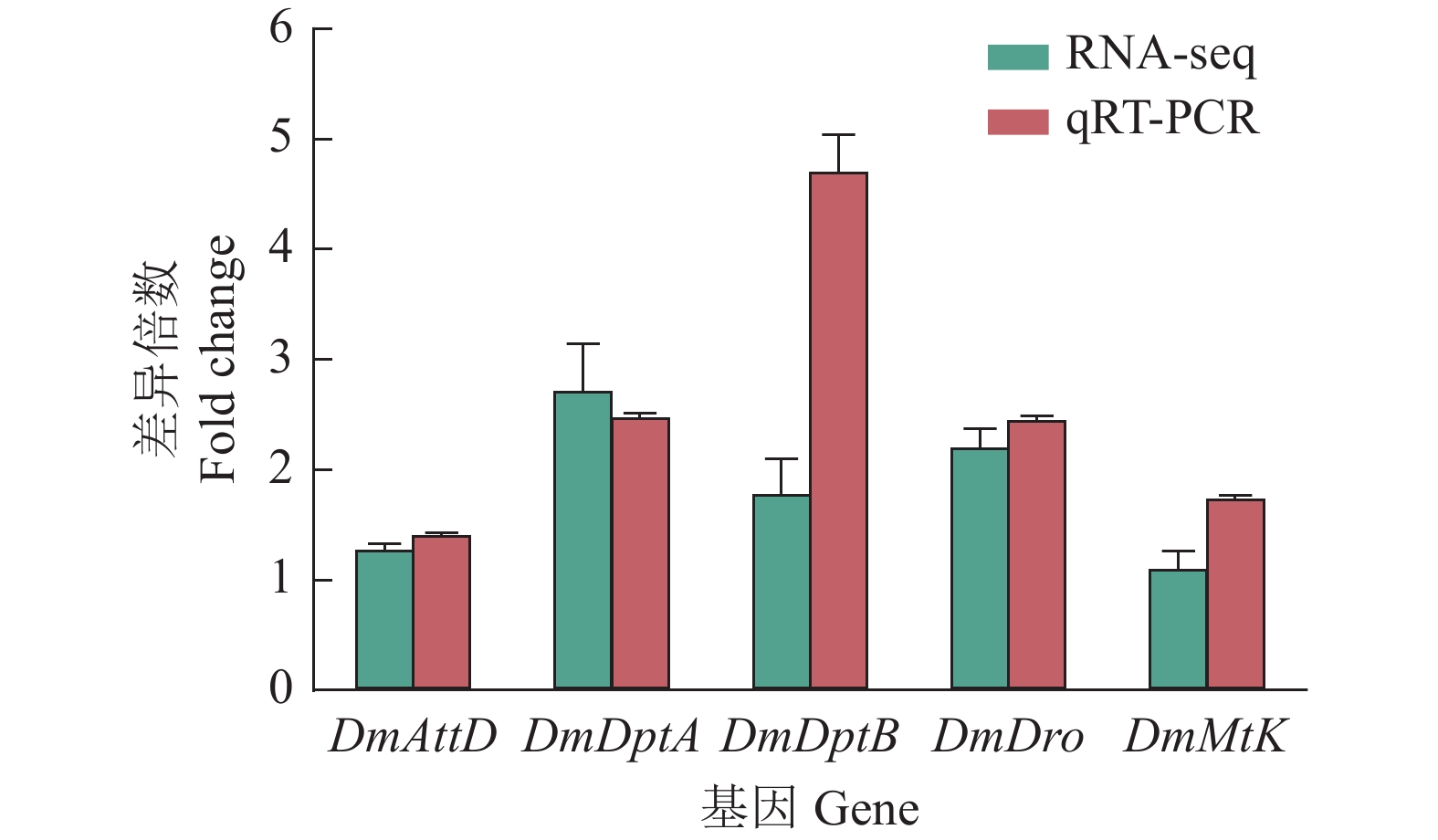
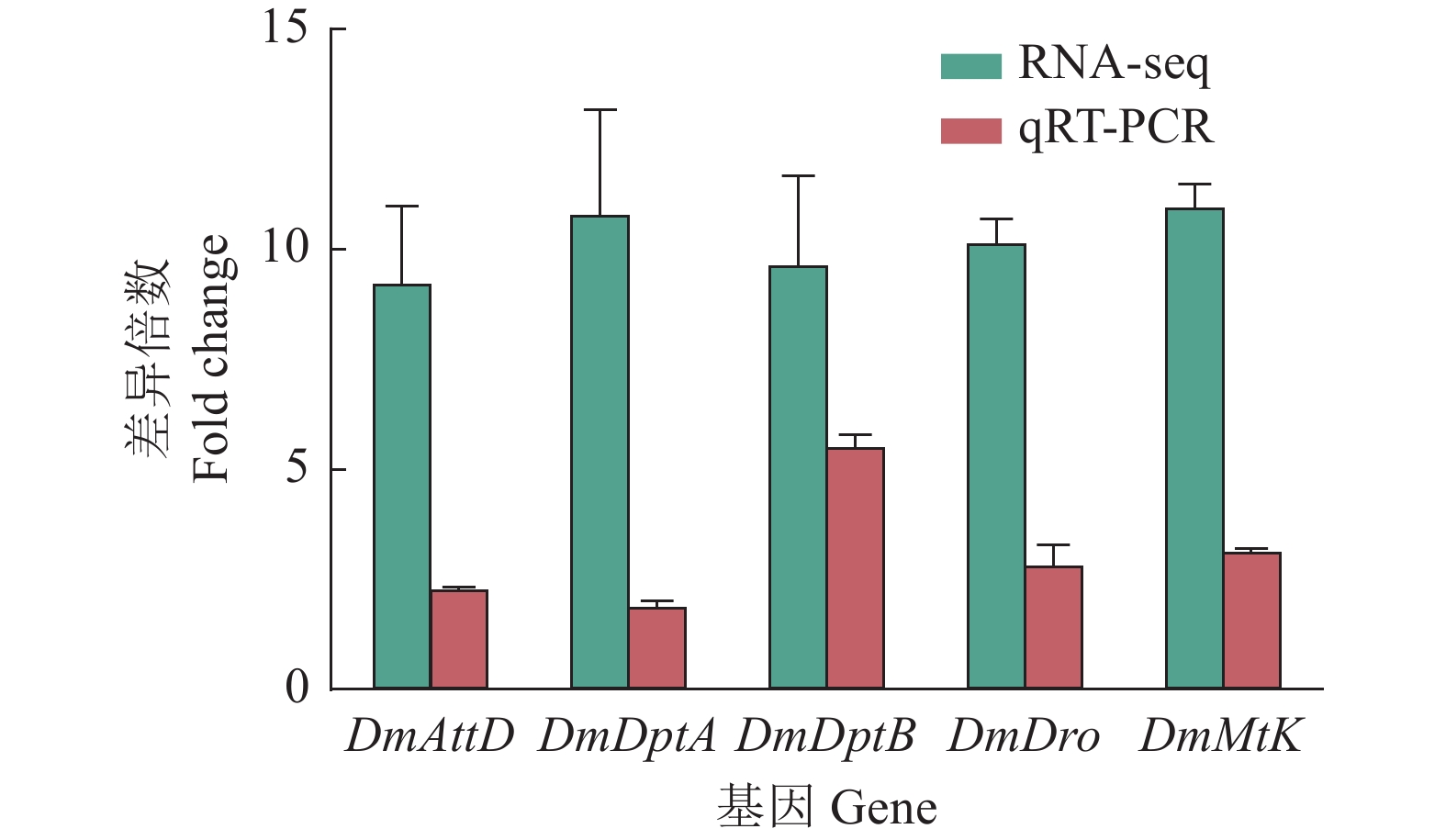
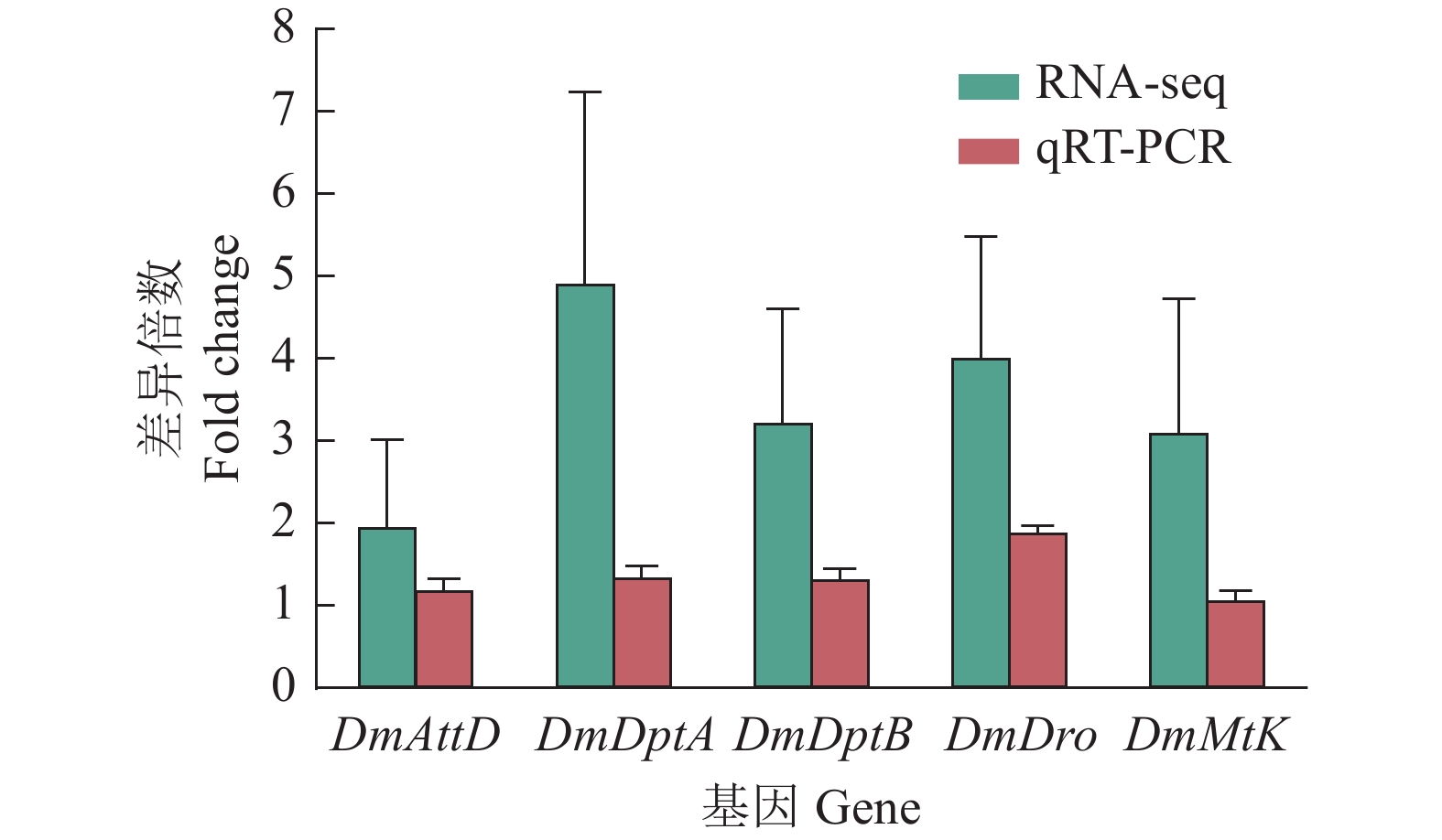
结果单增李斯特菌、格氏李斯特菌和威尔斯李斯特菌侵染3 h后果蝇S2细胞中共有18个DEGs,特有DEGs分别为104、28和33个。3种李斯特菌侵染组共有的DEGs被注释到代谢过程、细胞过程、细胞器、对刺激反应和免疫系统过程等共有的GO条目;此外,单增李斯特菌侵染组特有的DEGs被注释到生物黏附、参与化学突触传递的突触前过程、生物过程的负调节、核酸结合转录因子活性和信号转导器活性等特有GO条目;格氏李斯特菌侵染组特有的DEGs被注释到多个生物体过程、细胞外区域等特有GO条目;威尔斯李斯特菌侵染组特有的DEGs则被注释到发育过程及膜包裹腔等特有GO条目。KEGG分析结果显示,3种李斯特菌侵染组的DEGs均富集到了Toll/Imd信号通路。qRT-PCR 验证5个Toll和Imd通路相关的抗菌肽基因转录水平,定量结果与测序结果基本吻合。
结论3种不同李斯特菌侵染后触发S2细胞的转录表达模式具有一定的相似性,但同时也存在不同之处,这些差异存在的信号通路可能与3种李斯特菌在S2细胞中的致病力强弱存在相关性。
Abstract:ObjectiveTo compare the gene expression patterns in S2 cells after infection of Listeria monocytogenes, L. grayi, and L. welshimeri using Drosophila as a host, and provide a research basis for exploring the innate immune defense mechanisms of the host and the differential pathogenic mechanisms of different Listeria strains.
MethodAfter infection of L. monocytogenes, L. grayi or L. welshimeri for 3 h, the mRNA expression profiles in Drosophila S2 cells were detected using transcriptome sequencing technology. Analysis of differentially expressed genes (DEGs) was performed using bioinformatics tools of GO annotation and KEGG analysis. Finally, the mRNA levels of five antimicrobial peptide genes associated with the Toll and Imd signaling pathways were validated using qRT-PCR technology.
ResultAfter infection of L. monocytogenes, L. gray or L. welshimeri, there were 18 DEGs in common, as well as 104, 28, and 33 unique DEGs in the Drosophila S2 cells respectively. The DEGs commonly existed in three Listeria infected groups were annotated in GO terms including metabolic process, cellular process, organelle, response to stimulus, immune system process and etc. The unique DEGs existed in L. monocytogenes infected group were annotated in GO terms such as biological adhesion, presynaptic process involved in chemical synaptic transmission, negative regulation of biological process, nucleic acid binding transcription factor activity, signal transducer activity, and etc. The unique DEGs in L. grayi infected group were annotated in GO terms such as multi-organism process and extracellular regions, while the unique DEGs in L. welshimeri infected group were annotated in GO terms such as developmental processes and membrane-enclosed lumen. The KEGG analysis results showed that the DEGs of three Listeria infected groups were enriched in the Toll/Imd signaling pathways. The quantitative results of qRT-PCR validation of five antimicrobial peptide genes related to Toll and Imd signaling pathways were consistent with the transcriptomic results.
ConclusionThe expression patterns in S2 cells affected by the infection of these three Listeria species exhibited some similarities but also differences. The variation of the signaling pathways may be related to the virulence of these Listeria species in S2 cells.
-
山羊Capra hircus繁殖力影响养羊业的经济效应,受遗传、营养、管理水平等因素影响,是复杂的数量性状。母羊的产羔数是山羊繁殖性能之一,由排卵数决定,主要受遗传因素影响,与卵泡发育息息相关[1]。卵泡发育是一个极其复杂的过程,在发育过程中绝大多数卵泡会闭锁,只有少数卵泡能成熟并排卵[2-3]。
目前,关于整个卵巢的研究越来越多[4-5],但卵巢作为一个动态发育的生殖器官,包含不同发育阶段的卵泡,如原始卵泡、生长卵泡、成熟卵泡等。因此,在研究卵巢时,有必要对其细分后再开展研究。Terenina等[6]对猪卵巢健康小卵泡和闭锁小卵泡转录组的研究发现CCDC80、GADD45A、ZNF628等基因在闭锁小卵泡中的表达量超过健康小卵泡5倍,认为可能是卵泡闭锁的标志基因。Zhang等[7]在猪健康卵泡和早期闭锁卵泡的转录组研究中发现450个显著差异表达基因。李鹏飞等[8-9]对牛的优势卵泡和从属卵泡进行转录组测序发现差异显著的PRSS35、PTGRF和ARID4B等基因与卵泡发育相关。Hatzirodos等[10]在牛健康大卵泡和健康小卵泡的转录组研究中发现,在大卵泡中STAT4和XBP1基因表达上调,MGEA5基因下调;而KIT、IHH和MEST基因在小卵泡中最为活跃。吴阳升等[11]通过分析绵羊小卵泡和中卵泡转录组发现,在中卵泡表达上调的基因有IGFBP1、INHBA和TIMP1等,下调的有C-FOS、EGR1、FOSB等。综上,猪、牛和绵羊不同发育阶段卵泡的基因表达模式不同,然而山羊的卵泡转录组研究以及卵巢基质和卵泡之间的基因表达模式鲜见报道。
川中黑山羊是我国优秀的地方山羊品种[12],经过长期的自然和人工选择,繁殖性能突出,遗传性能稳定,母羊平均产羔率为236.78%,羔羊成活率为91%[13]。目前对川中黑山羊繁殖性能的研究较少。转录组包括编码RNA和非编码RNA,指在某一特定条件下细胞或组织内所有转录产物的总和,从整体水平研究基因结构和功能[14]。转录组测序是通过高通量测序技术,对生物体全部基因的测序,反映生命体处于不同环境条件下的不同基因表达水平及调控模式[15]。本研究以发情期的川中黑山羊为研究对象,应用高通量测序技术对卵巢基质、大卵泡和小卵泡进行转录组测序,比较分析三者的转录水平差异,探索卵巢基质、大卵泡和小卵泡间的mRNA表达模式。旨在从转录组水平揭示卵巢不同部位的遗传差异,丰富山羊卵巢基质、大卵泡和小卵泡的转录组数据信息,筛选出的基因可为山羊卵泡发育提供一定的基因资源和新的研究方向。
1. 材料与方法
1.1 试验动物和样品采集
选用南方某种羊场10头健康的川中黑山羊母羊,年龄3.5~4.5岁。采用前列腺素1次注射法对每只母羊注射氯前列烯醇0.1 mg,24 h后第1次发情,18 d后确认第2次发情,采用公羊试情法确认母羊是否发情,以母羊摇尾、站立并接受爬跨视为发情[16-19]。发情后24 h内进行屠宰并采集卵巢。
卵巢用PBS缓冲液洗3次,分离小卵泡(Small follicle,SF,d<3 mm)、大卵泡(Large follicle,LF,d>1 cm)和卵巢基质,各6个重复组,其中1个小卵泡样本包含8~10个小卵泡,1个大卵泡样本仅包含1个大卵泡,连同分离后的卵巢基质放入冻存管,立即置于液氮中,带回实验室于−80 ℃冰箱保存,用于后续试验。试验所得的卵巢基质样品中有少量d<3 mm的小卵泡,所有d>1 cm的大卵泡已分离出来。
1.2 总RNA抽提、cDNA文库构建和转录组测序
将卵巢基质、大卵泡和小卵泡完全研磨,用Trizol试剂提取总RNA(Invitrogen,美国)。通过DanoDrop ND-2000分光度计(Thermo Science,美国)、10 g/L琼脂糖凝胶分别检测RNA的浓度和纯度、完整性。每个样品取3 μL总RNA,构建cDNA文库,利用Illumina HiSeq测序平台进行双末端测序。整个流程委托上海派森诺生物科技股份公司完成。
1.3 转录组数据分析
对测序数据采用Cutadapt软件去除3′端的接头,去除的部分与接头有至少10 bp重叠(AGATCGGAAG),允许20%碱基错配;去除平均占比低于Q20(碱基识别准确率在99%以上的碱基所占百分比)的Reads。通过Bowtie2软件(GCF_001704415.1_ARS1_genomic; http://www.ensembl.org/)建立参考基因组索引,使用Tophat2软件的microexon-search和library-type=fr-firststrand参数将过滤后的Reads比对到参考基因组上,默认Reads和参考基因组序列的碱基错配在2个之内即为比对成功。采用R语言DESeq程序包对差异基因进行筛选,条件为:|log2表达差异倍数|>1,显著性P<0.05,至少有4个样品的FPKM(每百万Reads中来自某一基因每千碱基长度的Reads数目,Fragments per kilobase per million of reads)大于1。通过R语言Pheatmap软件包对所有基因和样品进行双向聚类分析。
1.4 差异基因功能注释
利用Gene ontology(GO)数据库对所有差异基因进行注释,通过Kyoto encyclopedia of genes and genomes(KEGG)数据库对差异基因进行通路分析,采用超几何分布计算差异基因显著富集的GO条目和KEGG通路,P<0.05为显著富集。
1.5 qRT-PCR验证分析
从差异基因中随机选择5个基因,利用qRT-PCR进行转录组表达谱验证分析,PCR反应体系10 μL:5 μL SYBR® Green Master Mix,上下游引物各0.5 μL,1 μL cDNA和3 μL ddH2O。反应条件:95 ℃,1 min;95 ℃,30 s,58 ℃,30 s,72 ℃,1 min,34个循环。引物设计参考NCBI的山羊相关基因序列,β-actin作为内参基因,用Primer 5.0软件设计,并送Sangon Biotech合成引物(表1)。
表 1 用于qRT-PCR检测的基因引物Table 1. Gene primers for qRT-PCR detection基因
Gene引物序列(5′→3′)
Primer sequence扩增长度/bp
Product lengthCYP19A1 F: CCGAAGTTGTGCCTATTGC
R: GCTGGGACCTGGTATTGAG101 INHBA F: CATGTGGGAAAAGTGGGGGA
R: TCAAAGTGCAGCGTCTTCCT146 KITLG F: CATTTATCTTCAACTGCTCCTA
R: CCACCATCTCGCTTATCC191 SERP1 F: GTCTTCCTGTGCTCGCTCTTC
R: CGATTATGGCTTCCGATTTCA268 GPNMB F: CCTTGTCCTTTGCCTTCAC
R: AAATCCACCAGGGAGTCGT230 β-actin F: TGCTTCTAGGCGGACTGATT
R: TACAATCAAAGTCCTCGGCCAC106 1.6 数据统计分析
试验数据用SPSS 22.0统计软件分析,通过独立样本t检验来进行差异显著性分析。
2. 结果与分析
2.1 测序数据质控
将本研究所得的测序数据经过质量控制,对Clean reads进行碱基含量分布和碱基质量分布统计,Q30(碱基识别准确率在99.9%以上的碱基所占百分比)在91%以上,测序数据与山羊的数据库序列相似性在83%以上(表2)。综合表明测序数据质量好,满足后续分析的要求。
表 2 测序数据质控Table 2. Quality control of sequencing data样品1)
Sample有效测序量
No. of clean reads有效测序的占比/%
Ratio of clean readsQ302)/% 样品1)
Sample有效测序量
No. of clean reads有效测序的占比/%
Ratio of clean readsQ302)/% O1 105 953 656 99.68 94.28 LF1 104 768 836 99.62 94.23 O2 102 197 046 99.34 92.71 LF2 100 396 168 99.70 94.85 O3 105 678 158 99.41 92.86 LF3 104 879 676 99.48 94.15 O4 101 844 278 99.44 92.86 LF4 102 209 432 99.37 92.57 O5 107 096 856 99.32 91.31 LF5 100 346 902 99.75 93.86 O6 104 231 700 99.57 92.09 LF6 101 449 700 99.50 92.26 SF1 102 014 414 99.69 94.88 SF4 105 756 846 99.19 91.52 SF2 102 993 890 99.45 94.24 SF5 103 645 956 99.58 93.79 SF3 106 653 668 99.77 94.11 SF6 106 529 902 99.45 92.38 1) O:卵巢基质;LF:大卵泡;SF:小卵泡;2) 碱基识别准确率在99.9%以上的碱基所占百分比
1) O: Ovarian stroma; LF: Large follicle; SF: Small follicle; 2) Percentage of bases with base recognition accuracy above 99.9%2.2 差异基因筛选
测序结果分析表明,卵巢基质和大卵泡之间显著差异表达的基因总数为524个,其中显著上调的有238个,显著下调的有286个;卵巢基质和小卵泡之间显著差异表达的基因总数为180个,其中显著上调的有25个,显著下调的有155个;大卵泡和小卵泡之间显著差异表达的基因总数为403个,其中显著上调的有281个,显著下调的有122个。结合近年哺乳动物中与卵泡发育相关的基因的报道[6, 20-33],我们发现本试验中15个差异表达的基因可能与卵泡发育相关,包括INHA、INHBA、CYP19A1、MGST1、BEX2、APOA1、APOA2、MMP15、CITED1、PRSS23、LRP8、ADAMTS1、SPARCL1、TNFRSF1B和TNFRSF19。按照P值大小筛选排名前10的差异表达基因,再根据差异倍数从大到小进行排序[34-35],结果如表3所示。通过聚类图(图1)发现,卵巢基质、大卵泡和小卵泡各样本的基因分别聚类在一起,且同一组几个不同样本聚类在同一个簇中。此外,卵巢基质与小卵泡间差异基因的数量最少,且大部分基因表达量相似(图1),推测小卵泡和卵巢基质间的表达模式较为相似。大卵泡与卵巢基质、小卵泡的差异表达基因数量较多,且表达量相差较大,推测大卵泡与卵巢基质、小卵泡的基因表达模式差异较大。
表 3 不同卵巢部位间排名前10位的差异表达基因Table 3. Top ten differentially expressed genes among different parts of ovary项目1)
Item基因
Genelog2(FC)2) P 项目1)
Item基因
Genelog2(FC)2) P 项目1)
Item基因
Genelog2(FC)2) P O vs LF EVI2A 1.69 3.45×10−6 O vs SF KCTD12 1.57 1.52×10−3 LF vs SF GLI1 2.13 1.22×10−6 IRF2BP2 1.24 5.84×10−7 CFD 1.43 3.02×10−4 SLC25A34 1.57 5.72×10−6 PNRC1 1.16 8.06×10−6 PTCH2 −2.23 2.59×10−3 PRRT2 1.35 2.55×10−5 GPR21 1.06 6.22×10−6 GDF5 −1.84 1.99×10−3 PPM1J 1.34 3.08×10−5 TMEM176B −1.86 1.27×10−5 GLI1 −1.48 3.80×10−4 TNRC6C 1.24 2.21×10−5 RDH11 −1.50 1.84×10−6 EMID1 −1.32 4.01×10−4 EFNB3 1.10 2.00×10−5 MINOS1 −1.37 6.17×10−6 MYCL −1.30 3.35×10−4 EFS 1.07 3.58×10−5 MPDU1 −1.09 1.09×10−5 DPEP3 −1.26 1.47×10−3 ADGRL1 1.04 1.63×10−5 NME1 −1.06 1.01×10−5 DPEP2 −1.23 1.68×10−3 KITLG −1.85 1.34×10−5 ARL2BP −1.05 3.12×10−6 CNRIP1 −1.20 1.81×10−3 TMEM176B −1.49 3.67×10−5 1) O:卵巢基质;LF:大卵泡;SF:小卵泡;2) FC:表达差异倍数
1) O: Ovarian stroma; LF: Large follicle; SF: Small follicle; 2) FC: Expression fold-change2.3 共有的差异表达基因
在卵巢基质vs大卵泡、卵巢基质vs小卵泡、小卵泡vs大卵泡这3组中有4个共有的差异表达基因;卵巢基质vs大卵泡、卵巢基质vs小卵泡之间有73个共有的差异表达基因;卵巢基质vs大卵泡、小卵泡vs大卵泡之间有186个共有的差异表达基因;卵巢基质vs小卵泡、小卵泡vs大卵泡之间有29个共有的差异表达基因。
2.4 差异表达基因的生物学功能分析
基于GO数据库,对差异基因进行功能分类,各功能分类下差异前10位的GO条目如表4、表5和表6所示。在卵巢基质vs大卵泡(表4)中有15个GO条目显著富集(P<0.05),包括催化活性、细胞质、细胞膜、细胞内膜结合细胞器、转移酶活性等。在卵巢基质vs小卵泡(表5)中有25个GO条目显著富集(P<0.05),包括小分子结合、碳水化合物衍生物结合、核苷酸结合、磷酸核苷结合、嘌呤核苷三磷酸结合等。在小卵泡vs大卵泡(表6)中有64个GO条目显著富集(P<0.05),包括细胞过程、细胞内部、线粒体部分、嘌呤核苷酸结合等。
表 4 卵巢基质vs大卵泡部分差异表达基因的GO功能分类Table 4. GO functional classification of partial differentially expressed genes in ovarian stroma vs large follicle分类
Category条目
Term基因数目
Gene countP 生物学过程 Biological process RNA加工过程 RNA processing 5 2.70×10−2 细胞组分 Cellular component 细胞 Cell 66 1.90×10−2 细胞部分 Cell part 65 2.40×10−2 细胞内部 Intracellular 63 1.70×10−3 细胞内的部分 Intracellular part 55 6.00×10−3 细胞质 Cytoplasm 39 9.80×10−4 细胞内膜结合细胞器 Intracellular membrane-bounded organelle 36 2.50×10−2 膜结合细胞器 Membrane-bounded organelle 36 2.50×10−2 细胞质部分 Cytoplasmic part 28 2.80×10−2 分子功能 Molecular function 催化活性 Catalytic activity 51 8.20×10−4 磷酸核苷结合 Nucleotide phosphate binding 22 4.60×10−2 核苷酸结合 Nucleotide binding 22 4.60×10−2 转移酶活性 Transferase activity 21 1.50×10−2 转移酶活性(转移含磷基团)
Transferase activity (Transferring phosphorus-containing groups)12 2.70×10−2 异构酶活性 Isomerase activity 5 2.00×10−2 表 5 卵巢基质vs小卵泡部分差异表达基因的GO功能分类Table 5. GO functional classification of partial differentially expressed genes in ovarian stroma vs small follicle分类
Category条目
Term基因数目
Gene countP 生物学过程 Biological process 初级代谢过程 Primary metabolic process 13 4.70×10−2 细胞对应激的反应 Cellular response to stress 4 4.40×10−2 分子功能 Molecular function 小分子结合 Small molecule binding 12 2.30×10−4 碳水化合物衍生物结合 Carbohydrate derivative binding 11 2.50×10−4 核苷酸结合 Nucleotide binding 11 5.90×10−4 磷酸核苷结合 Nucleoside phosphate binding 11 5.90×10−4 核糖核苷结合 Ribonucleoside binding 10 6.00×10−4 核苷结合 Nucleoside binding 10 6.00×10−4 嘌呤核苷三磷酸结合 Purine ribonucleoside triphosphate binding 10 6.00×10−4 嘌呤核苷结合 Purine nucleoside binding 10 6.00×10−4 嘌呤核糖核苷结合 Purine ribonucleoside binding 10 6.00×10−4 核糖核苷酸结合 Ribonucleotide binding 10 6.70×10−4 表 6 小卵泡vs大卵泡部分差异表达基因的GO功能分类Table 6. GO functional classification of partial differentially expressed genes in small follicle vs large follicle分类
Category条目
Term基因数目
Gene countP 生物学过程
Biological process细胞进程 Cellular process 73 3.40×10−2 有机环状化合物代谢过程 Organic cyclic compound metabolic process 31 1.70×10−2 有机氮化合物代谢过程 Organonitrogen compound metabolic process 17 3.80×10−2 有机磷代谢过程 Organophosphate metabolic process 10 1.60×10−2 含核苷的小分子代谢过程 Nucleobase-containing small molecule metabolic process 9 3.50×10−2 嘌呤核糖核苷代谢过程 Purine ribonucleoside metabolic process 8 2.00×10−2 嘌呤核苷代谢过程 Purine nucleoside metabolic process 8 2.00×10−2 核糖核苷代谢过程 Ribonucleoside metabolic process 8 2.00×10−2 糖基化合物代谢过程 Glycosyl compound metabolic process 8 2.90×10−2 核苷代谢过程 Nucleoside metabolic process 8 2.90×10−2 细胞组分
Cellular component细胞内 Intracellular 64 2.70×10−3 细胞质 Cytoplasm 35 2.80×10−2 细胞质部分 Cytoplasmic part 29 2.30×10−2 线粒体 Mitochondrion 14 9.20×10−4 线粒体部分 Mitochondrial part 10 5.20×10−3 细胞器膜 Organelle membrane 10 2.30×10−2 线粒体膜 Mitochondrial membrane 9 5.70×10−3 细胞器包膜 Organelle envelope 9 9.00×10−3 线粒体包膜 Mitochondrial envelope 9 9.00×10−3 囊膜 Envelope 9 9.00×10−3 分子功能
Molecular function磷酸核苷结合 Nucleoside phosphate binding 28 2.90×10−3 核苷酸结合 Nucleotide binding 28 2.90×10−3 嘌呤核苷酸结合 Purine nucleotide binding 25 2.10×10−3 嘌呤核苷结合 Purine nucleoside binding 24 3.30×10−3 嘌呤核苷三磷酸结合 Purine ribonucleoside triphosphate binding 24 3.30×10−3 嘌呤核糖核苷结合 Purine ribonucleoside binding 24 3.30×10−3 核苷结合 Nucleoside binding 24 3.30×10−3 核糖核苷结合 Ribonucleoside binding 24 3.30×10−3 嘌呤核糖核苷酸结合 Purine ribonucleotide binding 24 4.10×10−3 基于KEGG数据库的通路富集分析(各组对比项目中前10位的信号通路见表7),在卵巢基质vs大卵泡中差异表达基因涉及55条通路,显著富集的通路有46个(P<0.05),包括氧化磷酸化、代谢途径、内质网蛋白质加工、柠檬酸循环、类固醇生物合成等。在卵巢基质vs小卵泡中差异表达基因涉及27条通路,显著富集的通路有17个(P<0.05),包括细胞周期、错配修复、DNA复制、卵母细胞减数分裂、RNA转运、孕酮介导的卵母细胞成熟等。在小卵泡vs大卵泡中差异表达基因涉及42条通路,显著富集的通路有30个(P<0.05),包括氧化磷酸化、蛋白酶体、内质网蛋白质加工等。
表 7 卵巢基质、大卵泡和小卵泡部分差异表达基因的KEGG代谢通路Table 7. KEGG metabolic pathway of partial differentially expressed genes in ovarian stroma, large follicle and small follicle项目1)
Item通路
Pathway数目
CountP O vs LF 代谢途径 Metabolic pathway 298 4.90×10−17 抗生素的生物合成 Antibiotic biosynthesis 79 1.30×10−14 氧化磷酸化 Oxidative phosphorylation 77 1.10×10−22 内质网蛋白质加工 Protein processing in endoplasmic reticulum 70 1.00×10−14 泛素介导的蛋白水解 Ubiquitin mediated proteolysis 34 6.30×10−3 细胞周期 Cell cycle 32 7.30×10−3 甲状腺激素信号通路 Thyroid hormone signaling pathway 29 5.00×10−3 柠檬酸循环 Citrate cycle 17 2.20×10−6 Notch信号通路 Notch signaling pathway 13 4.90×10−2 类固醇生物合成 Steroid biosynthesis 12 1.90×10−4 O vs SF 癌症的途径 Pathway in cancer 28 3.20×10−2 细胞周期 Cell cycle 25 4.50×10−9 抗生素的生物合成 Antibiotic biosynthesis 17 4.00×10−2 RNA转运 RNA transport 16 1.10×10−2 嘌呤代谢 Purine metabolism 15 3.70×10−2 卵母细胞减数分裂 Oocyte meiosis 13 8.30×10−3 嘧啶代谢 Pyrimidine metabolism 11 1.30×10−2 DNA复制 DNA replication 9 2.30×10−4 孕酮介导的卵母细胞成熟 Progesterone-mediated oocyte maturation 9 4.30×10−2 错配修复 Mismatch repair 8 8.90×10−5 SF vs LF 代谢途径 Metabolic pathway 248 5.60×10−4 氧化磷酸化 Oxidative phosphorylation 74 3.80×10−19 内质网蛋白质加工 Protein processing in endoplasmic reticulum 67 6.00×10−12 抗生素的生物合成 Antibiotic biosynthesis 54 7.80×10−4 泛素介导的蛋白水解 Ubiquitin mediated proteolysis 40 2.90×10−4 甲状腺激素信号通路 Thyroid hormone signaling pathway 32 1.40×10−3 AMPK信号通路 AMPK signaling pathway 32 8.70×10−3 蛋白酶体 Proteasome 31 1.10×10−12 Notch信号通路 Notch signaling pathway 15 1.50×10−2 类固醇生物合成 Steroid biosynthesis 9 2.10×10−2 1) O:卵巢基质;LF:大卵泡;SF:小卵泡
1) O: Ovarian stroma; LF: Large follicle; SF: Small follicle2.5 基因筛选及qRT-PCR验证分析
从3组差异基因中筛选5个基因,分别是CYP19A1和INHBA(卵巢基质vs大卵泡)、GPNMB(卵巢基质vs小卵泡)、KITLG和SFRP1(小卵泡vs大卵泡),进行qRT-PCR验证(图2)。这些基因的表达变化趋势与测序结果基本一致,说明测序结果可靠。
3. 讨论
山羊性成熟时,以卵泡波的形式募集卵泡,并使卵泡发育至成熟排卵[36]。在形成有腔卵泡之前,卵泡的发育不受生殖激素调控,发育比较缓慢;在形成腔体时,性腺轴会分泌激素,促进卵泡发育或闭锁[37-38]。山羊是单胎或双胎动物[3],与人类相似,研究卵泡不同发育阶段的作用机制,山羊是较好的动物模型。
本研究采用高通量测序技术,首次对川中黑山羊的卵巢基质、大卵泡和小卵泡的mRNA表达量进行研究。结果表明卵巢基质、大卵泡和小卵泡的表达模式不同,其中卵巢基质和小卵泡的表达模式更相近,可能是因为在分离卵泡时,每个卵巢基质中d>1 cm的大卵泡已分离出来,但不可避免的,卵巢基质中含有少量的d<3 mm的小卵泡,从而造成卵巢基质和小卵泡的基因表达模式更相近。
通过差异基因分析,筛选出与其他哺乳动物卵泡发育或闭锁有关的基因,包括在大卵泡上调的抑制素基因家族、细胞色素P450家族19亚家族成员1(CYP19A1)、MGST1、BEX家族、APO家族、MMP家族、CITED家族、PRSS23、LRP8;在大卵泡下调的ADAMTS1、SPARCL1、TNFRSF家族。在猪[6]和牛[8, 11]的研究中,抑制素基因家族在闭锁卵泡或小卵泡中低表达,通过负反馈调节抑制垂体分泌促卵泡激素,影响卵泡闭锁。INHA和INHBA在牛大卵泡上调[10],有助于卵泡膜产生雄激素并抑制垂体产生卵泡刺激素。CYP19A1的过表达增加大窦状卵泡类固醇合成[6-8],促进卵泡发育。谷胱甘肽代谢MGST1与细胞的代谢和解毒能力有关,其高表达可能表示健康卵泡的解毒能力更好[39]。BEX家族是X染色体连锁基因家族,BEX2在小卵泡低表达,引起线粒体凋亡,使细胞周期阻滞于G1期,从而调控颗粒细胞增殖和凋亡[9, 40-41]。载脂蛋白APOA1在中卵泡中高表达,可能参与了卵泡的选择及发育过程[11]。基质金属蛋白酶(Matrix metallopeptidase,MMP)家族是一类依赖金属锌离子的蛋白水解酶家族,能有效地降解细胞外基质,可能与卵泡体积的快速增大有关[11]。ADAMTS1基因家族与膜外结构域的分裂有关,在小卵泡中表达上调[9],在经产奶牛排卵前的卵泡中上调[42]。SPARCL1是SPARC家族一员,在小卵泡中高表达,阻滞细胞停留在G1期,抑制细胞增殖[9, 43]。但IGFBP5、IGF1、MMP9、PGRMC1等基因的表达量与其他研究报道的不一致。值得注意的是,在本研究中经典颗粒细胞凋亡标志物TNF受体超家族(TNFRSF1B、TNFRSF19)在大卵泡中下调[8],与一些研究报道的无显著差异不同[6-7]。
进一步对差异基因功能分析发现,卵巢基质vs大卵泡、卵巢基质vs小卵泡、小卵泡vs大卵泡三者富集的通路大不相同,其中,卵巢基质vs大卵泡、小卵泡vs大卵泡富集的通路较多,且有大部分是相同的;而卵巢基质vs小卵泡富集的通路较少,与其他2组差异较大。说明可能是大卵泡的生物学功能较为丰富。功能富集表明不同组织表现出不同的富集特征,核苷酸结合和磷酸核苷结合过程在3组中都共同富集,但在卵巢基质vs大卵泡中,特有的GO条目包括RNA加工过程、细胞质、细胞内膜、酶的催化活性等,可能是因为大卵泡阶段代谢过程比较旺盛,大卵泡与卵巢基质之间物质运输比较频繁。在卵巢基质vs小卵泡中特有的GO条目有细胞应激反应、初级代谢过程等,可能是因为小卵泡正处于初级发育阶段。在小卵泡vs大卵泡中特有的GO条目有线粒体和线粒体膜、细胞器内膜等,可能是因为卵泡发育过程主要受到线粒体膜的调控。
与卵巢基质相比,柠檬酸循环和类固醇生物合成信号通路在大卵泡中上调。柠檬酸循环保证机体能量供给和促进物质代谢[44]。类固醇生物合成在卵泡发育中起着重要的作用[45],例如类固醇合成快速调节蛋白(Steroidogenic acute regulatory protein,STAR)可介导胆固醇进入线粒体内膜,调控孕酮和雌激素的合成[46-48],影响卵泡发育;健康卵泡中CYP19A1的上调,促进孕烯醇酮形成,孕烯醇酮是黄体酮(Progesterone,P4)和雌二醇(Estradiol,E2)的底物,可能是促进卵泡发育的主要因素[7]。
在卵巢基质vs小卵泡中,差异基因富集的信号通路与细胞分裂周期有关。有研究[49-50]报道,在小鼠卵泡发育阶段,细胞周期、DNA复制和减数分裂等多个信号通路被激活,与本试验结果一致,可能在小卵泡阶段,卵母细胞发育上调了与细胞分裂相关的信号通路。
在小卵泡vs大卵泡中,差异基因富集的甲状腺激素信号通路、内质网蛋白质加工、AMPK信号通路、类固醇生物合成和Notch信号通路在猪[7]和牛[10]的卵泡转录组研究中均有报道。内质网蛋白质加工和甲状腺激素信号通路的显著富集,可能是因为随着卵泡发育,颗粒细胞分化成熟,甾体激素合成逐渐增强[51-52],物质代谢也因此增强。在人和大鼠的研究中发现[53],类固醇激素(如孕酮)与不同膜受体结合,抑制颗粒细胞的有丝分裂或凋亡,从而抑制卵泡发育或闭锁,维持卵泡的平衡状态。关于绵羊的研究发现,Notch信号通路调节细胞增殖、分化和凋亡,参与卵巢基质卵泡的发育[54]。
4. 结论
卵巢基质、大卵泡、小卵泡的差异mRNA表达模式不相同,其中卵巢基质与小卵泡更相近。就卵泡发育而言,深入研究这些特异性mRNA在卵泡不同发育阶段的作用有着重要意义。本文从生物信息学角度分析卵巢不同组织表达谱,为下一步差异基因的功能验证及其在卵泡发育中的作用提供一定的理论基础。
致谢:感谢练志全帮助分离小卵泡和大卵泡,感谢赵智锋帮助分析转录组测序结果!
-
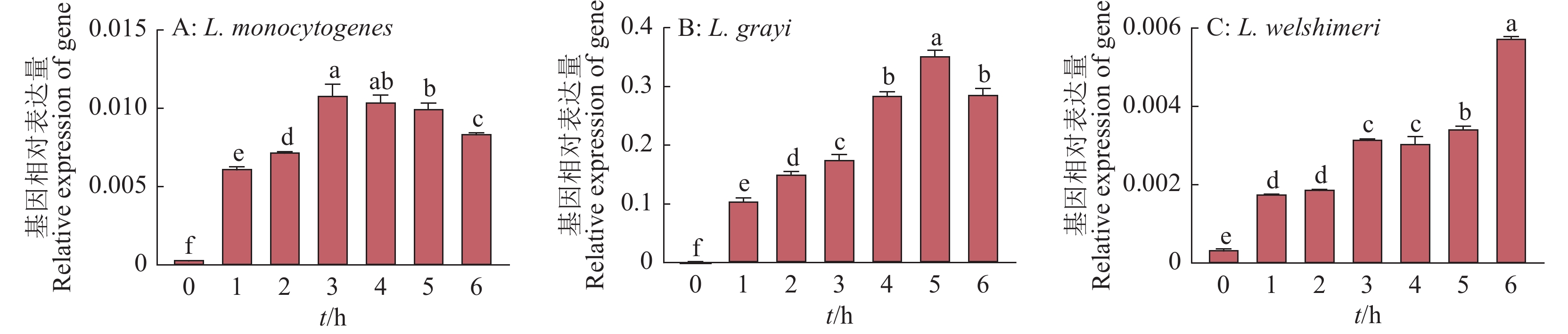
图 1 单增李斯特菌、格氏李斯特菌和威尔斯李斯特菌侵染果蝇S2细胞后的增殖情况
各图中,柱子上方的不同小写字母表示差异显著(P<0.05,LSD法)。
Figure 1. The proliferation of Listeria monocytogenes, L. grayi and L. welshimeri after their infection in Drosophila S2 cells
In each figure, different lowercase letters on bars indicate significant differences (P<0.05, LSD method).
表 1 qRT-PCR检测基因引物
Table 1 Primers for qRT-PCR detection
基因
Gene正向引物序列(5′→3′)
Forward primer sequence反向引物序列(5′→3′)
Reverse primer sequence产物长度/bp
Product lengthPutative oxidoreductase GGATAAAGGCATTATTGAACGT CAACTACTGGGTGCGGGAT 227 Species-specific
Oxidoreductase-codingAACTCCGTCCATCCATCACC TCGTCCGAGGCTAGGAATAAG 173 ScrA ACCACCAAGACAAGCAGTTAGA CATGTAGAAATGATTGCCCAAA 237 DmRp49 GACAGTATCTGATGCCCAACA CTTCTTGGAGGAGACGCCGT 170 DmAttD AATAACGCCGCTCTGAATG ATTGAGTCCTCCGCCAAA 75 DmDptA ACTGAATGGAGGATATGG AATCTGTAGGTGTAGGTG 90 DmDef ATGAAGTTCTTCGTTCTC CATGATCCTCTGGAATTG 100 DmDptB TTCACCGCTAGTCTTCTA GGGATCAGGATAGGGATAA 75 DmDro TCCTGCTGCTTGCTTGCG ATGGGTGGTGGCCTGATGG 163 DmDrs ATGATGCAGATCAAGTAC AGGGACCCTTGTATCTTC 109 DmMtk TACATCAGTGCTGGCAGAG TTGGTTGGTTAGGATTGAAGG 83 表 2 单增李斯特菌侵染后果蝇S2细胞特有上调基因的KEGG富集分析
Table 2 KEGG enrichment of unique up-regulated genes in Drosophila S2 cells after Listeria monocytogenes infection
通路
PathwayP 所含基因
Contained gene背腹轴形成 Dorso-ventral axis formation 0.019 aos 铂类药物抗药性 Platinum drug resistance 0.041 GstE9 化学诱发癌变 Chemical carcinogenesis 0.042 GstE9 药物代谢−细胞色素 P450 Drug metabolism-cytochrome P450 0.042 GstE9 细胞色素 P450代谢异物质 Metabolism of xenobiotics by cytochrome P450 0.042 GstE9 谷胱甘肽代谢 Glutathione metabolism 0.049 GstE9 表 3 单增李斯特菌侵染后果蝇S2细胞特有下调基因的KEGG富集分析
Table 3 KEGG enrichment of unique down-regulated genes in Drosophila S2 cells after Listeria monocytogenes infection
通路
PathwayP 所含基因
Contained gene苯丙氨酸代谢 Phenylalanine metabolism 0.042 CG11796 泛醌和其他萜类醌的生物合成 Ubiquinone and other terpenoid-quinone biosynthesis 0.047 CG11796 胰液分泌 Pancreatic secretion 0.050 iotaTry, RyR 表 4 格氏李斯特菌侵染后果蝇S2细胞特有上调基因的KEGG富集分析
Table 4 KEGG enrichment of unique up-regulated genes in Drosophila S2 cells after Listeria grayi infection
通路
PathwayP 所含基因
Contained geneToll和Imd信号传导途径 Toll and Imd signaling pathway 0.000 Def, Drs, CecC, CecB, CecA1, pirk 补体和凝血级联反应 Complement and coagulation cascades 0.003 Hml 细胞外基质−受体相互作用 ECM-receptor interaction 0.016 Hml 表 5 格氏李斯特菌侵染后果蝇S2细胞特有下调基因的KEGG富集分析
Table 5 KEGG enrichment of unique down-regulated genes in Drosophila S2 cells after Listeria grayi infection
通路
PathwayP 所含基因
Contained gene2型糖尿病 Type II diabetes mellitus 0.010 CG7069 癌细胞中的中心碳代谢 Central carbon metabolism in cancer 0.012 CG7069 丙酮酸代谢 Pyruvate metabolism 0.015 CG7069 糖酵解/糖异生 Glycolysis/gluconeogenesis 0.017 CG7069 胰高血糖素信号传导途径 Glucagon signaling pathway 0.021 CG7069 病毒致癌 Viral carcinogenesis 0.043 CG7069 嘌呤代谢 Purine metabolism 0.046 CG7069 表 6 威尔斯李斯特菌侵染后果蝇S2细胞特有下调基因的KEGG富集分析
Table 6 KEGG enrichment of unique down-regulated genes in Drosophila S2 cells after Listeria welshimeri infection
通路
PathwayP 所含基因
Contained gene抗原处理与呈递 Antigen processing and presentation 0.000 Hsp70Bbb, Hsp68, Hsp70Bc, Hsp70Bb 军团菌病 Legionellosis 0.000 Hsp70Bbb, Hsp68, Hsp70Bc, Hsp70Bb 甲型流感 Influenza A 0.000 Hsp70Bbb, deltaTry, Hsp70Bc, Hsp68, Hsp70Bb 弓形虫感染 Toxoplasmosis 0.000 Hsp70Bbb, Hsp68, Hsp70Bc, Hsp70Bb 寿命调控途径−多个物种 Longevity regulating pathway-multiple species 0.000 Hsp70Bbb, Hsp68, Hsp70Bc, Hsp70Bb 雌激素信号传导途径 Estrogen signaling pathway 0.000 Hsp70Bbb, Hsp68, Hsp70Bc, Hsp70Bb 麻疹 Measles 0.000 Hsp70Bbb, Hsp68, Hsp70Bc, Hsp70Bb MAPK信号传导途径 MAPK signaling pathway 0.000 Hsp70Bbb, Hsp68, Hsp70Bc, Hsp70Bb 剪接体 Spliceosome 0.000 Hsp70Bbb, Hsp68, Hsp70Bc, Hsp70Bb 内吞作用 Endocytosis 0.000 Hsp70Bbb, Hsp68, Hsp70Bc, Hsp70Bb 内质网蛋白加工 Protein processing in endoplasmic reticulum 0.000 Hsp70Bbb, Hsp68, Hsp70Bc, Hsp70Bb 表 7 单增李斯特菌、格氏李斯特菌以及威尔斯李斯特菌侵染后果蝇S2细胞共有下调基因的KEGG富集分析
Table 7 KEGG enrichment of common down-regulated genes in Drosophila S2 cells after Listeria monocytogenes, L. grayi and L. welshimeri infection
通路
PathwayP 所含基因
Contained gene咖啡因代谢 Caffeine metabolism 0.007 AOX2 维生素B6代谢 Vitamin B6 metabolism 0.007 CG14212 牛磺酸和低牛磺酸代谢 Taurine and hypotaurine metabolism 0.010 Ggt-1 花生四烯酸代谢 Arachidonic acid metabolism 0.020 Ggt-1 矿物质吸收 Mineral absorption 0.024 Ctr1B 集合管酸分泌 Collecting duct acid secretion 0.037 Vha100-4 类风湿性关节炎 Rheumatoid arthritis 0.050 Vha100-4 -
[1] COLLINS M D, WALLBANKS S, LANE D J, et al. Phylogenetic analysis of the genus Listeria based on reverse transcriptase sequencing of 16S rRNA[J]. International Journal of Systematic Bacteriology, 1991, 41(2): 240-246. doi: 10.1099/00207713-41-2-240
[2] 何冬梅, 邓峰, 赖蔚苳, 等. 单核细胞增生李斯特菌生物学研究进展[J]. 华南预防医学, 2006, 32(6): 26-29. [3] 柴文琴. 李斯特菌属不同种型在模拟食品加工环境相关胁迫下的存活率与耐药性研究[D]. 杭州: 浙江大学, 2020. [4] KAPTCHOUANG TCHATCHOUANG C D, FRI J, DE SANTI M, et al. Listeriosis outbreak in South Africa: A comparative analysis with previously reported cases worldwide[J]. Microorganisms, 2020, 8(1): 135. doi: 10.3390/microorganisms8010135
[5] CHARLIER C, PERRODEAU É, LECLERCQ A, et al. Clinical features and prognostic factors of listeriosis: The MONALISA national prospective cohort study[J]. The Lancet Infectious Diseases, 2017, 17(5): 510-519. doi: 10.1016/S1473-3099(16)30521-7
[6] 中华人民共和国国家卫生健康委员会, 国家市场监督管理总局. 食品安全国家标准 预包装食品中致病菌限量: GB 29921—2021[S]. 北京: 中国标准出版社, 2021. [7] 中华人民共和国家卫生和计划生育委员会. 食品安全国家标准 食品中致病菌限量: GB 29921—2013[S]. 北京: 中国标准出版社, 2014. [8] VÁZQUEZ-BOLAND J A, KUHN M, BERCHE P, et al. Listeria pathogenesis and molecular virulence determinants[J]. Journal of Personalized Medicine, 2001, 14(3): 584-640.
[9] 李钊, 刘阳泰, 李卓思, 等. 单增李斯特菌毒力因子及调控机制研究进展[J]. 食品与发酵工业, 2024, 50(11): 327-335. [10] IRETON K, MORTUZA R, GYANWALI G C, et al. Role of internalin proteins in the pathogenesis of Listeria monocytogenes[J]. Molecular Microbiology, 2021, 116(6): 1407-1419. doi: 10.1111/mmi.14836
[11] ZHOU H, COVENEY A P, WU M, et al. Activation of both TLR and NOD signaling confers host innate immunity-mediated protection against microbial infection[J]. Frontiers in Immunology, 2019, 9: 3082. doi: 10.3389/fimmu.2018.03082
[12] MITCHELL G, CHENG M I, CHEN C, et al. Listeria monocytogenes triggers noncanonical autophagy upon phagocytosis, but avoids subsequent growth-restricting xenophagy[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 115(2): E210-E217.
[13] CHEN G Y, PENSINGER D A, SAUER J D. Listeria monocytogenes cytosolic metabolism promotes replication, survival, and evasion of innate immunity[J]. Cellular Microbiology, 2017, 19(10): e12762-e12771. doi: 10.1111/cmi.12762
[14] GABALLA A, GUARIGLIA-OROPEZA V, WIEDMANN M, et al. Cross talk between SigB and PrfA in Listeria monocytogenes facilitates transitions between extra- and intracellular environments[J]. Microbiology and Molecular Biology Reviews, 2019, 83(4): e00034-19.
[15] BIERNE H, HAMON M, COSSART P. Epigenetics and bacterial infections[J]. Cold Spring Harbor Perspectives in Medicine, 2012, 2(12): a010272
[16] MINÁROVITS J. Microbe-induced epigenetic alterations in host cells: The coming era of patho-epigenetics of microbial infections: A review[J]. Acta Microbiologica et Immunologica Hungarica, 2009, 56(1): 1-19. doi: 10.1556/AMicr.56.2009.1.1
[17] HAMON M A, COSSART P. Histone modifications and chromatin remodeling during bacterial infections[J]. Cell Host & Microbe, 2008, 4(2): 100-109.
[18] COSTA A C, PINHEIRO J, REIS S A, et al. Listeria monocytogenes interferes with host cell mitosis through its virulence factors InlC and ActA[J]. Toxins, 2020, 12(6): 411-424. doi: 10.3390/toxins12060411
[19] 袁江北. 定量蛋白质组学揭示免疫细胞对李斯特菌感染的防御机制[D]. 重庆: 重庆大学, 2020. [20] IMLER J L. Overview of Drosophila immunity: A historical perspective[J]. Developmental & Comparative Immunology, 2014, 42(1): 3-15.
[21] GOTTAR M, GOBERT V, MICHEL T, et al. The Drosophila immune response against Gram-negative bacteria is mediated by a peptidoglycan recognition protein[J]. Nature, 2002, 416: 640-644. doi: 10.1038/nature734
[22] SCHMID M W, NG E Y W, LAMPIDIS R, et al. Evolutionary history of the genus Listeria and its virulence genes[J]. Systematic and Applied Microbiology, 2005, 28(1): 1-18. doi: 10.1016/j.syapm.2004.09.005
[23] PIZARRO-CERDA J, COSSART P. Listeria monocytogenes: Cell biology of invasion and intracellular growth[J]. Microbiology Spectrum, 2018, 6(6). doi: 10.1128/microbiolspec.GPP3-0013-2018.
[24] FREITAG N E, PORT G C, MINER M D. Listeria monocytogenes: From saprophyte to intracellular pathogen[J]. Nature Reviews Microbiology, 2009, 7: 623-628. doi: 10.1038/nrmicro2171
[25] 刘甜, 罗开珺. 果蝇Toll和IMD信号通路中的功能结构域[J]. 环境昆虫学报, 2011, 33(3): 388-395. doi: 10.3969/j.issn.1674-0858.2011.03.015 [26] LEULIER F, RODRIGUEZ A, KHUSH R S, et al. The Drosophila caspase Dredd is required to resist gram-negative bacterial infection[J]. EMBO Reports, 2000, 1(4): 353-358. doi: 10.1093/embo-reports/kvd073
[27] BALDWIN D N, VANCHINATHAN V, BROWN P O, et al. A gene-expression program reflecting the innate immune response of cultured intestinal epithelial cells to infection by Listeria monocytogenes[J]. Genome Biology, 2002, 4(1): R2. doi: 10.1186/gb-2002-4-1-r2
[28] 吴坤钟, 李荣松, 曹阳, 等. 英诺克李斯特菌侵染果蝇S2细胞后的转录组分析[J]. 华南农业大学学报, 2020, 41(5): 17-26. doi: 10.7671/j.issn.1001-411X.202001023 [29] 王旭. 基于比较基因组学和转录组学技术揭示单增李斯特菌毒力因子的研究[D]. 上海: 上海海洋大学, 2016. [30] PIZARRO-CERDÁ J, COSSART P. Subversion of cellular functions by Listeria monocytogenes[J]. Journal of Pathology, 2006, 208(2): 215-223. doi: 10.1002/path.1888
[31] KRYPOTOU E, SCORTTI M, GRUNDSTRÖM C, et al. Control of bacterial virulence through the peptide signature of the habitat[J]. Cell Reports, 2019, 26(7): 1815-1827. e5.
[32] SAISAWANG C, WONGSANTICHON J, KETTERMAN A J. A preliminary characterization of the cytosolic glutathione transferase proteome from Drosophila melanogaster[J]. Biochemical Journal, 2012, 442(1): 181-190. doi: 10.1042/BJ20111747
[33] XIE A, ODATE S, CHANDRAMOULY G, et al. H2AX post-translational modifications in the ionizing radiation response and homologous recombination[J]. Cell Cycle (Georgetown, Tex), 2010, 9(17): 3602-3610. doi: 10.4161/cc.9.17.12884
[34] WU M H, ZHANG X D, WEI W, et al. CRISPR/Cas9 mediated genetic resource for unknown kinase and phosphatase genes in Drosophila[J]. Scientific Reports, 2020, 10: 7383. doi: 10.1038/s41598-020-64253-4
[35] SCHERFER C, KARLSSON C, LOSEVA O, et al. Isolation and characterization of hemolymph clotting factors in Drosophila melanogaster by a pullout method[J]. Current Biology, 2004, 14(7): 625-629. doi: 10.1016/j.cub.2004.03.030
[36] 单颖. 单核细胞增生李斯特菌: 无菌斑马鱼感染模型及Mmp-9在抗细菌感染中的作用机制[D]. 杭州: 浙江大学, 2016. -
期刊类型引用(32)
1. 牛潘婷,张宝林,潘丽杰,郭建鹏. 基于改进GoogLeNet的玉米叶片病害识别及其可解释性研究. 中国农机化学报. 2025(01): 204-212 .  百度学术
百度学术
2. 朱德明,程香平,邱伊健,李天赐,万翔,孙旭,韦江,付远,饶雄海. 基于深度学习的农作物图像识别技术研究进展. 江西科学. 2025(01): 154-161 .  百度学术
百度学术
3. 蔡竹轩,蔡雨霖,曾凡国,岳学军. 基于改进YOLOv5l的田间水稻稻穗识别. 华南农业大学学报. 2024(01): 108-115 .  本站查看
本站查看
4. 牛潘婷,张宝林,潘丽杰,郭建鹏,李瑞鑫. 基于深度学习的农作物病虫害研究进展. 内蒙古师范大学学报(自然科学汉文版). 2024(01): 93-102 .  百度学术
百度学术
5. 冼远清,江颖龙,初庆柱,彭小红. 基于深度学习的鱼病防治APP教学实践系统设计. 实验室研究与探索. 2024(03): 12-17 .  百度学术
百度学术
6. 张会敏,吉秉彧,谢泽奇. 基于多尺度注意力机制网络的玉米害虫识别方法. 江苏农业科学. 2024(09): 241-247 .  百度学术
百度学术
7. 贾金豹,朱成娟,汪家全,周鹏. 基于机器学习结合多因子组合的玉米估产研究. 中国农机化学报. 2024(10): 206-214 .  百度学术
百度学术
8. 李杰,李广明,蔡钰珊. 改进的轻量级GSE-YOLOv8荔枝虫害检测方法. 数字技术与应用. 2024(09): 220-222 .  百度学术
百度学术
9. 方文博,郭永刚,关法春,张伟,刘倩倩,王树文,张正超,于皓然. 基于改进YOLO v5s算法的大豆叶片虫洞的识别. 湖南农业大学学报(自然科学版). 2023(01): 127-132 .  百度学术
百度学术
10. 阮炬全,刘朔. 基于高阶残差和注意力机制的轻量型作物害虫识别. 计算机系统应用. 2023(03): 104-115 .  百度学术
百度学术
11. 温艳兰,陈友鹏,王克强,程杏安,林钦永,蔡肯,马佳佳,孔翰博. 基于迁移学习和改进残差网络的复杂背景下害虫图像识别. 江苏农业科学. 2023(08): 171-177 .  百度学术
百度学术
12. 高家军,张旭,郭颖,刘昱坤,郭安琪,石蒙蒙,王鹏,袁莹. 融合Swin Transformer的虫害图像实例分割优化方法研究. 南京林业大学学报(自然科学版). 2023(03): 1-10 .  百度学术
百度学术
13. 张彦通,苏前敏. 基于迁移学习的玉米病害图像识别. 中国农业科技导报. 2023(10): 119-125 .  百度学术
百度学术
14. 赵法川,徐晓辉,宋涛,郝淼淼,汪曙,朱伟龙. 融合多头注意力的轻量级作物病虫害识别. 华南农业大学学报. 2023(06): 986-994 .  本站查看
本站查看
15. 段新涛,王伸,赵晴,张杰,郑国清,李国强. 基于改进YOLOv4的夏玉米主要害虫检测方法研究. 山东农业科学. 2023(10): 167-173 .  百度学术
百度学术
16. 邹玮,岳延滨,冯恩英,彭顺正,张爱民,肖玖军. 基于YOLO v2的辣椒叶部蚜虫图像识别. 山东农业大学学报(自然科学版). 2023(05): 700-709 .  百度学术
百度学术
17. 李睿,王莹,王恒. 基于树莓派的室内植物病虫害识别系统设计. 电子设计工程. 2022(08): 114-118 .  百度学术
百度学术
18. 朱伟,马立新,张平,刘德营. 基于GoogLeNet和无人机图像的水稻秧苗形态识别. 华南农业大学学报. 2022(03): 99-106 .  本站查看
本站查看
19. 徐聪,王旭启,刘裕. 一种改进可形变FCN的农作物害虫检测方法. 江苏农业科学. 2022(09): 211-219 .  百度学术
百度学术
20. 雷建云,陈楚,郑禄,帖军,赵捷. 基于改进残差网络的水稻害虫识别. 江苏农业科学. 2022(14): 190-198 .  百度学术
百度学术
21. 叶名炀,张杰强. 基于轻量化网络MobileNetV2的玉米病害识别研究. 现代计算机. 2022(11): 46-50 .  百度学术
百度学术
22. 刘阳,高国琴. 基于轻量级SSD模型的夜间金蝉若虫检测. 农业工程学报. 2022(10): 166-175 .  百度学术
百度学术
23. 彭红星,徐慧明,刘华鼐. 基于改进ShuffleNet V2的轻量化农作物害虫识别模型. 农业工程学报. 2022(11): 161-170 .  百度学术
百度学术
24. 范博文,段敏. 采用模糊支持向量机算法的前车识别系统. 重庆理工大学学报(自然科学). 2022(09): 172-178 .  百度学术
百度学术
25. 叶云,赵小娟,姜晟. 基于深度学习的荔枝虫害识别技术的应用与实现. 中国农业信息. 2022(04): 30-37 .  百度学术
百度学术
26. 温艳兰,陈友鹏,王克强,刘展眉,林钦永,蔡肯,马佳佳,孔翰博. 基于机器视觉的病虫害检测综述. 中国粮油学报. 2022(10): 271-279 .  百度学术
百度学术
27. 尚皓玺,郭小燕,朱恒宇. 基于迁移学习与GhostNet模型的农业害虫图像识别研究. 软件导刊. 2022(11): 137-143 .  百度学术
百度学术
28. 丁作坤,丁晶晶. 基于信息化技术的人居环境整治优化. 现代计算机. 2021(08): 54-59 .  百度学术
百度学术
29. 李小玄,董雷. 基于深度学习的消防器材自动识别研究. 电子设计工程. 2021(19): 53-57 .  百度学术
百度学术
30. 张重豪,贾俊铖. 农作物病虫害智能识别APP设计与开发. 福建电脑. 2021(10): 1-7 .  百度学术
百度学术
31. 骆润玫,王卫星. 基于卷积神经网络的植物病虫害识别研究综述. 自动化与信息工程. 2021(05): 1-10 .  百度学术
百度学术
32. 樊湘鹏,周建平,许燕. 基于改进区域卷积神经网络的田间玉米叶部病害识别. 华南农业大学学报. 2020(06): 82-91 .  本站查看
本站查看
其他类型引用(22)



 下载:
下载: