Detection and analysis of QTL for panicle length in rice using a high-density genetic map
-
摘要:目的
深入挖掘与穗长相关的新基因,为水稻穗长调控的遗传机理研究及分子育种提供依据。
方法以2个优良亲本‘ZP37’和‘R8605’及其杂交衍生的208个高世代重组自交系(Recombinant inbred lines,RILs)为作图群体,利用全基因组重测序高密度连锁图谱对3个不同环境下的穗长数量性状座位(Quantitative trait locus,QTL)进行定位,同时分析它们的聚合效应。
结果共检测到11个穗长QTL,分别分布在第3、4、7、8、9和12号染色体上,其似然函数比对数值(Log of odds,LOD)介于3.07~12.87之间,贡献率在2.17%~10.94%之间,有7个QTL是新位点,其余4个QTL位点与已报道的穗长基因和QTL位置重叠或相近。在2个不同环境下重复检测到4个稳定的QTL位点;对聚合了不同数量穗长QTL株系的分析结果表明,穗长QTL表现出累加效应,QTL数量的增加能显著增加水稻穗长。
结论本研究结果为水稻穗长QTL的克隆和功能解析奠定坚实的基础,为水稻高产育种提供理论依据和遗传资源。
Abstract:ObjectiveTo deeply explore new genes related to panicle length and provide a basis for the study of genetic mechanism of panicle length regulation and molecular breeding in rice.
MethodTwo superior parents, ‘ZP37’ and ‘R8605’, as well as 208 recombinant inbred lines (RILs) derived from the cross of ZP37/R8605 were used as a mapping population to locate quantitative trait loci (QTLs) for panicle length in three different environments through the high-density linkage map of whole genome resequencing, and to analyze their pyramiding effects.
ResultA total of 11 QTLs for panicle length were detected on chromosomes 3, 4, 7, 8, 9 and 12, with the logs of odds (LODs) ranging from 3.07 to 12.87 and contribution rates ranging from 2.17% to 10.94%, seven of the QTLs were new loci, and the remaining four QTLs overlapped or were close to the reported panicle length genes and QTLs. Among them, four stable QTLs were detected repeatedly in two different environments, and by analyzing the lines that pyramiding different numbers of panicle length QTLs, the results showed that the panicle length QTLs showed an additive effect, and the increase in the number of QTLs significantly increased the panicle length of rice.
ConclusionThe results of this study provide a solid foundation for cloning and functional analysis of rice panicle length QTLs, as well as a theoretical basis and genetic resources for high-yield rice breeding.
-
Keywords:
- Oryza sativa L. /
- Spike length /
- QTL mapping /
- High-density genetic map
-
体内脂肪过度沉积对人体健康和动物胴体品质有不良影响[1],降低体内脂肪沉积可改善动物胴体品质[2-3],提高人体健康。钙作为机体必需元素,参与脂肪沉积调控[4-5],但其具体作用及调节机制尚不完全清楚。因此,研究钙对动物机体脂肪沉积的影响及机制,对于提高动物胴体品质及人体健康具有重要意义。
肠道菌群数量众多,作用广泛,对机体健康具有重要意义[6]。肠道菌群和肥胖的关系是现在研究的热点之一。有研究报道,肥胖与肠道细菌分布有关,厚壁菌门Firmicutes多于拟杆菌门Bacteroidetes导致肠道更有效吸收食物中的热量从而导致肥胖[7]。但目前的研究中,有关钙离子是否通过改变肠道菌群来调控肥胖的报道却较少。因此,研究钙离子对肠道菌群的影响有利于进一步深入了解钙离子调控脂肪沉积的机制。
本试验以C57BL小鼠为试验对象,研究饮水添加氯化钙对高脂日粮饲喂小鼠的脂肪沉积的影响,同时分析钙离子对肠道菌群的影响,以及肠道菌群与脂肪沉积可能的关联,旨在为提高动物胴体品质和人体健康提供理论依据。
1. 材料与方法
1.1 试验动物与试验设计
选用27只4周龄C57BL雄性小鼠(购于广东省医学实验动物中心),试验用的高脂日粮(High-fat diet, HFD)购于广东省医学实验动物中心,氯化钙购于Sigma公司。预饲小鼠正常日粮1周后,根据体质量随机分为2组:高脂组(HFD组),n=15,饲喂高脂日粮,脂肪提供60%的能量;高脂日粮+饮水添加氯化钙组(HFD+Ca2+组),n=12,饲喂高脂日粮,脂肪提供60%的能量,饮水中添加16.647 g/L的氯化钙。将小鼠分笼饲养,自由采食和饮水;每天光照与黑暗各12 h,环境温度控制在(25±1) ℃,湿度(60±5)%。饲养13周后,通过颈部脱臼方式处死小鼠,采集小鼠附睾脂肪、皮下脂肪组织并称质量。并在小鼠饲养至10~12周时采集小鼠的新鲜粪便样,于–80 ℃条件下进行保存。
1.2 测定指标及方法
饲养期间每周测定小鼠体质量和体内脂肪含量,体内脂肪含量通过核磁共振成像仪(上海纽迈科技有限公司,MesoQMR23-060H型)进行测定。样品采样后测定小鼠附睾脂肪、皮下脂肪质量。粪便样菌群送公司(上海微基生物科技有限公司)进行16S rRNA高通量测序,测定指标包括菌群丰度,菌群多样性。
1.3 统计分析
数据结果用平均值±标准误表示,统计分析采用SigmaPlot 12.5软件分析,采用t检验比较不同分组的统计差异。
2. 结果与分析
2.1 饮水添加氯化钙对高脂日粮饲喂小鼠体质量和脂肪沉积的影响
本研究中,与高脂组相比,饮水添加氯化钙使小鼠的体质量(图1A)、体脂含量(图1B)、皮下脂肪指数(图1C)和附睾脂肪指数(图1D)分别下降了12.85%、32.69%、26.65%和18.60%,差异均显著。
![图 1 饮水添加氯化钙对高脂日粮饲喂小鼠体质量(a)、体脂含量(b)、皮下脂肪指数(c)和附睾脂肪指数(d)的影响]() 图 1 饮水添加氯化钙对高脂日粮饲喂小鼠体质量(a)、体脂含量(b)、皮下脂肪指数(c)和附睾脂肪指数(d)的影响“*” 和 “**” 分别表示与HFD组差异达到0.05和0.01的显著水平(t检验)Figure 1. Effects of calcium chloride supplementation in drinking water on body weight (a), body fat content (b), subcutaneous fat index (c) and epididymal fat index (d) of mice fed high-fat diet“*” and “**” indicate the significant difference between HFD and HFD+Ca2+ at 0.05 and 0.01 levels, respectively (t test)
图 1 饮水添加氯化钙对高脂日粮饲喂小鼠体质量(a)、体脂含量(b)、皮下脂肪指数(c)和附睾脂肪指数(d)的影响“*” 和 “**” 分别表示与HFD组差异达到0.05和0.01的显著水平(t检验)Figure 1. Effects of calcium chloride supplementation in drinking water on body weight (a), body fat content (b), subcutaneous fat index (c) and epididymal fat index (d) of mice fed high-fat diet“*” and “**” indicate the significant difference between HFD and HFD+Ca2+ at 0.05 and 0.01 levels, respectively (t test)2.2 饮水添加氯化钙对高脂日粮饲喂小鼠粪便菌群的影响
2.2.1 小鼠粪便菌群的物种丰度及多样性
利用16S rRNA高通量测序方法,得到了粪便菌群的菌群多样性和菌群丰度。香农指数显示,饮水添加氯化钙可提高小鼠的粪便菌群群落多样性(图2A)。Chao指数显示,饮水添加氯化钙能提高小鼠的粪便菌群丰度(图2B)。
2.2.2 小鼠粪便菌群组成
PCA结果显示,高脂组的菌群聚集与饮水添加氯化钙组的菌群聚集有明显的分离情况(图3)。
2.2.3 小鼠粪便菌群的物种相对丰度
16S rRNA测序结果表明,粪便菌群主要由6个细菌门构成:拟杆菌门Bacteroidetes、厚壁菌门Firmicutes、变形菌门Proteobacteria、脱铁杆菌门Deferribacteres、放线菌门Actinobacteria、软壁菌门Tenericutes,其中拟杆菌门和厚壁菌门为主要优势菌群。在门水平下,高脂饲喂小鼠粪便中的拟杆菌门、脱铁杆菌门、放线菌门的相对丰度较高,饮水添加氯化钙饲喂小鼠粪便中的厚壁菌门、变形菌门、软壁菌门的相对丰度较高。但这6种菌门的丰度在2个处理组的差异不显著。
在纲水平下,与高脂组相比,饮水添加氯化钙可显著降低丹毒丝菌纲Erysipelotrichia和放线菌纲Actinobacteria的相对丰度,并且显著提高与胆汁酸代谢相关的梭菌纲Clostridia的相对丰度,Negativicoccus只存在高脂组中。
在目水平下,与高脂组相比,饮水添加氯化钙可显著降低丹毒丝菌目Erysipelotrichia和放线菌目Coriobacteriaceae的相对丰度,并且显著提高梭菌目Clostridia的相对丰度。
在科水平下,与高脂组相比,饮水添加氯化钙可显著降低丹毒丝菌科Erysipelotrichia的相对丰度,并且显著提高瘤胃菌科Ruminococcaceae、优干菌科Eubacteriaceae、消化链球菌科Peptostreptococcaceae的相对丰度。动球菌科Planococcaceae、短杆菌科Brevibacteriaceae、肉杆菌科Carnobacteriaceae只存在于饮水添加氯化钙组。韦荣氏杆菌科Veillonellaceae只存在于高脂组。
在属水平下,与高脂组相比,饮水添加氯化钙能显著降低Allobaculum的相对丰度。与高脂组相比,饮水添加氯化钙能显著提高紫单胞菌属Parabacteroides、Anaerofustis的相对丰度。但土壤芽胞杆菌属Solibacillus、短杆菌属Brevibacterium、Turicibacter、Desemzia只存在于饮水添加氯化钙中。其中,微生物Turicibacter已被证明与抗炎表型相关[8]。
3. 讨论与结论
3.1 饮水添加氯化钙对高脂日粮饲喂小鼠脂肪沉积的影响
有文献报道,高钙源的摄入可以显著减少肥胖病人的腹部脂肪[9],降低肥胖小鼠体内皮下和附睾脂肪含量[10]。与以上报道相一致的是,本研究结果表明,饮水添加氯化钙显著减少高脂日粮饲喂小鼠体质量,减少体内皮下和附睾脂肪含量。但也有研究发现,高钙饮食并不能起到减轻肥胖的作用[11],这可能与添加钙的浓度、动物品种和动物个体差异的因素有关。因此,钙的摄入对动物和人的体质量及肥胖具有一定的调控作用。
本研究结果表明,虽然本试验钙的饮水添加方式与膳食钙的添加方式不同,但是也起到了降低肥胖的作用。机体的低钙水平会对正常的生理活动造成影响,而过高剂量的钙摄入对机体也会造成损伤,尤其是对心血管、肾和肝组织损伤明显。有研究报道,高钙饲料(钙质量分数为3%)的摄入未对大鼠肝肾脏造成损伤[12],而本研究添加的氯化钙质量浓度为16.647 g/L,因此,本研究添加的剂量应该对小鼠机体健康无明显影响。
3.2 饮水添加氯化钙对高脂日粮饲喂小鼠粪便菌群的影响
本试验中,香农指数和Chao指数显示,饮水添加氯化钙可提高高脂日粮饲喂小鼠粪便菌群群落多样性和菌群丰度。样品组成越相似,反映在PCA图中的距离越近。本试验中,通过PCA分析可知,饮水添加氯化钙已经影响高脂日粮饲喂小鼠粪便菌群样本的组成。Duncan等[13]研究表明使肥胖受试者体质量减轻的饮食可显著改变肠道微生物群的物种组成,但没有发现粪便细菌中拟杆菌和厚壁菌的比例在人类肥胖中起作用的证据。我们的研究结果发现饮水添加氯化钙显著降低体质量,其饲喂小鼠粪便中的厚壁菌门相对丰度增加,拟杆菌门相对丰度减少。但Ley等[14]发现肥胖人群粪便菌群里的厚壁菌门和拟杆菌门比值高于正常体质量人群,给予肥胖志愿者低脂饮食1年后,其体质量出现下降,而体内的厚壁菌门比例有了下降,拟杆菌门的比例则上升,这与本研究的结果不一致。但这一观点受到后续研究的挑战。最近的研究表明,肥胖与微生物多样性或丰度降低相关[15]。也有文献报道,随着身体质量指数(Body mass index, BMI)大小上升,肠道菌群多样性与丰富度呈下降趋势[16]。因此,饮水添加氯化钙可能是通过改变高脂日粮饲喂小鼠粪便菌群物种丰度与群落多样性来减少脂肪沉积。
肠道菌群虽然有很大的个体差异,但也有一定的共性[17]。有研究表明,粪便菌群和盲肠内容物的菌群变化趋势是相似的[18]。Nadeem等[18]报道,高钙饮食能提高盲肠内容物里的瘤胃菌科水平,降低盲肠内容物丹毒丝菌科水平。这与本试验的饮水添加氯化钙组能提高粪便菌群里的瘤胃菌科相对丰度和降低丹毒丝菌科相对丰度结果一致。
胆汁酸在脂质代谢中起重要的调节作用。胆汁酸不仅参与胆固醇的调节,而且在三酰甘油的代谢中也发挥着重要作用[19]。肠道微生物群通过促进初代胆汁酸的去共轭、脱氢和脱羟基作用对胆汁酸代谢具有深远的影响[20]。胆汁盐脱羧由胆汁盐水解酶催化。这种酶存在于各种肠细菌中,包括:梭菌属、拟杆菌属、乳杆菌属、双歧杆菌属和肠球菌属[21]。在饮水添加氯化钙的高脂日粮饲喂小鼠中梭菌属显著增加,说明饮水添加氯化钙可能影响小鼠粪便菌群中胆汁酸在脂质代谢方面的作用,从而显著减少高脂日粮饲喂小鼠的脂肪含量。
综上所述,饮水添加氯化钙能缓解高脂日粮诱导的肥胖,降低体质量和体内脂肪含量,而这可能与饮水添加氯化钙提高小鼠粪便菌群多样性和特定菌群丰度有关。研究结果为深入了解钙离子调节机体脂肪沉积机制以及提高动物胴体品质和人体健康具有重要意义。
-
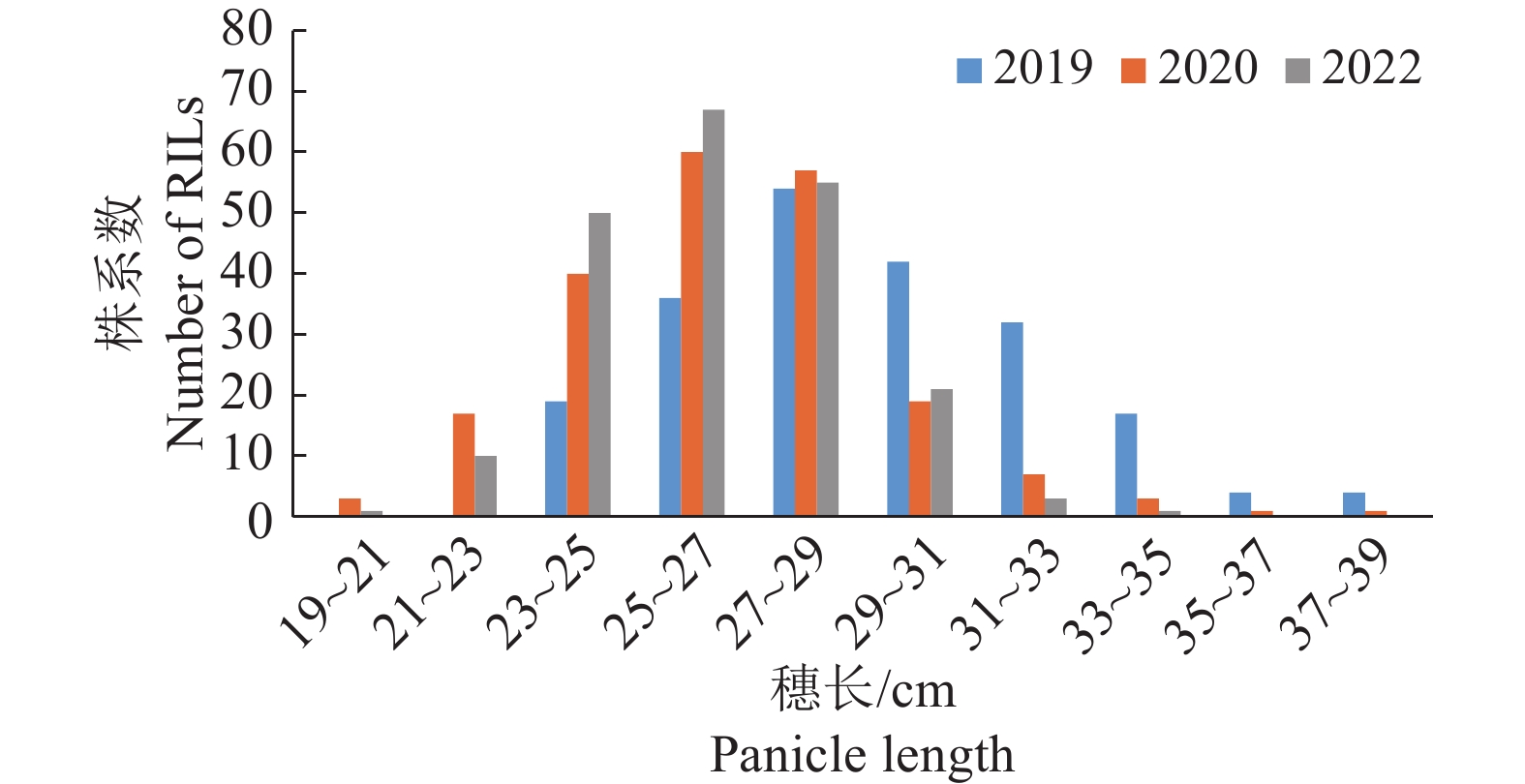
表 1 穗长在RIL群体中的分布情况
Table 1 Distribution of panicle length traits in RIL population
环境
Environment亲本穗长/cm
Parent panicle length重组自交系穗长
RIL panicle lengthZP37 R86051) 平均值/cm
Mean变幅/cm
Range偏度
Skewness峰度
Kurtosis变异系数/%
CV2019 23.10 37.56** 29.08 23.20~38.67 0.42 −0.13 0.11 2020 23.57 34.63** 26.50 19.87~37.20 0.50 1.04 0.11 2022 23.48 32.05** 26.29 20.86~33.80 0.29 0.18 0.08 1)“**”表示与‘ZP37’株系相比差异显著(P < 0.01,t 检验)
1) “**” indicates significant difference from ‘ZP37’ strains (P < 0.01, t test)表 2 不同环境下水稻穗长QTL 分析
Table 2 QTL analysis of panicle length under different environments
位点
QTL染色体
Chr.物理位置/bp
Physical position2019 2020 2022 LOD 加性效应
Additive
effect贡献率/%
PVELOD 加性效应
Additive
effect贡献率/%
PVELOD 加性效应
Additive
effect贡献率/%
PVEqPL3-1 3 6992384—7197429 3.11 −0.63 2.28 qPL3-2 3 7324223—7890278 3.43 −0.71 2.89 qPL4-1 4 20619799—20702562 6.22 −0.68 4.54 qPL4-2 4 24259618—24399641 3.81 −0.69 2.17 3.94 −0.68 2.66 qPL7-1 7 14356324—14982725 5.21 0.66 2.45 3.14 0.53 2.75 qPL7-2 7 17321645—17418744 3.12 0.53 2.77 qPL8-1 8 25112712—26010194 12.87 −1.39 10.94 6.33 −0.80 6.39 qPL8-2 8 27500184—27682717 5.99 −1.13 5.87 qPL9 9 20564403—20770874 3.22 −0.82 3.08 5.42 −0.70 4.88 qPL12-1 12 7167759—7401747 3.31 −0.94 4.02 qPL12-2 12 14722574—14918715 3.07 −0.77 2.70 表 3 穗长QTL的聚合效应分析1)
Table 3 Pyramiding effect of the QTLs for panicle length
株系类型 QTL RIL数量
No. of RILs不同年份穗长/cm Panicle length in different years qPL4-2 qPL8-1 qPL9 2019 2020 2022 Hap 1 + + + 24 32.57a 29.86a 28.75a Hap 2 − + + 18 29.48bc 27.15b 26.92bc Hap 3 + + − 17 30.39b 27.80b 27.23b Hap 4 + − + 34 29.63bc 26.81bc 26.91bc Hap 5 − + − 15 28.91bcd 26.34bcd 25.71cd Hap 6 − − + 30 28.47cde 25.61cde 25.62d Hap 7 + − − 37 27.89de 25.43de 25.57d Hap 8 − − − 20 26.91e 24.44e 24.19e 1) “+”和“−”分别表明含有和不含增效等位基因;同列数据后的不同小写字母表示相同环境下不同株系类型之间差异显著(P < 0.05, LSD法)
1) “+” and “−” indicate the presence and absence of favorable alleles respectively; Different lowercase letters of the same column indicate significant differences among different types of strains under the same environment (P < 0.05,LSD method) -
[1] XING Y, ZHANG Q. Genetic and molecular bases of rice yield[J]. Annual Review of Plant Biology, 2010, 61: 421-442. doi: 10.1146/annurev-arplant-042809-112209
[2] LI X, YAN W, AGRAMA H, et al. Mapping QTLs for improving grain yield using the USDA rice mini-core collection[J]. Planta, 2011, 234(2): 347-361. doi: 10.1007/s00425-011-1405-0
[3] LIU T, LI L, ZHANG Y, et al. Comparison of quantitative trait loci for rice yield, panicle length and spikelet density across three connected populations[J]. Journal of Genetics, 2011, 90(2): 377-382. doi: 10.1007/s12041-011-0083-9
[4] HUANG X, QIAN Q, LIU Z, et al. Natural variation at the DEP1 locus enhances grain yield in rice[J]. Nature Genetics, 2009, 41(4): 494-497. doi: 10.1038/ng.352
[5] LI S, QIAN Q, FU Z, et al. Short panicle1 encodes a putative PTR family transporter and determines rice panicle size[J]. The Plant Journal, 2009, 58(4): 592-605. doi: 10.1111/j.1365-313X.2009.03799.x
[6] LI F, LIU W, TANG J, et al. Rice DENSE AND ERECT PANICLE 2 is essential for determining panicle outgrowth and elongation[J]. Cell Research, 2010, 20(7): 838-849. doi: 10.1038/cr.2010.69
[7] QIAO Y, PIAO R, SHI J, et al. Fine mapping and candidate gene analysis of dense and erect panicle 3, DEP3, which confers high grain yield in rice (Oryza sativa L.)[J]. Theoretical and Applied Genetics, 2011, 122(7): 1439-1449. doi: 10.1007/s00122-011-1543-6
[8] LI M, TANG D, WANG K, et al. Mutations in the F-box gene LARGER PANICLE improve the panicle architecture and enhance the grain yield in rice[J]. Plant Biotechnology Journal, 2011, 9(9): 1002-1013. doi: 10.1111/j.1467-7652.2011.00610.x
[9] LI X, QIAN Q, FU Z, et al. Control of tillering in rice[J]. Nature, 2003, 422(6932): 618-621. doi: 10.1038/nature01518
[10] KOMATSU M, MAEKAWA M, SHIMAMOTO K, et al. The LAX1 and FRIZZY PANICLE 2 genes determine the inflorescence architecture of rice by controlling rachis-branch and spikelet development[J]. Developmental Biology, 2001, 231(2): 364-373. doi: 10.1006/dbio.2000.9988
[11] LU Z, YU H, XIONG G, et al. Genome-wide binding analysis of the transcription activator IDEAL PLANT ARCHITECTURE 1 reveals a complex network regulating rice plant architecture[J]. The Plant Cell, 2013, 25(10): 3743-3759. doi: 10.1105/tpc.113.113639
[12] YUAN H, QIN P, HU L, et al. OsSPL18 controls grain weight and grain number in rice[J]. Journal of Genetics and Genomics, 2019, 46(1): 41-51. doi: 10.1016/j.jgg.2019.01.003
[13] CHO Y, KANG H, LEE J, et al. Identification of quantitative trait loci in rice for yield, yield components, and agronomic traits across years and locations[J]. Crop Science, 2007, 47(6): 2403-2417. doi: 10.2135/cropsci2006.08.0509
[14] MARATHI B, GULERIA S, MOHAPATRA T, et al. QTL analysis of novel genomic regions associated with yield and yield related traits in new plant type based recombinant inbred lines of rice (Oryza sativa L.)[J]. BMC Plant Biology, 2012, 12(1): 137. doi: 10.1186/1471-2229-12-137
[15] WEI M, LUO T, HUANG D, et al. Construction of high-density genetic map and QTL mapping for grain shape in the rice RIL population[J/OL]. Plants, 2023, 12(16): 2911. https://doi.org/10.3390/plants12162911.
[16] MENG L, LI H, ZHANG L, et al. QTL IciMapping: Integrated software for genetic linkage map construction and quantitative trait locus mapping in biparental populations[J]. The Crop Journal, 2015, 3: 269-283. doi: 10.1016/j.cj.2015.01.001
[17] 王建康. 数量性状基因的完备区间作图方法[J]. 作物学报, 2009, 35(2): 239-245. [18] MCCOUCH S, KOCHERT G, YU Z, et al. Molecular mapping of rice chromosomes[J]. Theoretical and Applied Genetics, 1988, 76(6): 815-829. doi: 10.1007/BF00273666
[19] PAN Q, ALI F, YANG X, et al. Exploring the genetic characteristics of two recombinant inbred line populations via high-density SNP markers in maize[J]. PLoS One, 2012, 7(12): e52777. doi: 10.1371/journal.pone.0052777
[20] CHEN Z, WANG B, DONG X, et al. An ultra-high density bin-map for rapid QTL mapping for tassel and ear architecture in a large F2 maize population[J]. BMC Genomics, 2014, 15(1): 433. doi: 10.1186/1471-2164-15-433
[21] GOLICZ A, BAYER P, EDWARDS D. Skim-based genotyping by sequencing[M]//Methods in Molecular Biology. New York: Springer New York, 2014: 257-270.
[22] 张亚东, 梁文化, 赫 磊, 等. 水稻RIL群体高密度遗传图谱构建及粒型QTL定位[J]. 中国农业科学, 2021, 54(24): 5163-5176. [23] 宋博文, 王朝欢, 赵哲, 等. 基于高密度遗传图谱对水稻粒形QTL定位及分析[J]. 作物学报, 2022, 48(11): 2813-2829. [24] 闫晓霞, 朱满山, 王丰, 等. 利用高密度遗传图谱定位水稻耐低氧萌发QTL[J]. 广东农业科学, 2023, 50(4): 13-21. [25] 潘俊峰, 崔克辉, 刘彦卓, 等. 利用高密度Bin图谱定位水稻叶绿素含量QTL[J]. 广东农业科学, 2022, 49(9): 132-140. [26] ZHANG M, ZHOU Z, CHEN Y, et al. Finding new addictive QTL for yield traits based on a high-density genetic map in hybrid rice[J]. Plant Growth Regultion, 2021, 93: 105-115. doi: 10.1007/s10725-020-00669-2
[27] LI X, WU L, WANG J, et al. Genome sequencing of rice subspecies and genetic analysis of recombinant lines reveals regional yield- and quality-associated loci[J]. BMC Biology, 2018, 16: 102. doi: 10.1186/s12915-018-0572-x
[28] XING Z, TAN F, HUA P, et al. Characterization of the main effects, epistatic effects and their environmental interactions of QTLs on the genetic basis of yield traits in rice[J]. Theoretical and Applied Genetics, 2002, 105(2/3): 248-257. doi: 10.1007/s00122-002-0952-y
[29] 贾佩陇, 李 彪, 黎明辉, 等. 基于水稻染色体片段代换系的苗期耐低氮QTL分析[J]. 华南农业大学学报, 2019, 40(4): 16-24. [30] 陈燕华, 黄大辉, 邱永福, 等. 水稻主要农艺性状的QTL分析[J]. 华南农业大学学报, 2014, 35(5): 42-51. [31] 淳 雁, 李学勇. 水稻穗型的遗传调控研究进展[J]. 植物学报, 2017, 52(1): 19-29. [32] LIU E B, LIU Y, WU G C, et al. Identification of a candidate gene for panicle length in rice (Oryza sativa L.) via association and linkage analysis[J]. Frontiers in Plant Science, 2016, 7: 596.
[33] 徐华山, 孙永建, 周红菊, 等. 构建水稻优良恢复系背景的重叠片段代换系及其效应分析[J]. 作物学报, 2007, 33(6): 979-986. [34] 魏少博, PRIYA L, 王文生, 等. 两个水稻骨干恢复系重要农艺性状的遗传基础研究[J]. 植物遗传资源学报, 2017, 18(5): 801-809. [35] ZHANG L, WANG J, WANG J, et al. Quantitative trait locus analysis and fine mapping of the qPL6 locus for panicle length in rice[J]. Theoretical and Applied Genetics, 2015, 128(6): 1151-1161. doi: 10.1007/s00122-015-2496-y
[36] FUJITA D, TRIJATMIKO K, TAGLE A, et al. NAL1 allele from a rice landrace greatly increases yield in modern indica cultivars[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(51): 20431-20436.
[37] ZHANG G, LI S, WANG L, et al. LSCHL4 from Japonica cultivar, which is allelic to NAL1, increases yield of Indica super rice 93-11[J]. Molecular Plant, 2014(8): 1350-1364.
[38] KUMAR A, DIXIT S, RAM T, et al. Breeding high-yielding drought-tolerant rice: Genetic variations and conventional and molecular approaches[J]. Journal of Experimental Botany, 2014, 65(21): 6265-6278.
[39] 韦敏益, 黄大辉, 张月雄, 等. 分子标记辅助选育香型优质稻新品种‘桂野香占’[J/OL]. 分子植物育种, 2021: 1-18. (2021-09-28) [2023-07-01]. http://kns.cnki.net/kcms/detail/46.1068.S.20210928.0926.012.html. [40] ZONG G, WANG A, WANG L, et al. A pyramid breeding of eight grain-yield related quantitative trait loci based on marker-assistant and phenotype selection in rice (Oryza sativa L.)[J]. Journal of Genetics and Genomics, 2012, 39(7): 335-350. doi: 10.1016/j.jgg.2012.06.004
[41] MALLIKARJUNA S, AHMED H U, HENRY A, et al. Genetic, physiological, and gene expression analyses reveal that multiple QTL enhance yield of rice mega-variety IR64 under drought[J]. PLoS One, 2013, 8(5): e62795. doi: 10.1371/journal.pone.0062795
-
期刊类型引用(2)
1. 马春丽,赵林昀,扈瑞平,李丽,曹丽丽,红梅,包玉龙. 钙离子对高脂饮食小鼠肠道菌群的影响. 畜牧与饲料科学. 2025(01): 1-9 .  百度学术
百度学术
2. 李玉萍,田晶晶,张凯,夏耘,王广军,郁二蒙,李志斐,龚望宝,谢骏. 皇竹草对草鱼脂肪蓄积及肠道菌群组成的影响. 上海海洋大学学报. 2022(04): 915-928 .  百度学术
百度学术
其他类型引用(1)




 下载:
下载: