Detection and analysis of QTL for panicle length in rice using a high-density genetic map
-
摘要:目的
深入挖掘与穗长相关的新基因,为水稻穗长调控的遗传机理研究及分子育种提供依据。
方法以2个优良亲本‘ZP37’和‘R8605’及其杂交衍生的208个高世代重组自交系(Recombinant inbred lines,RILs)为作图群体,利用全基因组重测序高密度连锁图谱对3个不同环境下的穗长数量性状座位(Quantitative trait locus,QTL)进行定位,同时分析它们的聚合效应。
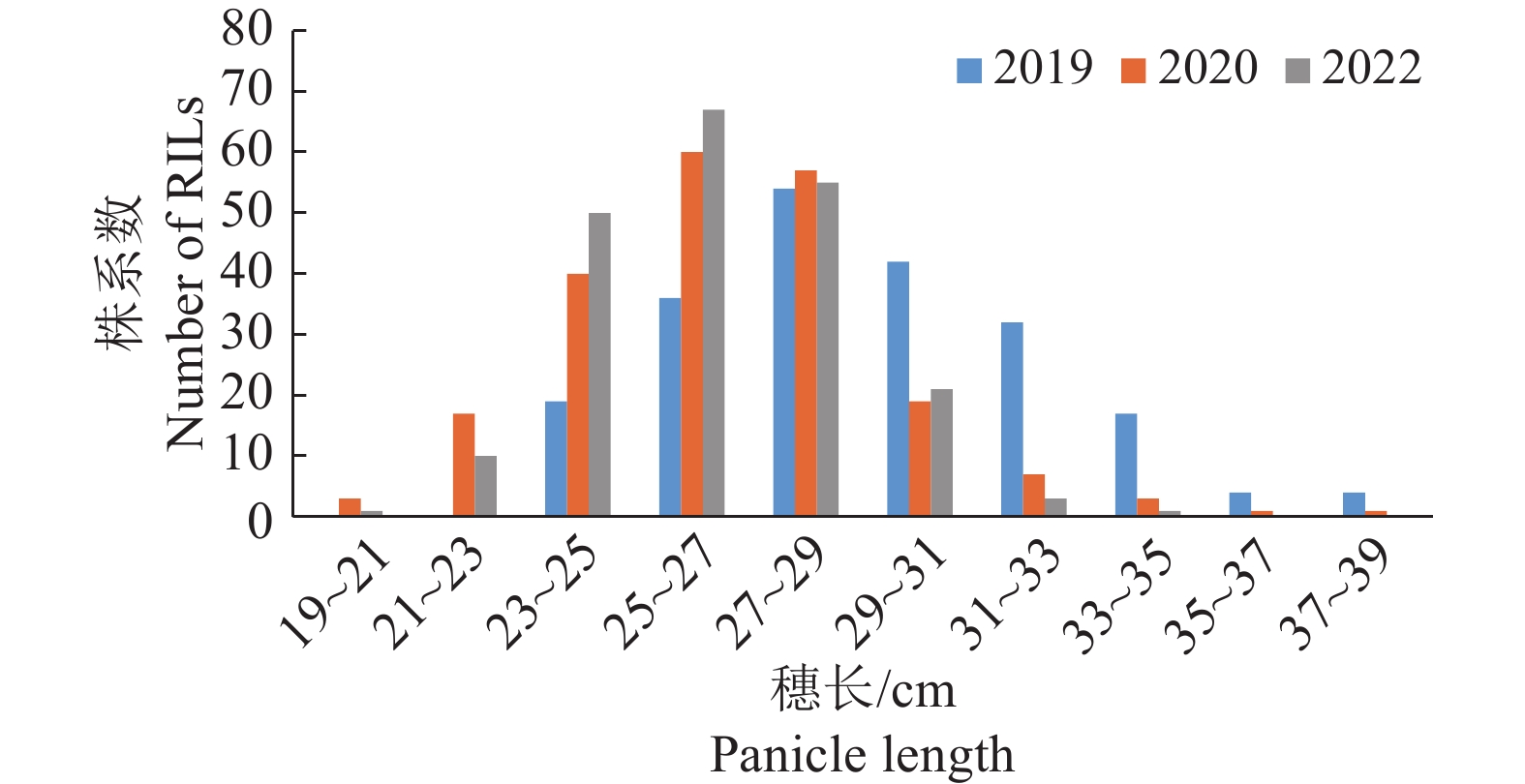
结果共检测到11个穗长QTL,分别分布在第3、4、7、8、9和12号染色体上,其似然函数比对数值(Log of odds,LOD)介于3.07~12.87之间,贡献率在2.17%~10.94%之间,有7个QTL是新位点,其余4个QTL位点与已报道的穗长基因和QTL位置重叠或相近。在2个不同环境下重复检测到4个稳定的QTL位点;对聚合了不同数量穗长QTL株系的分析结果表明,穗长QTL表现出累加效应,QTL数量的增加能显著增加水稻穗长。
结论本研究结果为水稻穗长QTL的克隆和功能解析奠定坚实的基础,为水稻高产育种提供理论依据和遗传资源。
Abstract:ObjectiveTo deeply explore new genes related to panicle length and provide a basis for the study of genetic mechanism of panicle length regulation and molecular breeding in rice.
MethodTwo superior parents, ‘ZP37’ and ‘R8605’, as well as 208 recombinant inbred lines (RILs) derived from the cross of ZP37/R8605 were used as a mapping population to locate quantitative trait loci (QTLs) for panicle length in three different environments through the high-density linkage map of whole genome resequencing, and to analyze their pyramiding effects.
ResultA total of 11 QTLs for panicle length were detected on chromosomes 3, 4, 7, 8, 9 and 12, with the logs of odds (LODs) ranging from 3.07 to 12.87 and contribution rates ranging from 2.17% to 10.94%, seven of the QTLs were new loci, and the remaining four QTLs overlapped or were close to the reported panicle length genes and QTLs. Among them, four stable QTLs were detected repeatedly in two different environments, and by analyzing the lines that pyramiding different numbers of panicle length QTLs, the results showed that the panicle length QTLs showed an additive effect, and the increase in the number of QTLs significantly increased the panicle length of rice.
ConclusionThe results of this study provide a solid foundation for cloning and functional analysis of rice panicle length QTLs, as well as a theoretical basis and genetic resources for high-yield rice breeding.
-
Keywords:
- Oryza sativa L. /
- Spike length /
- QTL mapping /
- High-density genetic map
-
柑橘黄龙病(Huanglongbing, HLB)是目前影响柑橘产业最严重的病害之一,在我国已有近百年历史[1-2]。广东省是柑橘种植的主要省份之一,但受黄龙病的影响,柑橘种植面积开始下降,种植区域向广西转移[3]。多聚酶链反应(Polymerase chain reaction, PCR)是目前应用于柑橘黄龙病识别的一种较为可靠且普遍的技术,但该方法存在的问题是难以实现实时在线检测。深度学习在图像分类和回归方面有较高的准确性,其精度优于目前现有的其他图像处理技术,已广泛应用于农业、医疗等领域。
邓小玲等[4]提出了基于高光谱成像和Fisher判别法的柑橘黄龙病识别方法,柑橘黄龙病病情识别准确率为90%。邓小玲等[5]通过荧光检测技术获取柑橘叶片荧光参数,提出了利用概率神经网络对黄龙病柑橘植株分类建模,诊断准确率为76.93%。马淏等[6]提出了利用高光谱成像技术生成的光谱及纹理特征进行柑橘黄龙病识别,结合最小二乘法和SVM分类器建立分类模型,平均预测准确率为88.5%。王克健[7]提出了采用高光谱检测与无人机技术采集柑橘黄龙病成像的方法,通过柑橘黄龙病的特征光谱建立了柑橘黄龙病识别模型,识别准确率为87%。肖怀春[8]提出一种基于高光谱成像技术的柑橘黄龙病叶片可视化判别模型,通过提取特征波长识别患病叶片的纹理特征,最低误判率为3.12%。戴泽翰等[9]通过深度卷积神经网络建立柑橘黄龙病识别模型,提出4种针对8类柑橘黄龙病症状的分类器,其中最优分类器的识别准确率为93.7%。综上所述,目前针对柑橘黄龙病的无损检测多采用高光谱检测技术,但是该检测技术尚未完善,在成分波长数量较少的情况下,准确率较低[10],而且前期硬件投入成本较高,难以大范围推广。
本研究利用深度学习卷积神经网络,研究柑橘黄龙病的在线无损识别方法,解决传统柑橘黄龙病人工识别困难、智能化程度低的问题。利用卷积神经网络[11-12]自动提取特征的特点,在Inception网络架构基础上,进行深度可分离卷积(Depthwise separable convolution,DSC)设计,提出一种基于卷积神经网络的柑橘黄龙病识别模型——X-ResNeXt模型。该模型在传统数据增强方法基础上,采用Mixup算法建立样本之间的线性关系,增强模型识别数据样本的鲁棒性;通过迁移Xception网络在ImageNet数据集上的先验知识与动量梯度下降优化方法,有效提高模型的准确率与收敛速度。X-ResNeXt模型在保持不增加网络参数的同时拓宽模型网络结构,在柑橘黄龙病的无损检测方面有一定的可行性和先进性。
1. 模型设计
1.1 数据采集
数据采集于2018年11月—2019年1月在广西壮族自治区贺州市昭平县北陀镇柑橘种植园进行。数据采集对象为初期显现病状的成年病树果实以及同期健康果实,采集设备为手持移动设备,采集距离为距柑橘果实1 m处。采集试验结束,共采集560张分辨率为1920×1080的柑橘果实图像样本(图1),其中,患病和健康柑橘果实图片分别为224和336张。采用3∶1∶1的比例将数据集划分为训练集(336张)、验证集(112张)和测试集(112张)。为了规范数据集使用,利用中心裁剪法把原始图像分辨率固定为299×299。后期,训练集将对模型进行数据样本拟合,然后利用验证集调整模型超参数以及对模型性能进行初评估;最后将测试集输入已训练完毕的卷积神经网络模型中,得出最终试验结果。
1.2 数据增强
1.2.1 同类数据增强
为提高模型训练的鲁棒性和泛化能力,本研究利用Imgaug数据增强库对采集的柑橘黄龙病原始图像数据集进行同类数据增强。增强方法包括水平翻转、垂直翻转、旋转、仿射、添加高斯噪声等[13-14]。利用Fliplr函数、Flipud函数对柑橘图像进行水平和垂直翻转;利用rotate函数对柑橘图像进行45°旋转;利用Affine函数对柑橘图像进行图像仿射。
经增强后的柑橘黄龙病图像数据集图像最终数量为7168张,图2为部分数据增强结果图。
1.2.2 Mixup混类数据增强
同类数据增强方法设定邻域内的样本属于同一类,不对非同类类别的样本做邻域关系的建模,该方法虽然能增强模型的泛化能力,但是严重依赖于原始数据集。本研究采用Mixup [15] 混类数据增强方法,以贝塔分布混合多类别数据,线性化处理样本与样本间区域,寻求增强模型预测训练样本以外的数据集的适应性,进一步提升模型对样本识别的鲁棒性以及错误类别的判断能力。图3为不同混合系数(
$ \lambda $ )的Mixup算法进行柑橘黄龙病数据集增强的结果示意图。1.3 X-ResNeXt模型的设计
1.3.1 X-ResNeXt模型网络结构设计
He等[16]提出了将152层深的ResNet神经网络引入Shortcut结构,以解决极深的神经网络难以训练的问题。Xie等[17]在ResNet神经网络的基础上提出了ResNeXt网络,其核心思想也是通过加深或加宽网络结构以寻求模型准确率的提高。但是随着模型网络结构的加深或者加宽,网络参数将急剧增多,网络计算开销也大大增加。
X-ResNeXt模型核心设计思想是在Inception网络架构基础上,进行深度可分离卷积设计,达到网络参数不增多的情况下拓宽模型网络结构的目的,以实现提高模型识别准确率的同时还具备良好的运算处理性能。该模型的具体网络结构由Xception网络[18]与ResNeXt模块融合构成,具体为:输入层的输入图像尺度为299×299×3;卷积层为深度可分离卷积模块,在1×1卷积之后,对每个通道做3×3的独立卷积;根据迁移学习的思想,冰冻Xception中间层;池化层均采用最大池化,核大小为3×3,滑动步长均为1,填充方式均为SAME;输出层采用全局平均池化层;卷积神经网络激活函数均采用ReLU函数;中间采用权重连接方式,连接上下层;最后采用ResNeXt模块替代Xception中最后一层输出层,实现了模型层数的加深以及特征的融合。图4为X-ResNeXt模型的网络结构图。
1.3.2 模型优化
为解决X-ResNeXt模型随着网络深度加深出现一定程度的梯度弥散,进一步提升模型性能,本研究迁移Xception网络[19]在ImageNet数据集[20]上的先验知识,进行模型的微调(Finetune)优化,挖掘领域间共享的潜在特征和结构;采用动量梯度下降法(Momentum gradient descent, MGD)[21]进行参数更新,降低模型收敛过程的震荡现象,进一步提高识别准确率。
图5为X-ResNeXt模型的训练流程图。首先,由原始图像数据集生成同类增强数据集,再结合Mixup算法对数据集建立线性关系,进行尺寸裁剪。然后,迁移ImageNet数据集上的先验知识对模型进行微调。再而,设置训练数据批次大小为32×2,学习率为0.1,进行X-ResNeXt模型训练。保存最佳权重文件以及模型结构,动量大小设置为0.9,当运行每5个epoch后,模型验证集损失值停止下降时,学习率设置为原来的一半。最后,权重参数收敛后,即可得到最优的X-ResNeXt柑橘黄龙病识别模型。
迁移学习是一类复用跨领域学习知识,弱化样本数据特征提取不足,改进目标领域或任务学习效果的机器学习方法。ImageNet是目前图像识别最大的数据库,超过了1 400万张图像。本研究保留已训练完毕的Xception模块中所有卷积层参数,并将其冰冻;利用迁移学习方法,在最后一层全连接层借鉴模型在ImageNet数据集上优化训练所得的先验知识,更新最后一层的权重参数,以期提高模型训练速度与准确率。
模型使用梯度下降法不断逼近损失函数最优化点时,有可能会越过最低点,产生在最低点来回震荡的现象。为了提高模型参数更新的稳定性,本研究提出MGD进行参数的优化更新。动量梯度下降法的设计思想是调整函数在纵轴下降的速度,从横轴方向趋向最低点。实际操作是通过计算梯度的指数加权平均数进行权重参数的更新,在横轴给予横向速度逼近最优点。动量梯度下降法缓解了随机梯度下降法 (Stochastic gradient descent, SGD)和批量随机梯度下降法的震荡问题,减弱了模型收敛过程的震荡影响。
1.4 试验方法
1.4.1 总体试验方案
本研究首先评估数据集进行图像预处理对模型性能的影响,即决定是否对数据集进行预处理。然后进行数据同类增强对模型性能影响的测试试验,获取图像数据集经同类数据增强前后,模型性能的变化结果,以证明对图像数据集进行同类数据增强的必要性。最后,X-ResNeXt模型通过迁徙学习试验、Mixup混类数据增强试验和梯度下降优化对比试验寻求模型的最佳性能指标,从而获得最优收敛模型。通过与经典识别模型的对比试验,最终验证模型的先进性和优越性。
1.4.2 图像预处理对模型性能影响的评估试验
由于光线、拍摄角度和光在传播时经过雾、霾等介质以及粒子的散射作用,图像传感器实际作业所采集的图像信息经常发生降质现象[22]。另外,果实和树叶之间相互遮挡,导致柑橘果实图像表面发生模糊和亮度不足的现象。为提高果实的识别率,研究一般采用直方图均衡化和图像锐化操作,进行柑橘图像的轮廓补偿[23],增强图像边缘位置颜色的对比度,以提高图像的清晰度。但是,本研究的目的是柑橘黄龙病的识别,直方图均衡化和图像锐化预处理方法,很容易造成原始图片的对比度过分增强,图片色彩失真,如图6所示,从而造成柑橘果实黄龙病表型特征的严重丢失,对模型训练产生误导作用。因此,应进行图像预处理对模型性能影响的评估试验,以决定是否对图像数据集进行预处理。
2. 模型性能影响相关试验与结果
试验基于TensorFlow后端的Keras框架进行,计算机硬件环境如下:处理器为Intel 6800k CPU,内存为32 G,GPU为 GTX 1080Ti ×2;软件操作系统为Centos7系统。评估结果采用准确率、召回率、F1值、损失值作为主要评价指标。
2.1 图像预处理对模型性能影响的评估试验
分别采用无预处理、直方均衡化预处理和图像锐化预处理的原始数据集训练X-ResNeXt柑橘黄龙病识别模型。图像数据集未作预处理时,模型的可信度为63.12%,数据集利用直方图均衡化和图像锐化分别进行预处理操作后,模型的可信度分别降低了7.91%和10.29%。可见,针对本研究的柑橘果实黄龙病识别任务,对图像数据集进行图像直方均衡化和锐化预处理操作将会对结果产生负面影响,因此本研究后续试验未对图像数据集进行预处理。
2.2 数据同类增强对模型性能的影响
分别采用原始数据集和经数据同类增强的数据集训练X-ResNeXt柑橘黄龙病识别模型,并对比2组模型的性能指标。数据集进行数据同类增强,后训练所得的模型性能指标准确率为91.38%,召回率为90.42%,F1值为0.909,与原始数据集训练所得的模型相关指标相比,分别提高了5.85%、5.96%和0.059。试验结果表明,对训练集进行数据同类增强,可在原始数据基础上增加样本特征的多样性,提高模型训练结果的准确率,体现了神经网络模型性能与训练样本数呈线性正相关的关系。从另一个角度,也说明后期随着柑橘黄龙病果实样本数的增加,模型的识别性能也将有进一步的提升。
2.3 迁移学习
利用迁移学习对X-ResNeXt模型进行微调优化,将ImageNet数据集上训练所得的权重知识迁移到柑橘黄龙病识别分类任务上。设置Dropout参数为0.5,学习率为0.1,mini-batch为32×2=64。由迁移学习的试验结果可见,模型在ImageNet数据集训练得到的权重和偏置参数经验基础上进行训练,模型训练时间由5 220 s减少为4 788 s,表明相同条件下,迁移学习提高了柑橘黄龙病识别模型的训练速度。与无迁移学习训练的模型相比,迁移学习的识别准确率、召回率分别提高了0.61%和0.34%,F1值提高了0.006。表明在柑橘黄龙病数据集规模较小的情况下,迁移学习可加快模型收敛速度,获取局部更优化权重值,有助于提升柑橘黄龙病识别任务的相关指标。
2.4 Mixup混类数据增强对模型性能的影响
在迁移学习和L2正则化等优化策略基础上,进行不同超参数条件下的Mixup混类数据增强对模型性能影响的试验。设置Dropout参数为0.5,mini-batch为32×2=64,正则化系数为0.02,学习率为0.1,混合系数λ分别设置为1.0、0.8、0.4、0.2和0。由表1可知,当λ为0.4时,X-ResNeXt模型表现最优,模型的准确率、召回率、F1值分别为93.74%、92.58%和0.932。在传统数据增强和迁移学习的基础上,这3个指标分别上升了1.77%、1.82%和0.018。试验表明,Mixup混类数据增强方法有利于提高模型识别图像样本的适应性,提升模型判别柑橘黄龙病病果的能力。
表 1 Mixup混类数据增强对模型性能的影响Table 1. Effects of Mixup mixed data enhancement on model performance模型大小 Model batchsize λ 准确率/% Accuracy rate 召回率/% Recall rate F1指标 F1 index 32×2 1.0 91.97 90.76 0.914 32×2 0.8 92.17 91.25 0.917 32×2 0.4 93.74 92.58 0.932 32×2 0.2 93.31 92.17 0.927 32×2 0 91.88 90.79 0.913 2.5 动量梯度下降优化试验
为验证X-ResNeXt模型动量梯度下降优化方法的有效性,模型分别采用MGD和SGD进行识别对比试验。设置学习率为0.1,L2正则化系数为0.01,Dropout参数默认为0.5,mini-batch为32×2=64,λ取0.4,MGD的动量值分别取0、0.5和0.9。由表2可见,模型采用MGD 动量值取0.9时,识别准确率、召回率和F1值最高。表明模型经MGD优化后,参数更接近于局部最优值,模型能更有效地收敛,从而实现模型对柑橘黄龙病病果识别能力的进一步提升。
表 2 模型梯度下降优化方法对比试验Table 2. Comparison test of model optimization methods based on gradient descent优化方法
Optimization method动量
Momentum准确率/%
Accuracy rate召回率/%
Recall rateF1指标
F1 index随机梯度下降法 Stochastic gradient descent (SGD) 93.53 92.42 0.930 动量梯度下降法 Momentum gradient descent (MGD) 0 93.53 92.42 0.930 0.5 93.74 92.58 0.932 0.9 94.29 93.69 0.939 2.6 算法对比试验
选择同等条件下训练所得的Inception-V3和Xception模型进行测试试验,与X-ResNeXt模型测试结果进行比较。X-ResNeXt模型最终收敛的准确率(94.29%)优于Xception模型(92.78%)和Inception-V3模型(90.31%);X-ResNeXt模型最终收敛的召回率(93.69%)也优于Xception模型(91.89%)和Inception-V3模型(89.23%)。结果表明,与Inception-V3和Xception模型相比,X-ResNeXt模型加深了网络的深度,拓宽了网络结构的宽度,表达柑橘黄龙病的果实特征更加全面,模型泛化程度更高。
3. 结论
柑橘出现红鼻子果是柑橘患黄龙病的初期症状,对该时期的柑橘黄龙病果实进行快速准确识别将进一步提高柑橘园区的产量和产品质量。本研究以柑橘黄龙病初期患病果实作为研究对象,应用卷积神经网络设计和构建一种基于Inception网络架构的柑橘黄龙病果实识别模型:X-ResNeXt模型,试验结论如下:
1)在数据量较少情况下,降低模型复杂度并迁移已有先验知识,有助于模型性能提升,说明正则化、迁移学习等模型优化策略可为柑橘黄龙病果实识别与分类模型提供高效的训练途径,可解决深度学习网络模型训练成本大、耗费资源多的问题。
2) Mixup混类数据增强方法有利于提高模型识别柑橘黄龙病果实图像样本的适应性,提升柑橘黄龙病识别模型性能,表明该新型数据增强方法在扩充数据集的同时,也提高了神经网络的泛化能力。本文将Mixup方法应用到农业智能化系统领域,并实现了对模型的优化,该结合方法可推广到未来的智慧农业研究中,进一步提升对农情监测的时效性。
3) 在准确率与召回率指标上X-ResNeXt模型优于经典识别模型,充分表明利用深度学习方法对柑橘黄龙病果实进行识别是判断柑橘植株是否患病的一种可行且可靠的方式,可为柑橘黄龙病的高精度、快速无损识别提供有益参考。
-
表 1 穗长在RIL群体中的分布情况
Table 1 Distribution of panicle length traits in RIL population
环境
Environment亲本穗长/cm
Parent panicle length重组自交系穗长
RIL panicle lengthZP37 R86051) 平均值/cm
Mean变幅/cm
Range偏度
Skewness峰度
Kurtosis变异系数/%
CV2019 23.10 37.56** 29.08 23.20~38.67 0.42 −0.13 0.11 2020 23.57 34.63** 26.50 19.87~37.20 0.50 1.04 0.11 2022 23.48 32.05** 26.29 20.86~33.80 0.29 0.18 0.08 1)“**”表示与‘ZP37’株系相比差异显著(P < 0.01,t 检验)
1) “**” indicates significant difference from ‘ZP37’ strains (P < 0.01, t test)表 2 不同环境下水稻穗长QTL 分析
Table 2 QTL analysis of panicle length under different environments
位点
QTL染色体
Chr.物理位置/bp
Physical position2019 2020 2022 LOD 加性效应
Additive
effect贡献率/%
PVELOD 加性效应
Additive
effect贡献率/%
PVELOD 加性效应
Additive
effect贡献率/%
PVEqPL3-1 3 6992384—7197429 3.11 −0.63 2.28 qPL3-2 3 7324223—7890278 3.43 −0.71 2.89 qPL4-1 4 20619799—20702562 6.22 −0.68 4.54 qPL4-2 4 24259618—24399641 3.81 −0.69 2.17 3.94 −0.68 2.66 qPL7-1 7 14356324—14982725 5.21 0.66 2.45 3.14 0.53 2.75 qPL7-2 7 17321645—17418744 3.12 0.53 2.77 qPL8-1 8 25112712—26010194 12.87 −1.39 10.94 6.33 −0.80 6.39 qPL8-2 8 27500184—27682717 5.99 −1.13 5.87 qPL9 9 20564403—20770874 3.22 −0.82 3.08 5.42 −0.70 4.88 qPL12-1 12 7167759—7401747 3.31 −0.94 4.02 qPL12-2 12 14722574—14918715 3.07 −0.77 2.70 表 3 穗长QTL的聚合效应分析1)
Table 3 Pyramiding effect of the QTLs for panicle length
株系类型 QTL RIL数量
No. of RILs不同年份穗长/cm Panicle length in different years qPL4-2 qPL8-1 qPL9 2019 2020 2022 Hap 1 + + + 24 32.57a 29.86a 28.75a Hap 2 − + + 18 29.48bc 27.15b 26.92bc Hap 3 + + − 17 30.39b 27.80b 27.23b Hap 4 + − + 34 29.63bc 26.81bc 26.91bc Hap 5 − + − 15 28.91bcd 26.34bcd 25.71cd Hap 6 − − + 30 28.47cde 25.61cde 25.62d Hap 7 + − − 37 27.89de 25.43de 25.57d Hap 8 − − − 20 26.91e 24.44e 24.19e 1) “+”和“−”分别表明含有和不含增效等位基因;同列数据后的不同小写字母表示相同环境下不同株系类型之间差异显著(P < 0.05, LSD法)
1) “+” and “−” indicate the presence and absence of favorable alleles respectively; Different lowercase letters of the same column indicate significant differences among different types of strains under the same environment (P < 0.05,LSD method) -
[1] XING Y, ZHANG Q. Genetic and molecular bases of rice yield[J]. Annual Review of Plant Biology, 2010, 61: 421-442. doi: 10.1146/annurev-arplant-042809-112209
[2] LI X, YAN W, AGRAMA H, et al. Mapping QTLs for improving grain yield using the USDA rice mini-core collection[J]. Planta, 2011, 234(2): 347-361. doi: 10.1007/s00425-011-1405-0
[3] LIU T, LI L, ZHANG Y, et al. Comparison of quantitative trait loci for rice yield, panicle length and spikelet density across three connected populations[J]. Journal of Genetics, 2011, 90(2): 377-382. doi: 10.1007/s12041-011-0083-9
[4] HUANG X, QIAN Q, LIU Z, et al. Natural variation at the DEP1 locus enhances grain yield in rice[J]. Nature Genetics, 2009, 41(4): 494-497. doi: 10.1038/ng.352
[5] LI S, QIAN Q, FU Z, et al. Short panicle1 encodes a putative PTR family transporter and determines rice panicle size[J]. The Plant Journal, 2009, 58(4): 592-605. doi: 10.1111/j.1365-313X.2009.03799.x
[6] LI F, LIU W, TANG J, et al. Rice DENSE AND ERECT PANICLE 2 is essential for determining panicle outgrowth and elongation[J]. Cell Research, 2010, 20(7): 838-849. doi: 10.1038/cr.2010.69
[7] QIAO Y, PIAO R, SHI J, et al. Fine mapping and candidate gene analysis of dense and erect panicle 3, DEP3, which confers high grain yield in rice (Oryza sativa L.)[J]. Theoretical and Applied Genetics, 2011, 122(7): 1439-1449. doi: 10.1007/s00122-011-1543-6
[8] LI M, TANG D, WANG K, et al. Mutations in the F-box gene LARGER PANICLE improve the panicle architecture and enhance the grain yield in rice[J]. Plant Biotechnology Journal, 2011, 9(9): 1002-1013. doi: 10.1111/j.1467-7652.2011.00610.x
[9] LI X, QIAN Q, FU Z, et al. Control of tillering in rice[J]. Nature, 2003, 422(6932): 618-621. doi: 10.1038/nature01518
[10] KOMATSU M, MAEKAWA M, SHIMAMOTO K, et al. The LAX1 and FRIZZY PANICLE 2 genes determine the inflorescence architecture of rice by controlling rachis-branch and spikelet development[J]. Developmental Biology, 2001, 231(2): 364-373. doi: 10.1006/dbio.2000.9988
[11] LU Z, YU H, XIONG G, et al. Genome-wide binding analysis of the transcription activator IDEAL PLANT ARCHITECTURE 1 reveals a complex network regulating rice plant architecture[J]. The Plant Cell, 2013, 25(10): 3743-3759. doi: 10.1105/tpc.113.113639
[12] YUAN H, QIN P, HU L, et al. OsSPL18 controls grain weight and grain number in rice[J]. Journal of Genetics and Genomics, 2019, 46(1): 41-51. doi: 10.1016/j.jgg.2019.01.003
[13] CHO Y, KANG H, LEE J, et al. Identification of quantitative trait loci in rice for yield, yield components, and agronomic traits across years and locations[J]. Crop Science, 2007, 47(6): 2403-2417. doi: 10.2135/cropsci2006.08.0509
[14] MARATHI B, GULERIA S, MOHAPATRA T, et al. QTL analysis of novel genomic regions associated with yield and yield related traits in new plant type based recombinant inbred lines of rice (Oryza sativa L.)[J]. BMC Plant Biology, 2012, 12(1): 137. doi: 10.1186/1471-2229-12-137
[15] WEI M, LUO T, HUANG D, et al. Construction of high-density genetic map and QTL mapping for grain shape in the rice RIL population[J/OL]. Plants, 2023, 12(16): 2911. https://doi.org/10.3390/plants12162911.
[16] MENG L, LI H, ZHANG L, et al. QTL IciMapping: Integrated software for genetic linkage map construction and quantitative trait locus mapping in biparental populations[J]. The Crop Journal, 2015, 3: 269-283. doi: 10.1016/j.cj.2015.01.001
[17] 王建康. 数量性状基因的完备区间作图方法[J]. 作物学报, 2009, 35(2): 239-245. [18] MCCOUCH S, KOCHERT G, YU Z, et al. Molecular mapping of rice chromosomes[J]. Theoretical and Applied Genetics, 1988, 76(6): 815-829. doi: 10.1007/BF00273666
[19] PAN Q, ALI F, YANG X, et al. Exploring the genetic characteristics of two recombinant inbred line populations via high-density SNP markers in maize[J]. PLoS One, 2012, 7(12): e52777. doi: 10.1371/journal.pone.0052777
[20] CHEN Z, WANG B, DONG X, et al. An ultra-high density bin-map for rapid QTL mapping for tassel and ear architecture in a large F2 maize population[J]. BMC Genomics, 2014, 15(1): 433. doi: 10.1186/1471-2164-15-433
[21] GOLICZ A, BAYER P, EDWARDS D. Skim-based genotyping by sequencing[M]//Methods in Molecular Biology. New York: Springer New York, 2014: 257-270.
[22] 张亚东, 梁文化, 赫 磊, 等. 水稻RIL群体高密度遗传图谱构建及粒型QTL定位[J]. 中国农业科学, 2021, 54(24): 5163-5176. [23] 宋博文, 王朝欢, 赵哲, 等. 基于高密度遗传图谱对水稻粒形QTL定位及分析[J]. 作物学报, 2022, 48(11): 2813-2829. [24] 闫晓霞, 朱满山, 王丰, 等. 利用高密度遗传图谱定位水稻耐低氧萌发QTL[J]. 广东农业科学, 2023, 50(4): 13-21. [25] 潘俊峰, 崔克辉, 刘彦卓, 等. 利用高密度Bin图谱定位水稻叶绿素含量QTL[J]. 广东农业科学, 2022, 49(9): 132-140. [26] ZHANG M, ZHOU Z, CHEN Y, et al. Finding new addictive QTL for yield traits based on a high-density genetic map in hybrid rice[J]. Plant Growth Regultion, 2021, 93: 105-115. doi: 10.1007/s10725-020-00669-2
[27] LI X, WU L, WANG J, et al. Genome sequencing of rice subspecies and genetic analysis of recombinant lines reveals regional yield- and quality-associated loci[J]. BMC Biology, 2018, 16: 102. doi: 10.1186/s12915-018-0572-x
[28] XING Z, TAN F, HUA P, et al. Characterization of the main effects, epistatic effects and their environmental interactions of QTLs on the genetic basis of yield traits in rice[J]. Theoretical and Applied Genetics, 2002, 105(2/3): 248-257. doi: 10.1007/s00122-002-0952-y
[29] 贾佩陇, 李 彪, 黎明辉, 等. 基于水稻染色体片段代换系的苗期耐低氮QTL分析[J]. 华南农业大学学报, 2019, 40(4): 16-24. [30] 陈燕华, 黄大辉, 邱永福, 等. 水稻主要农艺性状的QTL分析[J]. 华南农业大学学报, 2014, 35(5): 42-51. [31] 淳 雁, 李学勇. 水稻穗型的遗传调控研究进展[J]. 植物学报, 2017, 52(1): 19-29. [32] LIU E B, LIU Y, WU G C, et al. Identification of a candidate gene for panicle length in rice (Oryza sativa L.) via association and linkage analysis[J]. Frontiers in Plant Science, 2016, 7: 596.
[33] 徐华山, 孙永建, 周红菊, 等. 构建水稻优良恢复系背景的重叠片段代换系及其效应分析[J]. 作物学报, 2007, 33(6): 979-986. [34] 魏少博, PRIYA L, 王文生, 等. 两个水稻骨干恢复系重要农艺性状的遗传基础研究[J]. 植物遗传资源学报, 2017, 18(5): 801-809. [35] ZHANG L, WANG J, WANG J, et al. Quantitative trait locus analysis and fine mapping of the qPL6 locus for panicle length in rice[J]. Theoretical and Applied Genetics, 2015, 128(6): 1151-1161. doi: 10.1007/s00122-015-2496-y
[36] FUJITA D, TRIJATMIKO K, TAGLE A, et al. NAL1 allele from a rice landrace greatly increases yield in modern indica cultivars[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(51): 20431-20436.
[37] ZHANG G, LI S, WANG L, et al. LSCHL4 from Japonica cultivar, which is allelic to NAL1, increases yield of Indica super rice 93-11[J]. Molecular Plant, 2014(8): 1350-1364.
[38] KUMAR A, DIXIT S, RAM T, et al. Breeding high-yielding drought-tolerant rice: Genetic variations and conventional and molecular approaches[J]. Journal of Experimental Botany, 2014, 65(21): 6265-6278.
[39] 韦敏益, 黄大辉, 张月雄, 等. 分子标记辅助选育香型优质稻新品种‘桂野香占’[J/OL]. 分子植物育种, 2021: 1-18. (2021-09-28) [2023-07-01]. http://kns.cnki.net/kcms/detail/46.1068.S.20210928.0926.012.html. [40] ZONG G, WANG A, WANG L, et al. A pyramid breeding of eight grain-yield related quantitative trait loci based on marker-assistant and phenotype selection in rice (Oryza sativa L.)[J]. Journal of Genetics and Genomics, 2012, 39(7): 335-350. doi: 10.1016/j.jgg.2012.06.004
[41] MALLIKARJUNA S, AHMED H U, HENRY A, et al. Genetic, physiological, and gene expression analyses reveal that multiple QTL enhance yield of rice mega-variety IR64 under drought[J]. PLoS One, 2013, 8(5): e62795. doi: 10.1371/journal.pone.0062795
-
期刊类型引用(23)
1. 池美香,陈韶萍,黄婷,陈世雄,梁勇,邱荣洲. 柑橘黄龙病田间症状识别图像数据集. 中国科学数据(中英文网络版). 2025(01): 52-61 .  百度学术
百度学术
2. 骆润玫,殷惠莉,刘伟康,胡凯,廖飞,刘泽乾,曹亚芃,李强,王卫星. 基于YOLOv5-C的广佛手病虫害识别. 华南农业大学学报. 2023(01): 151-160 .  本站查看
本站查看
3. 殷献博,邓小玲,兰玉彬,陈欣. 基于改进YOLOX-Nano算法的柑橘梢期长势智能识别. 华南农业大学学报. 2023(01): 142-150 .  本站查看
本站查看
4. 梁生珺,于明鑫. 应用于无人机平台的轻量Transformer排水口检测框架. 电子技术与软件工程. 2023(01): 165-168 .  百度学术
百度学术
5. 汪莹,刘静远,吕文刚,龙海,刘宵宵,余道坚,方毅,郑铭森,张浩,陈冬美,高瑞芳. 基于卷积神经网络的昆虫和杂草标本智能全维识别方法初探. 植物检疫. 2023(02): 40-45 .  百度学术
百度学术
6. 王慧,李康顺,蔡铁,王文祥,董纯铿. 基于约束性多目标优化算法的柑橘黄龙病识别算法. 江苏农业科学. 2023(06): 159-167 .  百度学术
百度学术
7. 马军磊,栗伟周,张赞. 基于PP-YOLO的电子焊接实训装置安全报警系统. 许昌学院学报. 2023(02): 116-121 .  百度学术
百度学术
8. 李志臣,凌秀军,李鸿秋,李志军. 基于改进ShuffleNet的板栗分级方法. 山东农业大学学报(自然科学版). 2023(02): 299-307 .  百度学术
百度学术
9. 杨雪. 基于DCGAN的数据增强方法. 江苏通信. 2023(02): 97-100+107 .  百度学术
百度学术
10. 边坤,梁慧. 基于机器学习的图案分类研究进展. 图学学报. 2023(03): 415-426 .  百度学术
百度学术
11. 胡凯,骆润玫,刘泽乾,曹亚芃,廖飞,王卫星,李强,孙道宗. 基于改进SSD的广佛手病虫害检测方法. 南京农业大学学报. 2023(04): 813-821 .  百度学术
百度学术
12. 王治兵,吴丽丽. 基于改进的MobilenetV3苹果叶片病害图像识别技术. 软件工程. 2023(08): 20-25 .  百度学术
百度学术
13. 刘拥民,胡魁,聂佳伟,谢铁强. 基于MSDB-ResNet的水稻病虫害识别. 华南农业大学学报. 2023(06): 978-985 .  本站查看
本站查看
14. 赵法川,徐晓辉,宋涛,郝淼淼,汪曙,朱伟龙. 融合多头注意力的轻量级作物病虫害识别. 华南农业大学学报. 2023(06): 986-994 .  本站查看
本站查看
15. 李志臣,凌秀军,李鸿秋,李志军. 基于改进EfficientNet的板栗分级方法. 中国农机化学报. 2023(12): 180-185 .  百度学术
百度学术
16. 徐兢成,王丽华. 基于AlexNet网络的交通标志识别方法. 无线电工程. 2022(03): 470-475 .  百度学术
百度学术
17. 周巧黎,马丽,曹丽英,于合龙. 基于改进轻量级卷积神经网络MobileNetV3的番茄叶片病害识别. 智慧农业(中英文). 2022(01): 47-56 .  百度学术
百度学术
18. 胡哲,徐承志,雷光波,徐丽. 基于改进YOLOv5的X射线图像铸件缺陷实时检测. 激光杂志. 2022(05): 54-59 .  百度学术
百度学术
19. 叶名炀,张杰强. 基于轻量化网络MobileNetV2的玉米病害识别研究. 现代计算机. 2022(11): 46-50 .  百度学术
百度学术
20. 钟友闻,车文刚,高盛祥. 轻型多尺度黑色素瘤目标检测网络模型的建立:基于注意力机制调控. 南方医科大学学报. 2022(11): 1662-1671 .  百度学术
百度学术
21. 滕明洪,谭立新. 基于EfficientNetV2和迁移学习的葡萄病害识别研究. 软件. 2022(11): 43-49 .  百度学术
百度学术
22. 陆健强,梁效,余超然,兰玉彬,邱洪斌,黄捷伟,尹梓濠,陈慧洁,郑胜杰. 基于坐标注意力机制与高效边界框回归损失的线虫快速识别. 农业工程学报. 2022(22): 123-132 .  百度学术
百度学术
23. 卢慧林,陈大嵩,陈逢浩,叶静文,欧阳革成. 基于纳米孔测序技术的柑橘黄龙病检测方法建立. 环境昆虫学报. 2021(06): 1596-1600 .  百度学术
百度学术
其他类型引用(14)




 下载:
下载: