Evaluation of heat stress resistance and its molecular machanism of four indica rice cultivars at seedling stage
-
摘要:目的
明确4种不同籼稻品种苗期耐热性差异,为水稻品种选育与推广应用提供理论参考和技术支持。
方法采用水稻基因组重测序、表型鉴定结合转录水平分析综合评价华南地区高产常规水稻品种‘南秀美占’、杂交稻恢复系‘R5518’及香稻品种‘九里香’和‘南晶香占’的苗期耐热性。
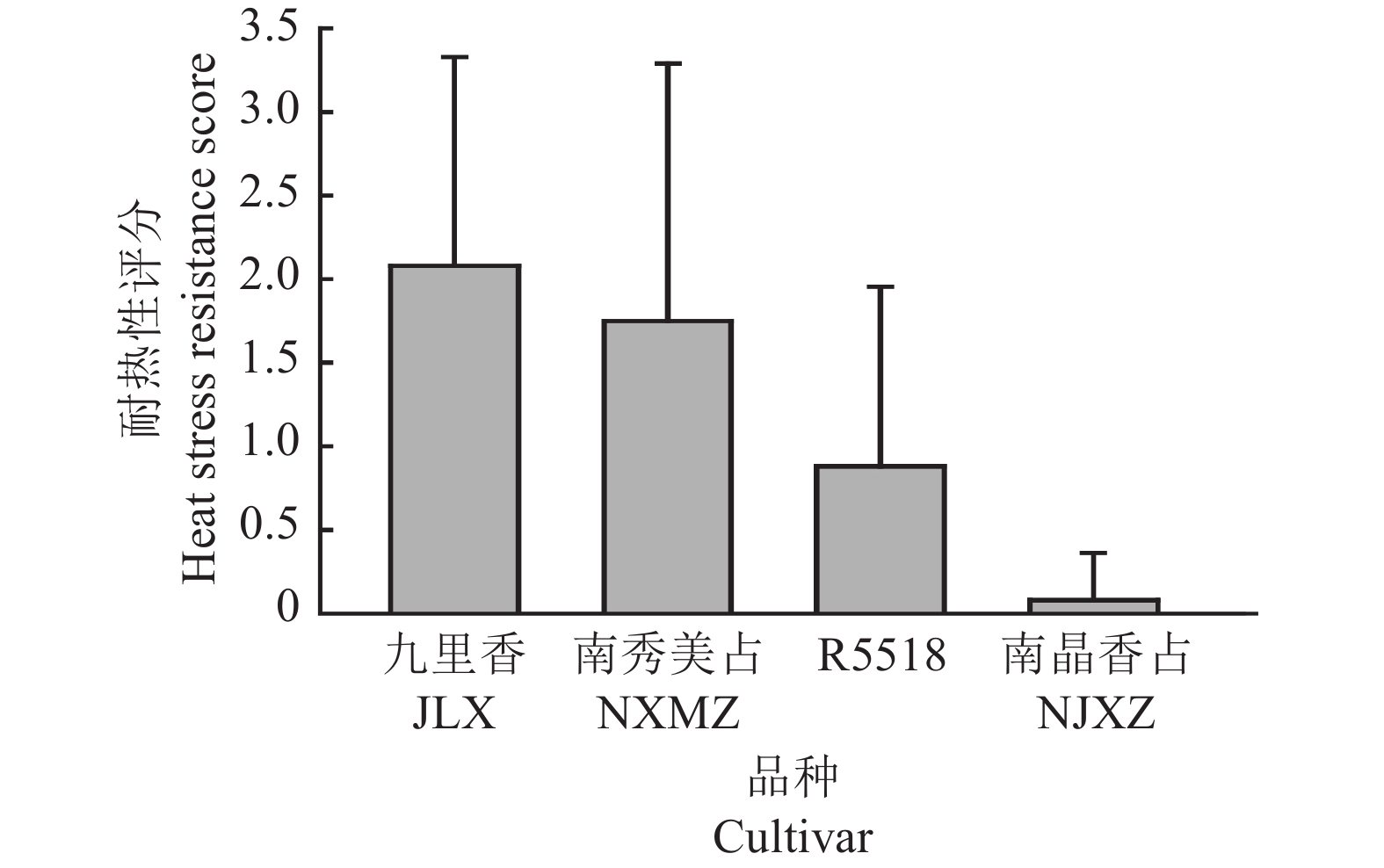
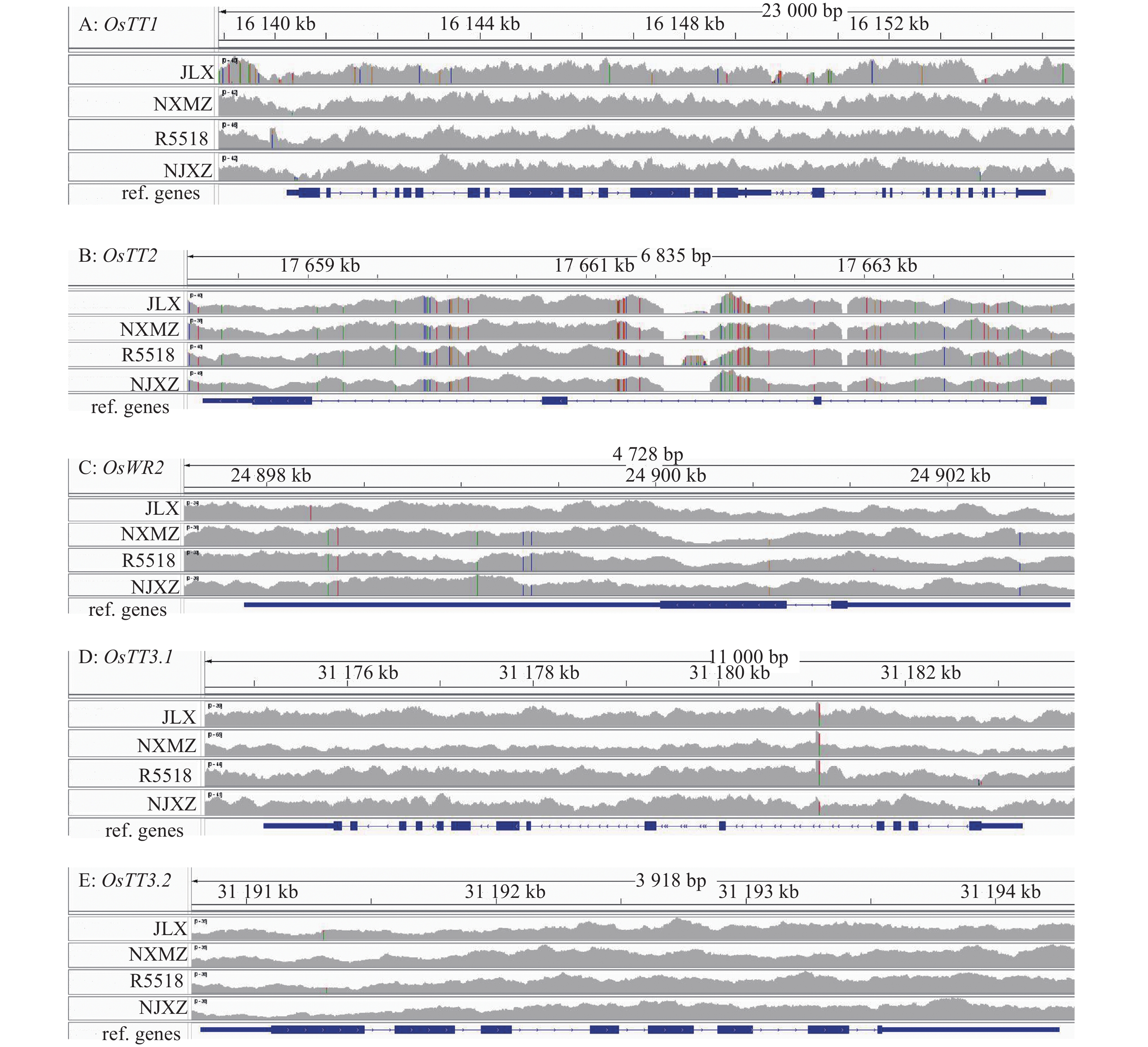
结果‘南晶香占’苗期耐热性相对较弱,‘R5518’耐热性中等,‘九里香’和‘南秀美占’表现出苗期耐热性强于另外2个品种;通过比较水稻抗热相关基因在这4个品种的单倍型、荧光定量数据与表型数据,发现‘九里香’在OsTT1表现出较多的变异,而其他水稻耐热QTLs上,4个品种单倍型较为一致,可能OsTT1贡献了‘九里香’部分耐热性;表达模式上,OsHSF7、OsHSP71.1和OsHTS1基因表达趋势与水稻耐热性评价比较一致,表明这3个基因可能参与该研究中的4个籼稻品种耐热性调控。
结论由于一些耐热相关基因的表达差异、基因移码、剪切转录本出错,造成基因失活,导致不同籼稻品种的苗期耐热性差异。该项研究结果可为水稻耐热性育种全基因组选择提供新的思路。
Abstract:ObjectiveTo identify the variation in heat stress resistance among seedlings of four different indica rice cultivars grown in South China, and provide theoretical reference and technical support for the breeding and promotion of rice varieties.
MethodWe performed whole-genome re-sequencing, phenotype identification, and transcriptional level analysis to comprehensively evaluate the heat stress resistance for seedlings of high-yield conventional rice cultivar ‘Nan Xiu Mei Zhan’ (NXMZ), hybrid rice restorer cultivar ‘R5518’, and aromatic rice cultivars ‘Jiu Li Xiang’ (JLX) and ‘Nan Jing Xiang Zhan’ (NJXZ) in South China.
ResultThe ‘NJXZ’ was sensitive to heat stress. The ‘R5518’ showed medium resistance to heat stress. The resistances to heat stress of ‘JLX’ and ‘NXMZ’at seedling stage were relatively high in comparison to other two rice cultivars. We compared the haplotypes of heat resistance related genes, the relative expression levels and phenotypes, and found that many SNPs appeared in OsTT1 from‘JLX’, while the haplotypes of four cultivars with other heat-related QTLs remained relatively consistent, suggesting that the OsTT1 might contribute to partial heat stress resistance in ‘JLX’. The gene expression patterns in OsHSF7, OsHSP71.1 and OsHTS1 were consistent with the evaluation in heat stress resistance of rice cultivars, indicating that these three genes might associate with regulation in heat stress resistance of four indica rice cultivars.
ConclusionThe gene expression difference, gene shift and transcript error in certain genes result in variations in heat stress resistance of different indica rice cultivars at seedling stage. These results can provide new ideas for genome-wide selection for heat tolerance breeding in rice.
-
气候变化已经对全球主要粮食作物的生产产生巨大影响,并且已经极大冲击农业生产及粮食安全[1]。作为世界范围内最主要的粮食作物之一,水稻Oryza sativa养活了世界近半数人口,我国有60%人口以水稻为主食[2]。夏季高温频发对我国乃至世界粮食安全生产造成极大威胁。据估算,全球平均气温每上升1 ℃,主要的粮食作物总产量平均减产约7.4%[3]。高温胁迫对水稻的影响主要在生殖生长时期造成危害,包括幼穗分化、抽穗、扬花和灌浆期等阶段[4]。高温热害能够阻碍配子体发育、水稻开花和受精过程,如水稻圆锥花序在减数分裂过程经历平均温度>33 ℃时,会致使雄蕊、雌蕊发育异常,小穗育性明显下降,结实率降低[5]。如果持续高温(>35 ℃)超过5 d,会造成花粉管伸长异常,花粉活性下降,正常散粉受阻,导致空瘪粒的产生[6]。除了抽穗期和扬花期,高温对籽粒灌浆期也会造成显著影响,如灌浆早期加快灌浆速度、灌浆时间变短、千粒质量下降及籽粒垩白率显著上升等,影响产量和稻米品质[7]。2022年夏秋,长江流域长达50余天的极端高温热害,导致我国长江中下游地区水稻生产受到严重影响。因此,研究水稻耐热性对水稻耐高温育种、提升水稻产量和品质具有重要生产应用意义。
近年来,关于水稻控制耐热性的QTLs已有许多报道。2015年,Li等[8]报道了水稻第1个抗热QTL,命名为OsTT1。OsTT1编码1个26S蛋白酶体α2小亚基,在籼稻和粳稻中由于清除高温引起的大量变性蛋白的效率有差异,造成籼稻更为耐热的性状。OsTT2编码G蛋白γ亚基,负调控水稻耐热性[9]。此外,G蛋白还可以通过热诱导的钙信号,调节2个钙调素结合的转录因子SCT1和SCT2对蜡质合成的重要转录因子OsWR2的激活。2022年,林鸿宣院士团队发现2个拮抗基因TT3.1和TT3.2调控叶绿体蛋白降解新机制,并且发现第1个潜在的作物高温感受器[10]。TT3.1是细胞质膜定位的E3泛素连接酶,响应高温信号转移到多囊泡体(Multivesicular body,MVB)将叶绿体前体蛋白TT3.2降解,减轻热胁迫下TT3.2积累造成的叶绿体损伤,从而提高水稻高温抗性。2022年万建民院士团队利用来源于普通野生稻O. rufipogon与亚洲栽培稻的近等基因系挖掘到1个位于5号染色体短臂端的耐热QTL,命名为qHTH5。该基因编码1个磷酸吡哆醛稳态蛋白,来源于普通野生稻的qHTH5能够通过增加热诱导的5'−磷酸吡哆醛(PLP)含量来减少高温下活性氧的积累进而增强水稻抗高温能力[11]。Wang等[12]通过togr1-1和‘中花11’杂交得到的F2群体将TOGR1精细定位到3号染色体28.5 kb区域。TOGR1编码1个位于核仁的DEAD盒RNA螺旋酶,该基因提供了水稻高温条件下正常生长的先决条件。通过将类受体激酶基因ERECTA过量表达到水稻和番茄,能够提高这2种作物的耐热性[13]。除了上述这些高温抗性QTLs外,还有一系列基因参与水稻高温胁迫调控过程[14]。通过克隆与鉴定这些水稻抗高温相关基因有利于构建高温的调控网络,并在水稻育种中开发新的分子标记及针对性培育新的抗高温水稻种质。
过去20年水稻基因组相关技术飞速发展,多个籼稻、粳稻、野生稻等品种的全基因组高精度组装序列均已完成,并针对全基因组关联分析(Genome wide association study,GWAS)的报道屡见不鲜[15-17]。利用水稻重测序技术对特定品种可以挖掘对应的基因型,结合目前多个水稻基因组数据库及表型鉴定,有助于我们对目标品种特定性状的调控基因预测和抗性进行综合评价,有针对性地为下一步进行品种改良导入新的位点,实现品种的“精准遗传改良”,提高育种效率。
由于生育期耐热植株的筛选复杂,条件苛刻,而苗期耐热性评价与筛选条件可控,操作方法与技术成熟。目前已克隆的3个水稻耐热QTLs均通过苗期耐热性克隆鉴定到,其在水稻苗期孕穗期和抽穗期均表现较强耐热性[8-10]。为加快选育适于长江流域推广应用的新品种,本研究对包括‘南晶香占’等4个籼稻品种进行苗期耐热性的表型鉴定、基因组差异比对及基因转录水平分析等系统评价,以期为提升水稻品种苗期、生殖期高温胁迫耐受性,培育耐热性强的水稻新品种,在育种方法与路径上提供应对策略。
1. 材料与方法
1.1 供试材料
以广东省农业科学院水稻研究所功能稻选育研究室刘传光研究员团队培育的香稻品种‘九里香’(JLX)、‘南晶香占’(NJXZ)及恢复系‘R5518’和高产优质常规稻‘南秀美占’(NXMZ)为试验材料[18]。重测序材料取自对应品种大丰基地种植的材料叶片。材料按照水稻常规方法田间管理及栽培。挑选健康饱满发芽率正常的种子进行苗期耐热性评价。
1.2 方法
1.2.1 重测序文库构建、比对及基因型分析
所用4个水稻品种委托北京诺禾致源公司进行文库构建及重测序。植物样品基因组DNA检测合格后,使用超声波破碎的方法对DNA进行随机打断,然后对片段化的DNA进行纯化、末端修复、添加接头,并进行PCR扩增形成测序文库,质检后用Illumina测序方法进行测序。高通量测序后通过Base calling算法得到原始测序序列,随后公司进行数据质控、与参考基因组比对,并对变异进行检测与注释等。最后通过横向比较4个水稻品种获得每个基因的启动子区(1.5 kb)、UTR区、编码区的所有基因变异信息。
1.2.2 水稻苗期耐热性鉴定及评价
水稻苗期耐热性鉴定参考前人提供的方法进行[8]。将健康饱满能正常发芽的种子浸入水中黑暗萌发,待露白后点播到露底的96孔板中,用水稻Yoshida液体培养基进行培养,定期换营养液,置于人工气候箱中进行苗期耐热性处理。正常培养环境为:28 ℃,13 h光照,湿度80%;25 ℃,11 h黑暗条件,湿度80%。自萌发算起2周后进行高温处理,环境温度调整为:42 ℃,13 h光照,湿度80%;42 ℃,11 h黑暗,湿度80%。处理时间为3 d。处理后将培养基置换,重新移回正常环境恢复培养,1周后进行存活率统计,拍照。每个处理进行3次生物学重复,选取其中1次进行表型展示。每个水稻品种每次处理植株为24株。基于存活率对每个恢复后的植株的耐热性进行打分,0分为死亡,5分为正常生长状态,通过计算得到每个品种的耐热性得分。分别进行3次不同的试验,得到的分数作为该品种的耐热性综合评价得分。
1.2.3 水稻品种基因组进化树分析
水稻品种基因组进化树利用iTOL进行制作(https://itol.embl.de/)[19]。
1.2.4 目标基因荧光定量PCR分析
对3 d高温处理完成的材料取样,分为3组作为生物学重复,用液氮冷冻后放入−80 ℃冰箱保存。采用RNA小量试剂盒(迈跟生物)提取幼苗RNA。RNA经过DNase I处理后采用Oligo d(T)反转录方法反转录成cDNA,设计目标基因的特异性qRT-PCR引物,内参基因使用Actin,引物信息如表1所示,利用ABI 7500荧光定量PCR仪运行,试剂使用Genestar,通过计算2−ΔΔCt值确定目标基因在4种不同遗传材料中的相对表达量[20]。
表 1 耐热相关基因及其对应的qRT-PCR引物信息Table 1. The corresponding qRT-PCR primer information of selected heat tolerance related genes基因
Gene基因号
Gene number正向序列(5′→3′)
Forward sequence反向序列(5′→3′)
Reverse sequence产物大小/bp
Product sizeTT1 LOC_Os03g26970 TGGAGCTTGACGATGCCATT CCTTGATCTCTGCAGGGCTC 141 TT2 Os03g0407400 CTCCAGATGCTGCAGAGAGG GCTCTGCACAAACAGCGAAA 132 TT3.1 LOC_Os03g49900 CTAGCTCATCATCAGCGGCA GTCAGGAAGACCACAGAGCC 104 OsWR2 LOC_Os06g40150 AATGGACGACGAGGAGAGGA GACGAGGCTACCTTCACCAC 112 续表 1 Continued table 1 基因
Gene基因号
Gene number正向序列(5′→3′)
Forward sequence反向序列(5′→3′)
Reverse sequence产物大小/bp
Product sizeTT3.2 LOC_Os03g49940 CACGATCCCCAAGCTGACTT AAGAACACCGCGGCTAAGAA 123 HTS1 LOC_Os04g30760 GACCCATGTTGCAGCTGTTG TGCACCCAGCTTTTCCAAGA 134 AET1 LOC_Os05g45890 GCCAACAGCGAGTACGAGTA AGAATCGGTGGAAGTGGCAG 109 TOGR1 LOC_Os03g46610 TAAGGTCGAGGTAGCGTCCA ATCATGCTCCCTGGCAACTC 126 OsER1 LOC_Os06g10230 CGACAATGCACGAGGTTGTG GCTGGTGCCTCTTAAGCTGA 143 OsMADS7 LOC_Os08g41950 GCAATTGAAAGCTAGCCGCA GCTGCTTCTCTAGGCTCTCG 137 OsFAD7 LOC_Os03g18070 GTTGAACAGCGTGGTTGGAC GACATGACCGTGGTTCTGGT 103 OsWRKY11 LOC_Os01g43650 GTTGATCACCTCGAGGACGG GCTTCTTCACACCGCACTTG 118 OsHCI1 LOC_Os10g30850 TCTCTCTCTTTTGCAGGGCG ATCCACTGCACGAGGAAGTG 106 OsHsfA2c LOC_Os10g28340 TGGAATCCCTGAGCTGGAGA AACAGCTCTGCCCAGAAGTC 129 OsHSBP1 LOC_Os09g20830 CCCGGCAGATATGACAGCAT TTCAGGTCGTTGACGCTCTG 144 OsHTAS LOC_Os09g15430 AACATCCGCATGCCCCTTTA CGGGTGGTTGTTCAGGTTCT 132 NAL11 LOC_Os07g09450 AAACGCCCATCCTGAGAAGG CCCCCTCCTTTTGTCTTCCC 141 HSA32 LOC_Os06g46900 CTCTACGGGCAGACATCGTC TCCACGAACAGATTCACCCG 133 OsHSBP2 LOC_Os06g16270 GCATCCCCATCAAGGCTGAT GGAACCTGGTTTGCATCTGC 105 HSP101 LOC_Os05g44340 GGTCGGCAAGAACTCCATGA ACCTTCCTGAGTTGCTCGTG 138 HTH5 LOC_Os05g05740 GCCTTGATCGTGTGGTAGCT CGAGATTCGGGCAGCCTAAT 150 OsHSP1 LOC_Os04g01740 CGTCAAGAAGCACTCCGAGT TCCTTGCCTTTGGAGTCGTC 117 OsHSP71.1 LOC_Os03g16860 CTGATCCCCAGGAACACCAC TCACCCTCGTACACCTGGAT 101 OsHSF7 LOC_Os03g06630 GATGGTGAAGGAGGAGTGGC CAGGTCGAACGTCTTGGTCA 115 2. 结果与分析
2.1 4个水稻品种的耐热性综合评价
通过3个不同批次的水稻苗期高温处理及耐热性评价,结果(图1)发现‘九里香’和‘南秀美占’2个品种表现出较为显著的苗期耐热抗性,恢复系‘R5518’则在水稻苗期耐热性评价表现中等,而‘南晶香占’的耐热性在4个水稻品种中表现较弱。对这4个水稻品种耐热性进行评分(图2),按照耐热性排序,4个水稻品种依次为‘九里香’>‘南秀美占’>‘R5518’>‘南晶香占’。
![]() 图 1 4个水稻品种苗期耐热性评价A~D:九里香(A)、南秀美占(B)、R5518(C)和南晶香占(D)种子萌发14 d正常培养表型图;E~H:九里香(E)、南秀美占(F)、R5518(G)和南晶香占(H)种子萌发14 d,高温处理3 d,正常条件下恢复生长7 d后的表型图;比例尺为2 cmFigure 1. Evaluation of heat stress resistance of four rice cultivars at seedling stageA−D: The phenotypes of 14 day seedlings after germination under normal condition of JLX (A), NXMZ (B), R5518 (C), and NJXZ (D); E−H: The phenotypes of JLX (E), NXMZ (F), R5518 (G), and NJXZ (H) seedlings after 14 days post germination, three days of heat treatment and seven days of recovery; Scale bar = 2 cm
图 1 4个水稻品种苗期耐热性评价A~D:九里香(A)、南秀美占(B)、R5518(C)和南晶香占(D)种子萌发14 d正常培养表型图;E~H:九里香(E)、南秀美占(F)、R5518(G)和南晶香占(H)种子萌发14 d,高温处理3 d,正常条件下恢复生长7 d后的表型图;比例尺为2 cmFigure 1. Evaluation of heat stress resistance of four rice cultivars at seedling stageA−D: The phenotypes of 14 day seedlings after germination under normal condition of JLX (A), NXMZ (B), R5518 (C), and NJXZ (D); E−H: The phenotypes of JLX (E), NXMZ (F), R5518 (G), and NJXZ (H) seedlings after 14 days post germination, three days of heat treatment and seven days of recovery; Scale bar = 2 cm2.2 4个水稻品种重测序建库及数据分析
为了进一步探究不同水稻品种在基因组水平的差异与相似性,我们对4个水稻品种进行重测序分析。本次测序共产生原始数据43.69 GB,过滤后的干净数据量为43.34 GB。重测序数据的GC含量在41%~42%之间,Q20达到95%~96%,Q30达到89%~90%,错误率在0.03%以下,数据有效率均在99.2%左右。因此,重测序数据质量较高,为接下来进一步数据分析打下良好基础。我们选用‘日本晴’的参考基因组进行测序比对。测序设计覆盖度统计汇总如表2。4个品种的测序平均深度都能达到约22×~24×。1×覆盖度均在94%以上,表明该重测序效果较佳,能够反映绝大部分这4个水稻品种的基因组性质。此外,进一步对这4个水稻品种进行变异情况统计,结果见表3。在基因组水平上,变异包括单核苷酸多态性(Single nucleotide polymorphism,SNP)、小片段的插入和缺失(Insertion-deletion,InDel)、结构变异(Structure variation,SV)及拷贝数变异(Copy number variation,CNV)等。由于这4个水稻品种均是近年来育成的华南地区的水稻品种,其内部差异及各种基因组变异情况相对较小。通过第2代重测序检测到的SNP位点在800 685~920 843之间,InDel个数在139 065~159 588之间,SV个数在12 201~13 125之间,CNV个数在6 375~7 208之间,变异检测结果符合预期。由于这4个品种是与‘珍汕97’数据库进行比对分析,通过进化树分析,发现虽然‘南晶香占’和‘九里香’在耐热性方面有较大的差异,但是基因组水平上2个品种亲缘关系较近。而这4个水稻品种中,‘R5518’与其他品种亲缘关系较远(图3)。综上,通过对4个水稻品种的重测序获得了一系列关于这4个水稻的基因组差异信息。
表 2 4个水稻品种测序深度及覆盖度统计1)Table 2. Sequencing depth and coverage of four rice cultivars品种
Cultivar比对读数
Comparative
reads总读数
Total
reads比对率/%
Comparison
rate平均测序深度
Average sequencing
depth1×覆盖度/%
1× coverage4×覆盖度/%
4× coverage5×覆盖度/%
5× coverage九里香 JLX 68 508 565 69 806 334 98.14 22.62 94.48 91.58 92.58 南秀美占 NXMZ 71 978 559 73 338 268 98.15 24.39 94.87 92.17 93.17 R5518 68 952 835 73 150 652 94.26 23.30 94.75 92.10 93.10 南晶香占 NJXZ 71 235 025 72 695 318 97.99 24.32 94.81 92.40 93.40 1) 比对读数是测序读数比对到参考基因组的总读数(包括单端比对和双端比对);总读数指有效测序数据的总reads数;比对率指比对到参考基因组的读数比例;平均测序深度指比对到参考基因组的碱基总数除以覆盖的基因组大小;1×、4×和5×覆盖度分别指参考基因组至少有1个、连续4个和5个碱基覆盖的位点占基因组的百分比
1) The comparative reads number is the total number of sequencing reads which paired to the reference genome (including single-end paired and double-end paired); The total reads refers to the total number of valid reads paired to the reference genome; The average sequencing depth refers to the total number of reads paired to the reference genome divided by the size of the genome; 1×, 4× and 5× coverages refer to the percentages of the genome covered by at least 1 base, serial 4 bases and 5 bases respectively in the reference genome表 3 4个水稻品种基因组不同种类变异的数量统计1)Table 3. Counting of different types of genomic variations in four rice cultivars品种 Cultivar SNP InDel SV CNV 九里香 JLX 857923 148394 12036 6802 南秀美占 NXMZ 800685 139065 12201 6375 R5518 920843 159588 13125 7050 南晶香占 NJXZ 859302 148878 12401 7208 1) SNP指由单个核苷酸的变异所引起的DNA序列多态性;InDel指小于50 bp的小片段的插入和缺失;SV指50 bp以上的大片段的插入、缺失、倒置、易位;CNV指基因组片段的拷贝数增加或者减少
1)SNP refers to the DNA polymorphism caused by variation in a single nucleotide; InDel refers to the insertions and deletions of small fragments (< 50 bp); SV refers to large fragments (≥ 50 bp) of insertions, deletions, inversions, and translocations; CNV refers to the increase or decrease of the copy gene numbers in genome2.3 4个水稻品种在多个耐热QTLs发生变异
根据文献检索等分析并参考目前已经报道的与水稻耐热性相关的QTLs和明显作用的单基因[14],基于4个水稻品种的特定基因信息,利用Integrative genomics viewer(IGV)可视化工具分析4个水稻品种基因组在这些基因上存在的差异[21]。分析发现,相较其他3个水稻品种,‘九里香’在OsTT1表现出较多的变异,而‘R5518’在5'UTR也有1处特殊SNP变异(图4A)。而4个水稻品种在OsTT2基因上发生了一致的大量的SNP变异,特别是在内含子区的变异非常集中(图4B)。而OsTT2调控的靶基因OsWR2的基因单倍型在‘南晶美占’‘R5518’及‘南晶香占’中更接近,‘九里香’则与参考基因组序列更接近(图4C)。对于OsTT3调控模块,相较参考基因组,则发现这4个水稻品种在内含子区有1个共同的变异,而‘R5518’在5'UTR存在1个特殊的变异(图4D)。OsTT3.2基因除了‘九里香’和‘R5518’存在1个同义突变外,其他序列在这4个水稻品种中与参考基因组完全一致(图4E)。综上所述,对这4个水稻品种的3个已发现的耐热QTL基因变异情况分析发现,OsTT1基因在‘九里香’存在更多基因变异。
2.4 qRT-PCR验证耐热相关基因的表达量与水稻基因型、处理表型的关联性
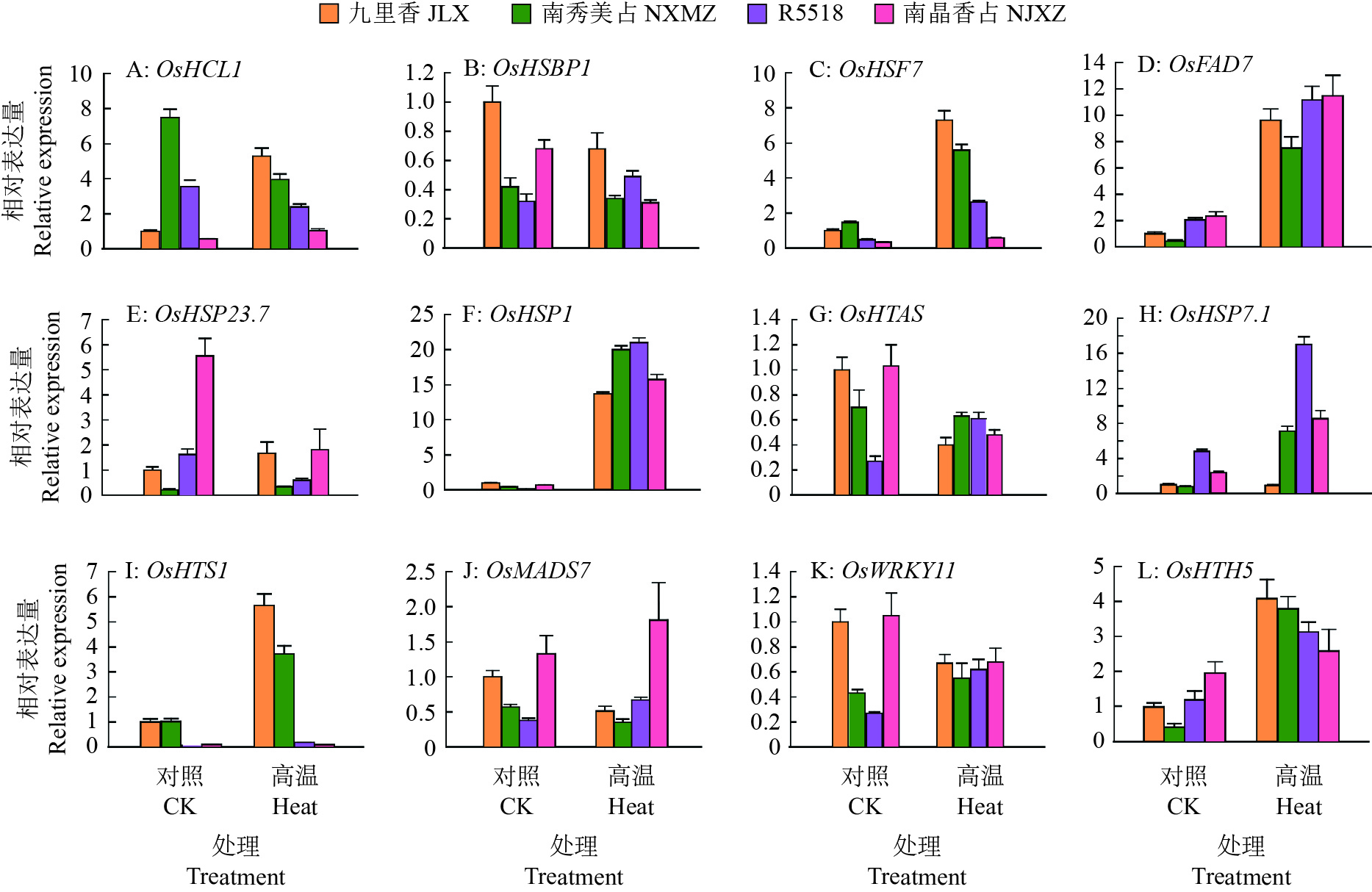
对这4个品种水稻处理前后材料进行qRT-PCR验证。结果发现,一些与耐热性相关的基因在不同的水稻品种热处理前后诱导上调的比例相差较大。其中一些基因表现出热处理前后并没有显著诱导的关系,如OsHCL1、OsHSBP1、OsHSP23.7、OsHTAS和OsWRKY11等(图5A、5B、5E、5G和5K)。而另外一些基因,如OsHSF7、OsFAD7、OsHSP1、OsHSP71.1、OsHTS1、OsMADS7和OsHTH5等,则有不同程度的受到温度诱导上调(图5C、5D、5F、5H、5I、5J和5L)。大部分这些热相关基因中,4个水稻品种的处理前及处理后的差异都比较明显。其中,OsHSF7和OsHTS1的表达模式非常接近,在‘九里香’和‘南晶美占’2个抗高温的品种中,处理前后该基因的相对表达量均比耐热性较差的‘R5518’和‘南晶香占’更高(图5C和5I)。与这2个基因表达模式相反的OsHSP71.1,该基因在‘九里香’和‘南晶美占’2个抗高温的品种表达量更低,而在热敏感的‘R5518’和‘南晶香占’材料中基因相对表达量更高(图5H)。还有一类基因在4个水稻品种没有热处理情况下本底表达都比较低,而热处理后基因相对表达量出现非常显著的上调,如OsFAD7和OsHSP1(图5D和5F),这些结果与前人的研究一致[22-23]。综上所述,通过对处理前后这些热相关基因的相对表达量变化的初步探索,认为OsHSF7、OsHSP71.1和OsHTS1可能是贡献了这4个水稻品种耐热性差异的部分原因。
![]() 图 5 4个水稻品种高温处理前后12个耐热相关基因的qRT-PCR转录水平差异图中数据为3次生物学重复的平均值±标准差;Actin作为内参进行均一化计算基因表达量;‘九里香’处理前样品标准化为1Figure 5. Differences in qRT-PCR transcription levels of twelve heat tolerance related genes among four rice cultivars before and after heat treatmentThe data in the figure represents the mean ± standard deviation of three biological replicates; Actin was used for internal control to normalize the gene expression data; The sample of ‘Jiulixiang’ before treatment was standardized to 1
图 5 4个水稻品种高温处理前后12个耐热相关基因的qRT-PCR转录水平差异图中数据为3次生物学重复的平均值±标准差;Actin作为内参进行均一化计算基因表达量;‘九里香’处理前样品标准化为1Figure 5. Differences in qRT-PCR transcription levels of twelve heat tolerance related genes among four rice cultivars before and after heat treatmentThe data in the figure represents the mean ± standard deviation of three biological replicates; Actin was used for internal control to normalize the gene expression data; The sample of ‘Jiulixiang’ before treatment was standardized to 13. 讨论与结论
本研究选用广东丝苗米品种‘南晶香占’、优质香稻品种‘九里香’、高产优质稻品种‘南秀美占’和三系杂交稻恢复系‘R5518’等4个品种作试验材料,在华南地区水稻品种中有一定代表性。对苗期耐热性处理评价发现,‘南晶香占’的苗期耐热性较弱,因此,‘南晶香占’在有高温危害地区种植时应采取防高温措施,如田间灌深水等。若遇到高温与干旱同时发生的季节,更要针对性做好田间管理,防范大面积空瘪粒发生、产量下降等。值得注意的是,我们的处理结果与水稻品种的亲缘关系呈现明显关联过程。由于‘九里香’和‘南晶香占’2个水稻品种都具有共同的亲本来源‘象牙香占’,两者在基因组水平上的亲缘关系非常接近,然而这2个品种的耐热性评价则表型较大差异,说明有可能‘九里香’具有的高耐热性是来源于另一个供给亲本。高产优质常规稻‘南秀美占’具有较好的耐热性,表明该品种向广东省外如江西省、湖南省或者长江中下游区域推广可能具有较大的潜力。
本研究发现,4个参试品种耐热性存在显著差异,可能OsTT1调控模块在‘九里香’耐热性差异上发挥重要作用,而其他的一些质量性状基因,包括OsHSF7、OsHSP71.1和OsHTS1等也可能贡献了这些水稻品种部分耐热性差异[8, 22-24]。因此,应加大对已知的及未知的耐热基因的挖掘,加快建立水稻高温环境下的调控网络,通过基因组水平重测序技术、基因编辑技术等新兴育种技术在某些高产优质基础材料上快速聚合、产生具有多种生物与非生物胁迫的突破产量品质天花板和广适应性的新型优质高产新种质,为保障我国粮食安全做出重要贡献。
本研究通过对4个水稻品种耐热性评价与重测序数据结合,并且应用qRT-PCR验证差异基因的表达水平差异,初步揭示这4个亲缘关系较近的水稻品种耐热性响应、调控能力的差异原因。基于本研究,我们明确了未来对于这4个水稻品种在田间管理技术上的不同侧重方向。目前的水稻品种审定标准中主要关注到审定品种的产量与品质,生物胁迫中稻瘟病抗性具有‘一票否决’制。此外,其他的生物胁迫与非生物胁迫方面不作强制要求。其次,本研究对这4个水稻品种的耐热性评价是对该品种的新的探索,但是本项研究也具有一定局限性:比如选取的品种代表性和数量不足;重测序只能覆盖到基因组约94%的1×覆盖度,仍然有许多区域并未覆盖到;本项研究用的是注释程度比较完善的‘日本晴’基因组,并未采用更多亲缘关系更近的籼稻品种,部分序列比对如高重复序列、大片段插入、缺失等可能比对性较差;本研究仅建立稳定筛选苗期耐热性评价体系及筛选平台,关于水稻生殖期耐热性评价体系和平台建立仍需加大更多投入。因此,在未来的工作中应拓展探索这4个水稻品种在响应其他的生物胁迫与非生物胁迫中的差异,更好地为种植业者提供这些主栽品种的栽培特点。如果有更多水稻品种重测序数据,我们可以依照本项目研究思路与方法建立水稻多品种的基因组关联分析,甚至挖掘出主栽品种存在的主要调控网络或者新的QTLs。
-
图 1 4个水稻品种苗期耐热性评价
A~D:九里香(A)、南秀美占(B)、R5518(C)和南晶香占(D)种子萌发14 d正常培养表型图;E~H:九里香(E)、南秀美占(F)、R5518(G)和南晶香占(H)种子萌发14 d,高温处理3 d,正常条件下恢复生长7 d后的表型图;比例尺为2 cm
Figure 1. Evaluation of heat stress resistance of four rice cultivars at seedling stage
A−D: The phenotypes of 14 day seedlings after germination under normal condition of JLX (A), NXMZ (B), R5518 (C), and NJXZ (D); E−H: The phenotypes of JLX (E), NXMZ (F), R5518 (G), and NJXZ (H) seedlings after 14 days post germination, three days of heat treatment and seven days of recovery; Scale bar = 2 cm
图 5 4个水稻品种高温处理前后12个耐热相关基因的qRT-PCR转录水平差异
图中数据为3次生物学重复的平均值±标准差;Actin作为内参进行均一化计算基因表达量;‘九里香’处理前样品标准化为1
Figure 5. Differences in qRT-PCR transcription levels of twelve heat tolerance related genes among four rice cultivars before and after heat treatment
The data in the figure represents the mean ± standard deviation of three biological replicates; Actin was used for internal control to normalize the gene expression data; The sample of ‘Jiulixiang’ before treatment was standardized to 1
表 1 耐热相关基因及其对应的qRT-PCR引物信息
Table 1 The corresponding qRT-PCR primer information of selected heat tolerance related genes
基因
Gene基因号
Gene number正向序列(5′→3′)
Forward sequence反向序列(5′→3′)
Reverse sequence产物大小/bp
Product sizeTT1 LOC_Os03g26970 TGGAGCTTGACGATGCCATT CCTTGATCTCTGCAGGGCTC 141 TT2 Os03g0407400 CTCCAGATGCTGCAGAGAGG GCTCTGCACAAACAGCGAAA 132 TT3.1 LOC_Os03g49900 CTAGCTCATCATCAGCGGCA GTCAGGAAGACCACAGAGCC 104 OsWR2 LOC_Os06g40150 AATGGACGACGAGGAGAGGA GACGAGGCTACCTTCACCAC 112 续表 1 Continued table 1 基因
Gene基因号
Gene number正向序列(5′→3′)
Forward sequence反向序列(5′→3′)
Reverse sequence产物大小/bp
Product sizeTT3.2 LOC_Os03g49940 CACGATCCCCAAGCTGACTT AAGAACACCGCGGCTAAGAA 123 HTS1 LOC_Os04g30760 GACCCATGTTGCAGCTGTTG TGCACCCAGCTTTTCCAAGA 134 AET1 LOC_Os05g45890 GCCAACAGCGAGTACGAGTA AGAATCGGTGGAAGTGGCAG 109 TOGR1 LOC_Os03g46610 TAAGGTCGAGGTAGCGTCCA ATCATGCTCCCTGGCAACTC 126 OsER1 LOC_Os06g10230 CGACAATGCACGAGGTTGTG GCTGGTGCCTCTTAAGCTGA 143 OsMADS7 LOC_Os08g41950 GCAATTGAAAGCTAGCCGCA GCTGCTTCTCTAGGCTCTCG 137 OsFAD7 LOC_Os03g18070 GTTGAACAGCGTGGTTGGAC GACATGACCGTGGTTCTGGT 103 OsWRKY11 LOC_Os01g43650 GTTGATCACCTCGAGGACGG GCTTCTTCACACCGCACTTG 118 OsHCI1 LOC_Os10g30850 TCTCTCTCTTTTGCAGGGCG ATCCACTGCACGAGGAAGTG 106 OsHsfA2c LOC_Os10g28340 TGGAATCCCTGAGCTGGAGA AACAGCTCTGCCCAGAAGTC 129 OsHSBP1 LOC_Os09g20830 CCCGGCAGATATGACAGCAT TTCAGGTCGTTGACGCTCTG 144 OsHTAS LOC_Os09g15430 AACATCCGCATGCCCCTTTA CGGGTGGTTGTTCAGGTTCT 132 NAL11 LOC_Os07g09450 AAACGCCCATCCTGAGAAGG CCCCCTCCTTTTGTCTTCCC 141 HSA32 LOC_Os06g46900 CTCTACGGGCAGACATCGTC TCCACGAACAGATTCACCCG 133 OsHSBP2 LOC_Os06g16270 GCATCCCCATCAAGGCTGAT GGAACCTGGTTTGCATCTGC 105 HSP101 LOC_Os05g44340 GGTCGGCAAGAACTCCATGA ACCTTCCTGAGTTGCTCGTG 138 HTH5 LOC_Os05g05740 GCCTTGATCGTGTGGTAGCT CGAGATTCGGGCAGCCTAAT 150 OsHSP1 LOC_Os04g01740 CGTCAAGAAGCACTCCGAGT TCCTTGCCTTTGGAGTCGTC 117 OsHSP71.1 LOC_Os03g16860 CTGATCCCCAGGAACACCAC TCACCCTCGTACACCTGGAT 101 OsHSF7 LOC_Os03g06630 GATGGTGAAGGAGGAGTGGC CAGGTCGAACGTCTTGGTCA 115 表 2 4个水稻品种测序深度及覆盖度统计1)
Table 2 Sequencing depth and coverage of four rice cultivars
品种
Cultivar比对读数
Comparative
reads总读数
Total
reads比对率/%
Comparison
rate平均测序深度
Average sequencing
depth1×覆盖度/%
1× coverage4×覆盖度/%
4× coverage5×覆盖度/%
5× coverage九里香 JLX 68 508 565 69 806 334 98.14 22.62 94.48 91.58 92.58 南秀美占 NXMZ 71 978 559 73 338 268 98.15 24.39 94.87 92.17 93.17 R5518 68 952 835 73 150 652 94.26 23.30 94.75 92.10 93.10 南晶香占 NJXZ 71 235 025 72 695 318 97.99 24.32 94.81 92.40 93.40 1) 比对读数是测序读数比对到参考基因组的总读数(包括单端比对和双端比对);总读数指有效测序数据的总reads数;比对率指比对到参考基因组的读数比例;平均测序深度指比对到参考基因组的碱基总数除以覆盖的基因组大小;1×、4×和5×覆盖度分别指参考基因组至少有1个、连续4个和5个碱基覆盖的位点占基因组的百分比
1) The comparative reads number is the total number of sequencing reads which paired to the reference genome (including single-end paired and double-end paired); The total reads refers to the total number of valid reads paired to the reference genome; The average sequencing depth refers to the total number of reads paired to the reference genome divided by the size of the genome; 1×, 4× and 5× coverages refer to the percentages of the genome covered by at least 1 base, serial 4 bases and 5 bases respectively in the reference genome表 3 4个水稻品种基因组不同种类变异的数量统计1)
Table 3 Counting of different types of genomic variations in four rice cultivars
品种 Cultivar SNP InDel SV CNV 九里香 JLX 857923 148394 12036 6802 南秀美占 NXMZ 800685 139065 12201 6375 R5518 920843 159588 13125 7050 南晶香占 NJXZ 859302 148878 12401 7208 1) SNP指由单个核苷酸的变异所引起的DNA序列多态性;InDel指小于50 bp的小片段的插入和缺失;SV指50 bp以上的大片段的插入、缺失、倒置、易位;CNV指基因组片段的拷贝数增加或者减少
1)SNP refers to the DNA polymorphism caused by variation in a single nucleotide; InDel refers to the insertions and deletions of small fragments (< 50 bp); SV refers to large fragments (≥ 50 bp) of insertions, deletions, inversions, and translocations; CNV refers to the increase or decrease of the copy gene numbers in genome -
[1] ZHAO C, LIU B, PIAO S, et al. Temperature increase reduces global yields of major crops in four independent estimates[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(35): 9326-9331.
[2] 陈祖龙, 郑珍珍, 王慧, 等. 水稻耐热育种研究及其展望[J]. 滁州学院学报, 2023, 25(2): 23-28. [3] LIU H, ZENG B, ZHAO J, et al. Genetic research progress: Heat tolerance in rice[J]. International Journal of Molecular Sciences, 2023, 24(8): 7140.
[4] NUBANKOH P, WANCHANA S, SAENSUK C, et al. QTL-seq reveals genomic regions associated with spikelet fertility in response to a high temperature in rice (Oryza sativa L.)[J]. Plant Cell Reports, 2020, 39(1): 149-162. doi: 10.1007/s00299-019-02477-z
[5] YE C, TENORIO F A, ARGAYOSO M A, et al. Identifying and confirming quantitative trait loci associated with heat tolerance at flowering stage in different rice populations[J]. BMC Genetics, 2015, 16: 41.
[6] ZHU S, HUANG R, WAI H P, et al. Mapping quantitative trait loci for heat tolerance at the booting stage using chromosomal segment substitution lines in rice[J]. Physiology and Molecular Biology of Plants, 2017, 23(4): 817-825. doi: 10.1007/s12298-017-0465-4
[7] 王丰, 程方民, 刘奕, 等. 不同温度下灌浆期水稻籽粒内源激素含量的动态变化[J]. 作物学报, 2006, 32(1): 25-29. [8] LI X M, CHAO D Y, WU Y, et al. Natural alleles of a proteasome alpha2 subunit gene contribute to thermotolerance and adaptation of African rice[J]. Nature Genetics, 2015, 47(7): 827-833. doi: 10.1038/ng.3305
[9] KAN Y, MU X R, ZHANG H, et al. TT2 controls rice thermotolerance through SCT1-dependent alteration of wax biosynthesis[J]. Nature Plants, 2022, 8(1): 53-67.
[10] ZHANG H, ZHOU J F, KAN Y, et al. A genetic module at one locus in rice protects chloroplasts to enhance thermotolerance[J]. Science, 2022, 376(6599): 1293-1300. doi: 10.1126/science.abo5721
[11] CAO Z B, TANG H W, CAI Y H, et al. Natural variation of HTH5 from wild rice, Oryza rufipogon Griff., is involved in conferring high-temperature tolerance at the heading stage[J]. Plant Biotechnology Journal, 2022, 20(8): 1591-1605.
[12] WANG D, QIN B X, LI X, et al. Nucleolar DEAD-Box RNA helicase TOGR1 regulates thermotolerant growth as a Pre-rRNA chaperone in rice[J]. PLoS Genetics, 2016, 12(2): e1005844. doi: 10.1371/journal.pgen.1005844
[13] SHEN H, ZHONG X B, ZHAO F F, et al. Overexpression of receptor-like kinase ERECTA improves thermotolerance in rice and tomato[J]. Nature Biotechnology, 2015, 33(9): 996-1003.
[14] YANG Y, YU J, QIAN Q, et al. Enhancement of heat and drought stress tolerance in rice by genetic manipulation: A systematic review[J]. Rice, 2022, 15(1): 67. doi: 10.1186/s12284-022-00614-z
[15] YU H, LIU Y, SUN B, et al. Genetic diversity and breeding signatures for regional Indica rice improvement in Guangdong of Southern China[J]. Rice, 2023, 16(1): 25. doi: 10.1186/s12284-023-00642-3
[16] WANG J, YANG W, ZHANG S, et al. A pangenome analysis pipeline provides insights into functional gene identification in rice[J]. Genome Biology, 2023, 24(1): 19.
[17] WEI X, QIU J, YONG K, et al. A quantitative genomics map of rice provides genetic insights and guides breeding[J]. Nature Genetics, 2021, 53(2): 243-253. doi: 10.1038/s41588-020-00769-9
[18] 刘传光, 周新桥, 陈达刚, 等. 广东丝苗米品种南晶香占的选育与栽培技术[J]. 农业与技术, 2022, 42(7): 94-97. [19] LETUNIC I, BORK P. Interactive tree of Life (iTOL) v5: An online tool for phylogenetic tree display and annotation[J]. Nucleic Acids Research, 2021, 49(W1): W293-W296. doi: 10.1093/nar/gkab301
[20] CHEN K, YE C, GUO J, et al. Agrobacterium-mediated transformation efficiency and grain phenotypes in six indica and japonica rice cultivars[J]. Seed Biology, 2023, 2: 4. doi: 10.48130/SeedBio-2023-0004.
[21] ROBINSON J T, THORVALDSDOTTIR H, TURNER D, et al. Igv. js: An embeddable JavaScript implementation of the Integrative Genomics Viewer (IGV)[J]. Bioinformatics, 2023, 39(1): btac830.
[22] GOPALAKRISHNAN N, KANG I, MOON B, et al. Effects of low temperature stress on rice (Oryza sativa L. ) plastid ω-3 desaturase gene, OsFAD8 and its functional analysis using T-DNA mutants[J]. Plant Cell, Tissue and Organ Culture, 2009, 98(1): 87-96. doi: 10.1007/s11240-009-9541-y
[23] MOON J, HAM D, HWANG S, et al. Molecular characterization of a heat inducible rice gene, OsHSP1, and implications for rice thermotolerance[J]. Genes & Genomics, 2014, 36(2): 151-161.
[24] LIU J G, QIN Q L, ZHANG Z, et al. OsHSF7 gene in rice, Oryza sativa L., encodes a transcription factor that functions as a high temperature receptive and responsive factor[J]. BMB Reports, 2009, 42(1): 16-21. doi: 10.5483/BMBRep.2009.42.1.016
-
期刊类型引用(1)
1. 庞丹丹,刘玉飞,蒋会兵,田易萍,陈春林,许燕,邓少春,陈林波. 茶树完全不育花内源激素含量分析. 南方农业学报. 2025(04): 1235-1244 .  百度学术
百度学术
其他类型引用(1)



 下载:
下载: