Rice panicle recognition in field based on improved YOLOv5l model
-
摘要:目的
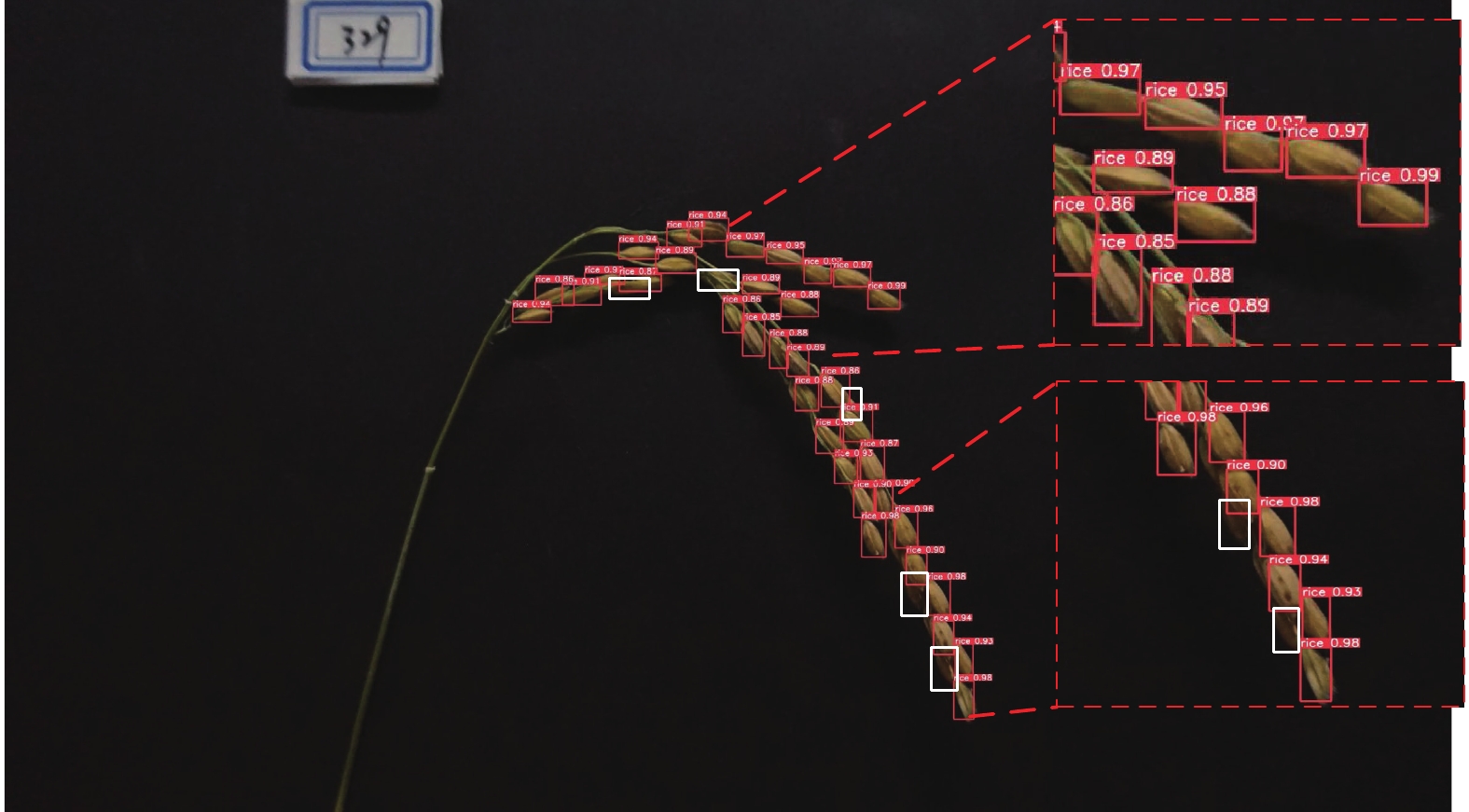
引入YOLOv5l算法模型并对其进行改进,以实现大田环境下水稻稻穗的精准、高效、无损检测。
方法以田间水稻为研究对象,通过数码单镜反光相机采集水稻图像样本,人工标注后对原始图像进行离线数据增强扩充,构建田间水稻图像数据集;对YOLOv5l算法进行适应性改进,在空间金字塔池化(Spatial pyramid pooling,SPP)层前以及Cross-stage-sartial-connections (CSP) 层中置入有效通道注意力(Efficient channel attention,ECA)机制,并进行对比试验。选取最优算法作为基准模型进行注意力机制和数据增强消融试验,并测试得到性能最优模型。将改进YOLOv5l与YOLOv5l、YOLOv5x、SSD和Faster R-CNN进行对比试验。
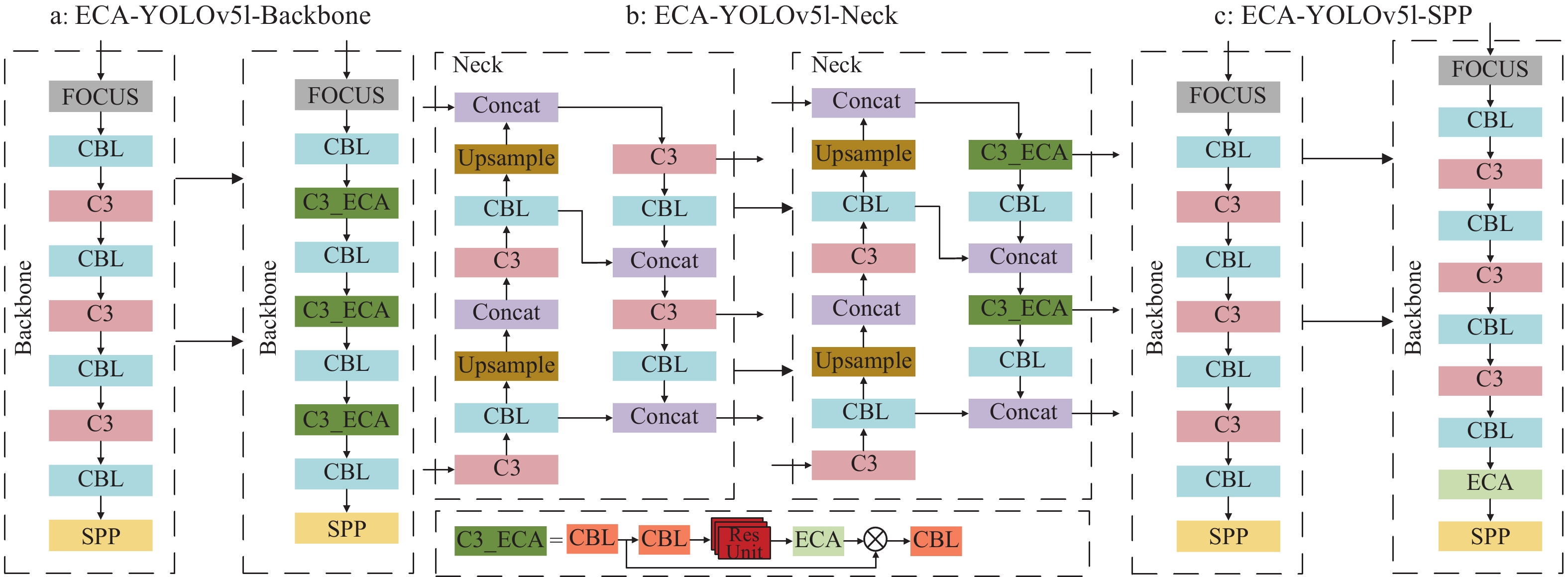
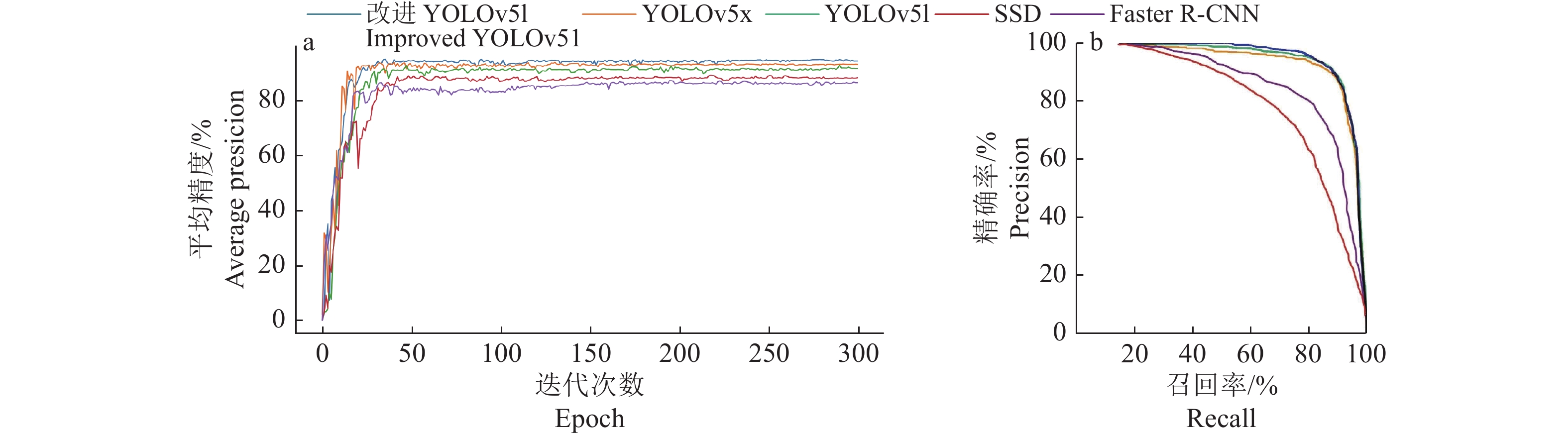
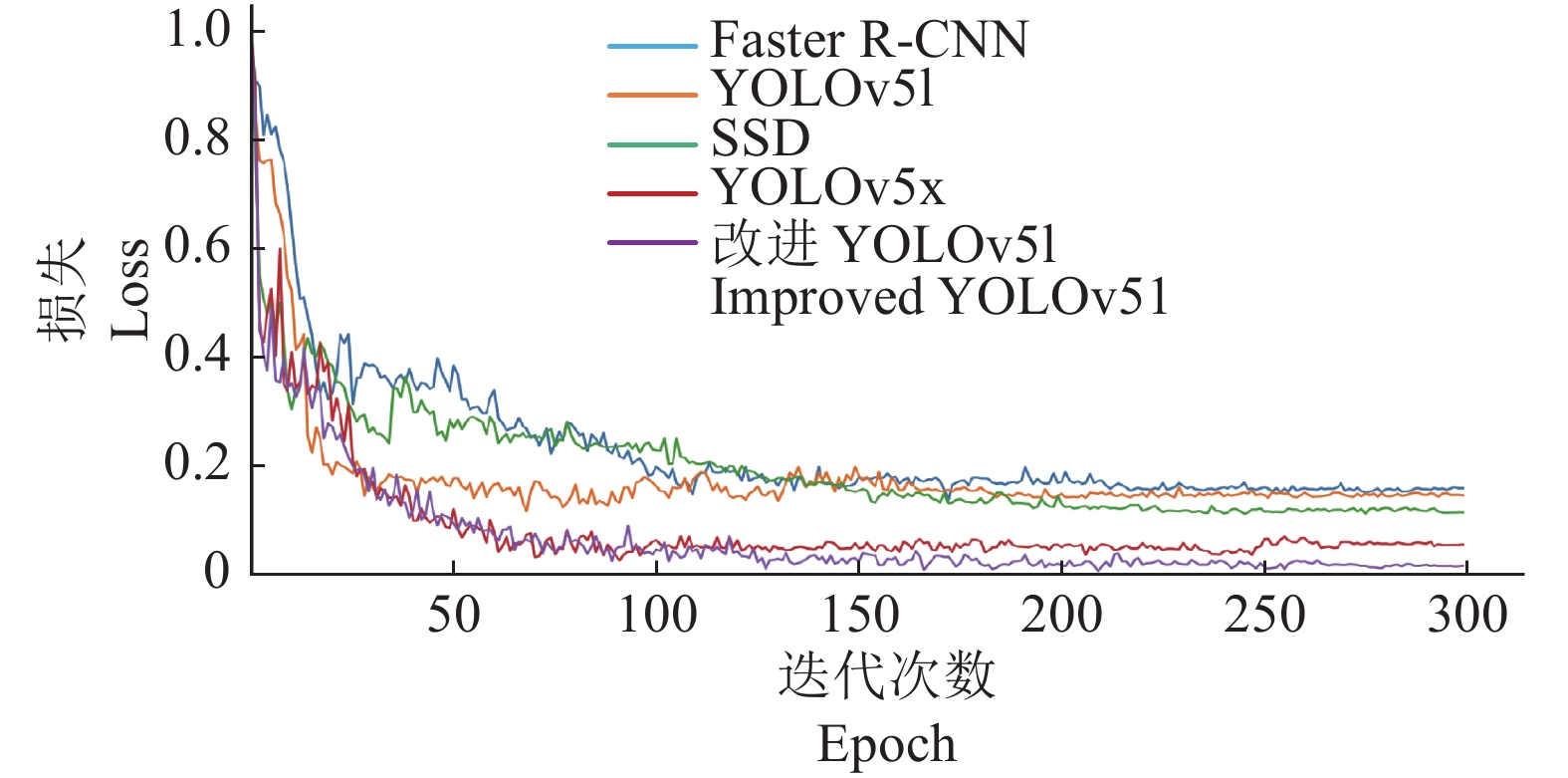
结果在改进YOLOv5l的水稻识别框架中,将ECA置入网络SPP层前有更出色的性能。利用测试集图像检验模型,识别结果的平均精确率为93.63%,平均召回率为90.94%,总体平均精度可达95.05%。与未融合YOLOv5l算法相比,改进的YOLOv5l算法平均精度高3.03个百分点,图像的检测速率快8.20帧/ms;与YOLOv5x算法相比,改进的YOLOv5l算法平均精度提高0.62个百分点,图像的检测速率快5.41帧/ms,内存占用减少74.1MB,在田间水稻稻穗检测方面,改进YOLOv5l算法的综合性能优于其他算法。
结论将改进后的YOLOv5l算法引入大田环境下的水稻稻穗检测是可行的,具有较高的精确率、较快的检测速度和较小的内存占用,能够避免传统人工检测的主观性,对稻穗检测和水稻的无损估产具有重要意义。
Abstract:ObjectiveYOLOv5l algorithm model was introduced and improved to realize accurate, efficient and nondestructive detection of rice panicles in field environment.
MethodTaking rice in the field as the research object, rice image samples were collected by digital single-mirror reflex camera. The original image data were augmented and expanded offline after manual labeling, so as to construct an image data set for field rice. The YOLOv5l algorithm was improved adaptively, the effective channel attention (ECA) mechanism was put in front of the spatial pyramid pooling (SPP) layer and in the cross-stage-partial-connections (CSP) layer, and a comparative experiment was conducted. The optimal algorithm was selected as the benchmark model to carry out attention mechanism and data-enhanced ablation experiments, and the optimal performance model was obtained by testing. The improved YOLOv5l was compared with YOLOv5l, YOLOv5x, SSD and Faster R-CNN.
ResultIn the improved rice recognition framework of YOLOv5l, placing ECA before the network SPP layer resulted in better performance. Using test set images to verify the model, the average accuracy of recognition results was 93.63%, the average recall rate was 90.94%, and the overall average accuracy reached 95.05%. Compared with the non-fused YOLOv5l algorithm, the average accuracy of the improved YOLOv5l algorithm was 3.03 percent higher and the detection rate was 8.20 frames per ms faster. Compared with the YOLOv5x algorithm, the average precision of the improved YOLOv5l algorithm was improved by 0.62 percent, the detection rate was faster by 5.41 frames per ms, and the memory occupation was reduced by 74.1 MB. The results showed that the comprehensive performance of the improved YOLOv5l algorithm was better than other algorithms in rice panicle detection in the field.
ConclusionIt is feasible to introduce the improved YOLOv5l algorithm into rice panicle detection in field environment. The algorithm has high accuracy, fast detection speed and small memory occupation, which can avoid the subjectivity of traditional manual detection and is of great significance for rice panicle detection and non-destructive yield estimation.
-
Keywords:
- Rice /
- Yield estimation /
- Rice panicle detection /
- YOLOv5l /
- Efficient channel attention /
- Attention mechanism
-
催乳素(Prolactin,PRL)又称促乳素,是由垂体前叶嗜酸性细胞合成与分泌的一种蛋白质激素,广泛存在于多种组织器官,在动物生殖、妊娠和哺乳、生长发育、内分泌和代谢、免疫调节、水电解质平衡、促进血管生成、行为和癌症等方面具有重要的调控作用[1-2],表现出广泛的生物多效性,但其多效性机制仍不十分清楚。
PRL作为经典的生殖激素可通过内分泌途径调控卵泡发育,也可以作为细胞因子对卵泡发育起局部调控作用。在妊娠期或哺乳期时,PRL的循环水平显著升高,高水平的PRL会通过抑制下丘脑促性腺激素释放激素(GnRH)的分泌而降低垂体促性腺激素(Gn)的分泌,进而抑制卵泡发育[2-3]。PRL在卵巢上可通过自分泌和旁分泌途径来调节卵巢功能[4-5],PRL通过与卵泡上的催乳素受体(PRLR)结合能够影响类固醇激素(如E2和P4)的合成[6],从而影响发育卵泡的数量和排卵时间。垂体PRL的分泌受下丘脑、神经递质、激素、细胞因子、生殖生理状态、昼夜节律、应激等多种因素影响,具有脉冲式波动的特点,正常PRL脉冲性释放对泌乳和卵巢功能起到重要的调节作用[7]。在临床上,PRL抑制性调节的减弱可引发高催乳素血症(Hyperprolactinemia,HPRL)[8],可造成女性月经不调甚至闭经、不孕不育、乳房肿大、溢乳和男性性功能障碍等[9]。
PRL是经产母猪卵巢卵泡功能的重要生理调控因子,母猪断奶后PRL水平降低,卵泡才能得以继续发育,在生产上通过调控母猪断奶后的PRL水平,能够实现卵泡发育和发情的同步化[10]。研究表明,PRL-PRLR系统存在于猪卵泡中,并且卵泡中的PRL可由颗粒细胞产生;PRL作用于卵巢,从而调节颗粒细胞FSHR和黄体细胞LHR的表达,调节卵泡的发育和排卵与孕酮的合成,此调节作用严格依赖于卵泡内PRL的激素水平[10]。此外,卵泡发育的一个关键过程是血管生成,PRL可能作为局部调节因子影响卵泡血管内皮细胞生长[11-12]。
PRL具有显著的分子多态性,包括不同百分比的糖基化形式,这可能是PRL多效性的基础[13]。PRL的糖基化形式对生理学研究和潜在的临床应用非常重要,然而,当前市场上的重组PRL多为原核表达产物,缺少糖基化修饰,难以保证其生物活性,这严重阻碍了PRL多效性的机制研究和临床应用。因此,本研究利用真核表达载体系统构建能够稳定表达猪源PRL重组蛋白的中国仓鼠卵巢(Chinese hamster ovary,CHO)细胞系,并验证猪源PRL重组蛋白的生物活性,以期为猪PRL的功能研究及生产应用提供理论和参考依据。
1. 材料与方法
1.1 试验材料
人胚肾细胞(HEK-293T)、中国仓鼠卵巢细胞(CHO-K1)、慢病毒表达载体pCDH-CMV-MCS-EF1-GFP+Puro和辅助质粒psPAX2、pMD2.G均由华南农业大学动物科学学院动物繁殖研究室保存;小鼠乳腺上皮细胞(HC11)购自武汉普诺赛生命科技有限公司;DH5α感受态细胞和Stbl3感受态细胞购自擎科生物科技有限公司;母猪垂体组织采集自广东省农业科学院动物科学(畜牧)研究所三元杂商品母猪。
DMEM高糖培养基、DMEM/F12培养基、RPMI 1640培养基、PBS缓冲液、胎牛血清(FBS)、胰酶−EDTA(含2.5 g/L胰酶)、青链霉素(双抗)购自Gibco公司;Premix TaqTM、DNA Loading Buffer、DNA Marker、pMD18-T、限制性内切酶EcoR I和BamH I购自宝生物工程(大连)有限公司;T4连接酶购自Thermo Fisher公司;慢病毒专用转染试剂LentiFitTM购自汉恒生物科技有限公司;HiScript®Ⅲ1st Strand cDNA Synthesis Kit(+gDNA wiper)反转录试剂盒、HiScript®ⅡQ RT SuperMix for qPCR(+gDNA wiper)反转录试剂盒、ChamQTM Universal SYBR® qPCR Master Mix购自南京诺唯赞生物有限公司;动物组织总RNA提取试剂盒、细胞总RNA提取试剂盒、无内毒素质粒小提中量试剂盒购自天根生化科技(北京)有限公司;凝胶DNA微量回收试剂盒购自广州美基生物科技有限公司;全蛋白提取试剂盒、BAC蛋白含量检测试剂盒购自凯基生物技术股份有限公司;SDS-PAGE凝胶配制试剂盒购自北京鼎国昌盛生物技术有限责任公司;嘌呤霉素、鼠源His-Tag单克隆抗体、兔源α-tubulin多克隆抗体、HRP−标记山羊抗小鼠IgG、HRP−标记山羊抗兔IgG、CCK-8试剂盒购自碧云天生物技术有限公司;兔源β−酪蛋白(CSN2)多克隆抗体购自博奥森生物技术有限公司;兔源Cyclin D1多克隆抗体购自正能生物技术有限公司;Western-blot Marker购自Fermentas公司。引物由上海生工生物股份有限公司合成。
1.2 试验方法
1.2.1 PRL基因的引物设计
根据GenBank数据库中猪PRL基因序列(登录号NM_213926.1),使用Premier 5.0软件设计1对引物Sus-PRL-F:5′-ATCACCGCCATGGACAACAC-3′,Sus-PRL-R:5′-GACGTGGGCTTAGCAGTTGC-3′,扩增出含编码PRL蛋白(CDs序列)的基因片段(708 bp);根据编码PRL蛋白的碱基序列再设计另1对引物HPH-F-EcoR I:5′-CGG/AATTCCGATG
$\uwave {\rm{CATCATCA}}$ $\uwave {\rm{CCATCACCAT}}$ ATGGACAACACAGGGTCGTCACAGA-3′,HPH-R-BamH I:5′-CGG/GATCCCGTTA$\uwave {\rm{ATGGTGATGGTGATGATG}}$ GCAGTTGCTGTCGTAGATGATTCGG-3′,分别在该引物中引入6His-Tag、EcoR I和BamH I酶切位点(注:下划直线处分别为EcoR I和BamH I酶切位点,其中“/”为酶切识别位点;下划波浪线处为6His-Tag序列),以扩增含EcoR I-6His-PRL-6His-BamH I的基因序列(749 bp)。1.2.2 pMD18-T-PRL 克隆载体的构建与鉴定
采集母猪垂体组织,按照动物组织总RNA提取试剂盒说明书提取RNA,对RNA的浓度进行检测并按照HiScript®Ⅲ 1st Strand cDNA Synthesis Kit(+gDNA wiper)反转录试剂盒说明书反转录成cDNA。以猪垂体组织cDNA为模板,用引物Sus-PRL-F和Sus-PRL-R扩增编码PRL蛋白的基因片段。扩增体系为:20 μL 2× Premix TaqTM,4 μL cDNA(500 ng/μL),上、下游引物(10 μmol/L)各2 μL,12 μL ddH2O。PCR反应程序为:98 ℃预变性30 s;98 ℃变性10 s,57 ℃退火30 s,72 ℃延伸1 min,共35个循环;72 ℃终延伸2 min。将扩增产物回收纯化后连接到pMD18-T载体,连接体系(10 μL):4.5 μL目的基因,0.5 μL pMD18-T载体,5 μL Solution I;16 ℃连接16 h。随后将连接产物转化到DH5α感受态细胞,经PCR鉴定为阳性的菌液送至上海生工生物股份有限公司进行测序,DNA测序后序列比对正确的克隆载体命名为pMD18-T-PRL。
1.2.3 pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro真核表达载体的构建
以pMD18-T-PRL克隆载体为模板,用引物HPH-F-EcoR I和HPH-R-BamH I扩增含EcoR I酶切位点-6His-PRL-6His-BamH I酶切位点序列的基因片段。扩增体系为:20 μL 2× Premix TaqTM,4 μL pMD18-T-PRL克隆载体(500 ng/μL),上、下游引物(10 μmol/L)各2 μL,12 μL ddH2O。PCR反应程序:98 ℃预变性30 s;98 ℃变性10 s,72 ℃退火30 s,72 ℃延伸1 min,共35个循环;72 ℃终延伸2 min。将扩增产物回收纯化后,通过内切酶EcoR I和BamH I双酶切后将6His-PRL-6His基因连接到pCDH-CMV-MCS-EF1-GFP+Puro慢病毒表达载体中。双酶切体系:1 μL EcoR I,1 μL BamH I,2 μL 10× K Buffer,1000 ng cDNA或质粒,加入ddH2O至20 μL,37 ℃水浴酶切2 h;连接反应体系(20 μL):2 μL 10× T4 DNA Ligase Buffer,0.2 μL T4 DNA Ligase,10 μL目的片段(40 ng/μL),0.53 μL载体(190 ng/μL),加入ddH2O至20 μL,16 ℃连接16 h。随后将连接产物转化到Stbl3感受态细胞,经PCR鉴定为阳性的菌液送至上海生工生物股份有限公司进行测序,DNA测序后序列比对正确的重组慢病毒表达载体命名为pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro。对序列正确的阳性菌进行扩培并按照无内毒素质粒小提中量试剂盒说明书抽提pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro质粒,并对其进行双酶切鉴定。
1.2.4 慢病毒的包装及滴度测定
取对数生长期的HEK-293T细胞接种于T75培养瓶中,用完全培养基(DMEM基础培养基、FBS和双抗的体积分数分别为89%、10%和1%)在37 ℃培养箱中将细胞培养至生长密度达70%~80%,按照慢病毒专用转染试剂LentiFitTM说明书操作,将60 μL转染试剂与16 μg pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro重组质粒或pCDH-CMV-MCS-EF1-GFP+Puro空载质粒和12 μg psPAX2、8 μg pMD2.G辅助质粒充分混匀,室温孵育20 min后转染T75培养瓶中的HEK-293T细胞,培养12 h后更换为完全培养基,继续培养48 h后,收集细胞培养液,并更换新的完全培养基,72 h继续收集细胞培养液;收集的培养液过滤、离心浓缩得到PRL−慢病毒浓缩液,于−80 ℃条件下保存。
将慢病毒浓缩液10倍梯度稀释,连续做6个稀释度(每个浓度梯度每100 μL稀释液分别含病毒浓缩液10、1、0.1、0.01、0.001和0.000 1 μL),吸弃96孔板内培养基,分别加入6个浓度梯度的100 μL慢病毒稀释液,感染96孔板的HEK-293T细胞,24 h后更换完全培养液,继续培养至48 h后观察绿色荧光蛋白(GFP)表达情况,计数荧光比例约为30%的孔的细胞数,按照以下公式算出病毒滴度:病毒滴度(TU/mL)=细胞数×荧光百分比×103/病毒原液体积。
1.2.5 CHO-K1-PRL阳性细胞的制备与鉴定
取对数生长期的CHO-K1细胞接种于T25培养瓶中,用完全培养基(DMEM/F12基础培养基、FBS和双抗的体积分数分别为89%、10%和1%)在37 ℃培养箱中将细胞培养至生长密度达50%~70%,吸弃原培养基,加入2 mL DMEM/F12完全培养基(不含双抗)、12 μL Polybrene和200 μL浓缩慢病毒液的混合液,培养4 h后补齐培养液至4 mL,24 h后更换为完全培养液,48 h后观察GFP表达效率,并换成含有6 μg/mL 嘌呤霉素的完全培养液,进行药物筛选7 d,每2~4 d换液1次,得到阳性细胞系,命名为CHO-K1-PRL。
分别提取上述药物筛选后的CHO-K1-PRL细胞、未感染慢病毒的CHO-K1细胞和感染空载体慢病毒的CHO-K1细胞的总RNA与总蛋白,同时收集CHO-K1-PRL细胞、未感染慢病毒CHO-K1细胞和感染空载体慢病毒CHO-K1细胞的培养液。细胞总RNA反转录为cDNA,利用引物HPH-F-EcoR I /HPH-R-BamH I进行RT-PCR检测(β-actin为内参基因,引物β-actin-F:5′-CCACCATGTACCCAGGCATT-3′,β-actin-R:5′-CGGACTCATCGTACTCCTGC-3′);以鼠源His-Tag单克隆抗体(用TBST缓冲液按照体积比1:1 000稀释)为一抗,再以HRP−标记山羊抗小鼠IgG(用TBST缓冲液按照体积比1:1 000稀释)为二抗,对细胞总蛋白及细胞培养液样品进行Western blot鉴定。
1.2.6 重组蛋白的纯化与鉴定
收集大量CHO-K1-PRL阳性细胞的培养液,用0.45 μm滤器过滤,由重庆革兰氏生物科技有限责任公司利用镍柱亲和层析法对培养液通过His-Tag蛋白进行蛋白纯化;对纯化蛋白用体积分数为12% 的蛋白凝胶进行SDS-PAGE电泳检测,并将目的条带切下,送至深圳市微纳菲生物技术有限公司进行LC-MS/MS质谱分析鉴定。
1.2.7 重组PRL对HC11细胞增殖及酪蛋白表达的影响
参照田青等[14-15]的方法,取对数生长期的HC11细胞以每孔1×104的密度接种于96孔板,或以每孔2×105的密度接种于6孔板,先用完全培养基(RPMI 1640、FBS和双抗的体积分数分别为89%、10%和1%)将细胞培养至生长密度达80%左右,再用无血清无激素含双抗的RPMI 1640培养基进行饥饿处理16 h,弃去旧培养基,增殖试验(96孔板):用含不同浓度重组PRL的激素诱导培养基(RPMI 1640、FBS和双抗的体积分数分别为89%、10%和1%,5 μg/mL 胰岛素,1 ng/mL氢化可的松,0/100/300/500 ng/mL重组PRL)培养20 h,每孔加入10 μL CCK-8试剂,继续培养4 h后,用酶标仪于450 nm波长测吸光度;诱导酪蛋白表达试验(6孔板):用含不同浓度重组PRL的激素诱导培养基(同增殖试验)培养24 h后收集细胞,提取细胞的总RNA与总蛋白,细胞总RNA反转录为cDNA,qRT-PCR检测PRLR、CSN1S1、CSN2、CSN3、Cyclin D1、PCNA等基因的相对表达量,GAPDH为内参基因,引物序列见表1,各基因的相对表达量用2−ΔΔCt值表示;以兔源β−酪蛋白(CSN2)多克隆抗体或兔源Cyclin D1多克隆抗体(用TBST缓冲液按照体积比1:1 000稀释)为一抗,再以HRP−标记山羊抗兔IgG(用TBST缓冲液按照体积比1:1 000稀释)为二抗,对细胞总蛋白样品进行Western blot检测,并通过ImageJ软件进行灰度值分析,计算CSN2和Cyclin D1蛋白的相对表达水平。
表 1 qPCR引物序列Table 1. Primer sequence for qPCR基因
Gene正向引物序列(5′→ 3′)
Forward primer sequence反向引物序列(5′→ 3′)
Reverse primer sequence产物大小/bp
Product lengthGAPDH GAGCGAGACCCCACTAACATC GCGGAGATGATGACCCTTTT 134 PRLR GTGGAATCCTGGGTCAGATG GGGCCACTGGTTTTGTAGTC 108 CSN1S1 CCTTTCCCCTTTGGGCTTAC TGAGGTGGATGGAGAATGGA 193 CSN2 GCAATCCCGTCCCACAAAAC GGGGCATCTGTTTGTGCTTG 138 CSN3 CCTTTTTGTGCCGTGGTGAG GGCTGGAGACCTAAGCAGAA 197 Cyclin D1 TGGCTAAACAAGGACCCCC ATGTCCACATCTCGCACGTC 203 PCNA AAAGATGCCGTCGGGTGAAT TGGTTACCGCCTCCTCTTCT 179 细胞增殖试验设空白组(不加细胞,加培养液)、对照组(无激素)和试验组(100、300、500 ng/mL重组PRL),每组设5个重复,每个重复1个孔,试验重复3次,细胞相对增殖率(Relative growth rate,RGR)=D450 nm(试验组)/D450 nm(对照组);诱导酪蛋白表达试验设对照组(无激素)和试验组(100、300、500 ng/mL重组PRL),每组设3个重复,每个重复1个孔,试验重复3次。
1.3 统计学分析
试验数据采用SPSS22.0进行单因素方差分析(One-way ANOVA),采用LSD法进行多重比较分析,数据结果用平均值±标准误表示。
2. 结果与分析
2.1 pMD18-T-PRL克隆载体的构建与鉴定
以猪垂体cDNA为模板,用引物Sus-PRL-F/Sus-PRL-R扩增猪源PRL基因片段(图1A),回收连接到pMD18-T载体后转化到DH5α感受态细胞,菌液PCR鉴定结果如图1B所示,分别提取5、6、9~16号菌液质粒进行DNA测序鉴定,并将DNA测序结果经Blast比对发现5、6、11号菌液质粒插入片段均仅有1个碱基突变(A突变为G),导致三联密码改变(ATG变为GTG,成熟肽第13氨基酸Met变为Val),而插入基因片段编码的PRL成熟肽段氨基酸序列与PRL亚型(XP_005665681.1)一致,pMD18-T-PRL克隆载体构建成功。
![图 1 pMD18-T-PRL克隆载体的构建与PCR鉴定]() 图 1 pMD18-T-PRL克隆载体的构建与PCR鉴定A:PRL基因的PCR产物,M为DL 2000 DNA Marker,1~4以母猪垂体组织cDNA为模板;B:pMD18-T-PRL菌液PCR产物,M为DL 2000 DNA Marker,1~16分别以挑选的不同pMD18-T-PRL菌落的菌液为模板Figure 1. Construction and PCR identification of pMD18-T-PRL cloning plasmidA: PCR products of PRL gene, M is DL 2000 DNA Marker, 1−4 use sow pituitary tissue cDNA as template; B: PCR products of pMD18-T-PRL colonies, M is DL 2000 DNA Marker, 1−16 respectively use different pMD18-T-PRL colonies as template
图 1 pMD18-T-PRL克隆载体的构建与PCR鉴定A:PRL基因的PCR产物,M为DL 2000 DNA Marker,1~4以母猪垂体组织cDNA为模板;B:pMD18-T-PRL菌液PCR产物,M为DL 2000 DNA Marker,1~16分别以挑选的不同pMD18-T-PRL菌落的菌液为模板Figure 1. Construction and PCR identification of pMD18-T-PRL cloning plasmidA: PCR products of PRL gene, M is DL 2000 DNA Marker, 1−4 use sow pituitary tissue cDNA as template; B: PCR products of pMD18-T-PRL colonies, M is DL 2000 DNA Marker, 1−16 respectively use different pMD18-T-PRL colonies as template2.2 pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro慢病毒表达载体的构建与鉴定
以pMD18-T-PRL克隆载体为模板,用引物HPH-F-EcoR I/HPH-R-BamH I扩增出EcoR I-6His-PRL-6His-BamH I序列片段(图2A);用EcoR I和BamH I内切酶分别对纯化回收的目的片段与pCDH-CMV-MCS-EF1-GFP+Puro慢病毒表达载体进行双酶切,分别得到6His-PRL-6His片段(图2B)和线性化pCDH-CMV-MCS-EF1-GFP+Puro质粒片段(图2C),并进行连接后转化到Stbl3感受态细胞,挑取单菌落进行菌液PCR鉴定(图2D),阳性菌液DNA测序结果经Blast比对,插入片段与pMD18-T-PRL中的测序序列一致;对重组质粒进行双酶切后,在约739 bp处有一条特异性条带,与目的基因大小一致(图2E),表明pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro慢病毒表达载体构建成功。
![图 2 pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro慢病毒表达载体的构建与PCR鉴定]() 图 2 pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro慢病毒表达载体的构建与PCR鉴定A:EcoR I-6His-PRL-6His-BamH I序列片段PCR产物,M为DL 2000 DNA Marker,1为EcoR I-6His-PRL-6His-BamH I序列片段;B:EcoR I-6His-PRL-6His-BamH I序列片段双酶切产物,M为DL 2000 DNA Marker,1为6His-PRL-6His片段;C:pCDH-CMV-MCS-EF1-GFP+Puro质粒双酶切产物,M为DL 15000 DNA Marker,1为pCDH-CMV-MCS-EF1-GFP+Puro线性片段;D:pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro菌液PCR产物,M为DL 2000 DNA Marker,1~12分别以挑选的不同单菌落的菌液为模板;E:重组质粒pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro的双酶切结果,M1为DL 15000 DNA Marker,M2为DL 2000 DNA Marker,1和2为重组质粒双酶切片段Figure 2. Construction and PCR identification of pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro lentivirus expression plasmidA: PCR product of EcoR I-6His-PRL-6His-BamH I sequence fragment, M is DL 2000 DNA Marker, 1 is EcoR I-6His-PRL-6His-BamH I sequence fragment; B: Double digested product of EcoR I-6His-PRL-6His-BamH I sequence fragment, M is DL 2000 DNA Marker, 1 is 6His-PRL-6His sequence fragment; C: Double digested product of pCDH-CMV-MCS-EF1-GFP+Puro plasmid, M is DL 15000 DNA Marker, 1 is pCDH-CMV-MCS-EF1-GFP+Puro plasmid linear fragment; D: PCR products of pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro colonies, M is DL 2000 DNA Marker, 1−12 respectively use selected bacterial solutions from different single colonies as templates; E: Double digested product of recombinant vector pCDH-CMV-MCS-EF1-GFP+Puro, M1 is DL 15000 DNA Marker, M2 is DL 2000 DNA Marker, 1 and 2 are double digested products of recombinant vector
图 2 pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro慢病毒表达载体的构建与PCR鉴定A:EcoR I-6His-PRL-6His-BamH I序列片段PCR产物,M为DL 2000 DNA Marker,1为EcoR I-6His-PRL-6His-BamH I序列片段;B:EcoR I-6His-PRL-6His-BamH I序列片段双酶切产物,M为DL 2000 DNA Marker,1为6His-PRL-6His片段;C:pCDH-CMV-MCS-EF1-GFP+Puro质粒双酶切产物,M为DL 15000 DNA Marker,1为pCDH-CMV-MCS-EF1-GFP+Puro线性片段;D:pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro菌液PCR产物,M为DL 2000 DNA Marker,1~12分别以挑选的不同单菌落的菌液为模板;E:重组质粒pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro的双酶切结果,M1为DL 15000 DNA Marker,M2为DL 2000 DNA Marker,1和2为重组质粒双酶切片段Figure 2. Construction and PCR identification of pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro lentivirus expression plasmidA: PCR product of EcoR I-6His-PRL-6His-BamH I sequence fragment, M is DL 2000 DNA Marker, 1 is EcoR I-6His-PRL-6His-BamH I sequence fragment; B: Double digested product of EcoR I-6His-PRL-6His-BamH I sequence fragment, M is DL 2000 DNA Marker, 1 is 6His-PRL-6His sequence fragment; C: Double digested product of pCDH-CMV-MCS-EF1-GFP+Puro plasmid, M is DL 15000 DNA Marker, 1 is pCDH-CMV-MCS-EF1-GFP+Puro plasmid linear fragment; D: PCR products of pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro colonies, M is DL 2000 DNA Marker, 1−12 respectively use selected bacterial solutions from different single colonies as templates; E: Double digested product of recombinant vector pCDH-CMV-MCS-EF1-GFP+Puro, M1 is DL 15000 DNA Marker, M2 is DL 2000 DNA Marker, 1 and 2 are double digested products of recombinant vector2.3 慢病毒包装及滴度测定
将慢病毒重组载体或空载体与辅助质粒共转染293T细胞,包装含有6His-PRL-6His片段的PRL-慢病毒,共转染24 h后,在荧光倒置显微镜下可见293T细胞中绿色荧光蛋白表达情况良好(图3),转染效率为30%~50%。对PRL−慢病毒浓缩液进行滴度测定,慢病毒滴度约为9.9×108 TU/mL,满足后续试验要求。
![图 3 慢病毒载体转染293T细胞24 h荧光表达情况]() 图 3 慢病毒载体转染293T细胞24 h荧光表达情况空白:未做处理的293T细胞;空载:转染空慢病毒载体的293T细胞;PRL:转染pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro慢病毒表达载体的293T细胞Figure 3. Fluorescent expression of 293T cells transfected with lentivirus plasmid for 24 hControl: Untreated 293T cells; Empty vector: 293T cells transfected with empty lentiviral vector; PRL: 293T cells transfected with pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro lentiviral vector
图 3 慢病毒载体转染293T细胞24 h荧光表达情况空白:未做处理的293T细胞;空载:转染空慢病毒载体的293T细胞;PRL:转染pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro慢病毒表达载体的293T细胞Figure 3. Fluorescent expression of 293T cells transfected with lentivirus plasmid for 24 hControl: Untreated 293T cells; Empty vector: 293T cells transfected with empty lentiviral vector; PRL: 293T cells transfected with pCDH-CMV-6His-PRL-6His-EF1-GFP+Puro lentiviral vector2.4 慢病毒感染CHO-K1细胞及阳性细胞系的筛选
用浓缩慢病毒液感染CHO-K1细胞72 h后,在倒置荧光显微镜下观察到感染慢病毒的CHO-K1细胞中绿色荧光蛋白表达情况良好(图4),感染效率达30%~50%,初步认定猪源PRL基因成功导入CHO-K1细胞中。PRL−慢病毒感染的CHO-K1细胞用嘌呤霉素进行连续7 d药物筛选后,表达绿色荧光蛋白的细胞达95%以上,获得阳性细胞系并命名为CHO-K1-PRL。
![图 4 慢病毒浓缩液感染CHO-K1细胞72 h及药筛7 d后荧光表达情况]() 图 4 慢病毒浓缩液感染CHO-K1细胞72 h及药筛7 d后荧光表达情况空白:未做处理的CHO-K1细胞;空载:感染空载体慢病毒液的CHO-K1细胞;PRL:感染PRL−慢病毒液的CHO-K1细胞;PRL(药筛后):感染PRL−慢病毒液的CHO-K1细胞经嘌呤霉素药物筛选7 d后Figure 4. Fluorescence expression of CHO-K1 cells infected with concentrated lentivirus solution for 72 h and seven days after drug screeningControl: Untreated CHO-K1 cells; Empty vector: CHO-K1 cells infected by the concentrated empty lentivirus solution; PRL: CHO-K1 cells infected by the concentrated PRL-lentivirus solution; PRL (after drug screening): CHO-K1 cells infected with PRL-lentivirus solution and then screened by purinomycin for seven days
图 4 慢病毒浓缩液感染CHO-K1细胞72 h及药筛7 d后荧光表达情况空白:未做处理的CHO-K1细胞;空载:感染空载体慢病毒液的CHO-K1细胞;PRL:感染PRL−慢病毒液的CHO-K1细胞;PRL(药筛后):感染PRL−慢病毒液的CHO-K1细胞经嘌呤霉素药物筛选7 d后Figure 4. Fluorescence expression of CHO-K1 cells infected with concentrated lentivirus solution for 72 h and seven days after drug screeningControl: Untreated CHO-K1 cells; Empty vector: CHO-K1 cells infected by the concentrated empty lentivirus solution; PRL: CHO-K1 cells infected by the concentrated PRL-lentivirus solution; PRL (after drug screening): CHO-K1 cells infected with PRL-lentivirus solution and then screened by purinomycin for seven days2.5 CHO-K1-PRL细胞的鉴定
以未感染慢病毒的CHO-K1细胞和感染空载体慢病毒的CHO-K1细胞为对照,对CHO-K1-PRL细胞进行RT-PCR(β-actin 作为内参基因)和Western blot鉴定。结果显示,只有CHO-K1-PRL细胞能扩增出与预期相符的条带(749 bp)(图5),并且只有CHO-K1-PRL阳性细胞的细胞蛋白与培养液有且均有检测到His-Tag蛋白(图6),相对分子质量约为25 000(猪PRL相对分子质量为23 000)。
![图 5 CHO-K1-PRL细胞的RT-PCR鉴定]() 图 5 CHO-K1-PRL细胞的RT-PCR鉴定M为DL 2000 DNA Marker; 1、3、4、6和7为749 bp 的EcoR I-6His-PRL-6His-BamH I片段,2、5和8为98 bp的β-actinFigure 5. RT-PCR identification of CHO-K1-PRL cellsM is DL 2000 DNA Marker; 1, 3, 4, 6 and 7 are the 749 bp EcoR I-6His-PRL-6His-BamH I fragments; 2, 5 and 8 are the 98 bp β-actin fragments
图 5 CHO-K1-PRL细胞的RT-PCR鉴定M为DL 2000 DNA Marker; 1、3、4、6和7为749 bp 的EcoR I-6His-PRL-6His-BamH I片段,2、5和8为98 bp的β-actinFigure 5. RT-PCR identification of CHO-K1-PRL cellsM is DL 2000 DNA Marker; 1, 3, 4, 6 and 7 are the 749 bp EcoR I-6His-PRL-6His-BamH I fragments; 2, 5 and 8 are the 98 bp β-actin fragments2.6 猪源PRL重组蛋白的纯化与LC-MS/MS质谱分析
收集CHO-K1-PRL细胞培养液,利用镍柱亲和层析对培养液通过His-Tag进行纯化,获得纯化的重组蛋白(质量浓度为50 μg/mL),对纯化蛋白进行SDS-PAGE(图7),并切取目的条带进行LC-MS/MS质谱分析,经与蛋白质数据库比对分析,重组蛋白与天然猪PRL(XP_005665681.1)比对覆盖率达94%,说明表达产物为猪源PRL重组蛋白。
![图 6 CHO-K1-PRL细胞中His-Tag蛋白Western blot鉴定]() 图 6 CHO-K1-PRL细胞中His-Tag蛋白Western blot鉴定A:细胞裂解液:B:细胞培养液;M为Western blot marker,1为未做处理的CHO-K1细胞,2为感染空载体慢病毒液的CHO-K1细胞,3为CHO-K1-PRL;PRL-6His重组蛋白和内参α-tubulin相对分子质量分别约为25 000和55 000Figure 6. Western blot identification of His-Tag protein in CHO-K1-PRL cellsA: Cell lysate; B: Cell culture fluid; M is Western blot marker, 1 is untreated CHO-K1 cells, 2 is CHO-K1 cells infected by the concentrated empty lentivirus solution, 3 is CHO-K1-PRL; The relative molecular weights of PRL-6His recombinant protein and α-tubulin are about 25 000 and 55 000 resprectively
图 6 CHO-K1-PRL细胞中His-Tag蛋白Western blot鉴定A:细胞裂解液:B:细胞培养液;M为Western blot marker,1为未做处理的CHO-K1细胞,2为感染空载体慢病毒液的CHO-K1细胞,3为CHO-K1-PRL;PRL-6His重组蛋白和内参α-tubulin相对分子质量分别约为25 000和55 000Figure 6. Western blot identification of His-Tag protein in CHO-K1-PRL cellsA: Cell lysate; B: Cell culture fluid; M is Western blot marker, 1 is untreated CHO-K1 cells, 2 is CHO-K1 cells infected by the concentrated empty lentivirus solution, 3 is CHO-K1-PRL; The relative molecular weights of PRL-6His recombinant protein and α-tubulin are about 25 000 and 55 000 resprectively2.7 猪源PRL重组蛋白对HC11细胞增殖及酪蛋白合成的影响
在HC11体外培养体系中分别加入不同质量浓度的重组PRL(0、100、300、500 ng/mL)处理24 h后,发现添加重组PRL能促进HC11细胞增殖(P<0.05)(图8A),并且与细胞增殖相关的Cyclin D1、PCNA基因及Cyclin D1蛋白表达水平显著上升(P<0.05)(图8B、8C、9A),与细胞相对增殖率的结果相一致。
![图 8 不同质量浓度重组PRL对HC11细胞增殖及酪蛋白表达的影响]() 图 8 不同质量浓度重组PRL对HC11细胞增殖及酪蛋白表达的影响柱子上方(图A)、相同蛋白(图B)或基因(图C)柱子上方的不同小写字母表示组间差异显著(P<0.05,LSD法),不同大写字母表示组间差异极显著(P<0.01,LSD法)Figure 8. Different mass concentrations of recombinant PRL on proliferation and casein expressions of HC11 cellsDifferent lowercase letters on bars (figure A), or bars of the same protein (figure B) or gene (figure C) indicate significant differences among groups (P<0.05, LSD method), different capital letters indicate highly significant differences among groups (P<0.01, LSD method)
图 8 不同质量浓度重组PRL对HC11细胞增殖及酪蛋白表达的影响柱子上方(图A)、相同蛋白(图B)或基因(图C)柱子上方的不同小写字母表示组间差异显著(P<0.05,LSD法),不同大写字母表示组间差异极显著(P<0.01,LSD法)Figure 8. Different mass concentrations of recombinant PRL on proliferation and casein expressions of HC11 cellsDifferent lowercase letters on bars (figure A), or bars of the same protein (figure B) or gene (figure C) indicate significant differences among groups (P<0.05, LSD method), different capital letters indicate highly significant differences among groups (P<0.01, LSD method)通过qPCR和Western blot检测,结果显示,添加300、500 ng/mL 重组PRL极显著下调了PRLR的表达水平(P<0.01)(图8C);添加100、300 ng/mL重组PRL处理对HC11中CSN1S1、CSN2基因表达水平无显著影响,而CSN3基因表达水平显著上调(P<0.05) (图8C),但CSN2蛋白表达水平显著高于对照组(P<0.05)(图8B、9B);添加500 ng/mL重组PRL显著上调了酪蛋白基因(CSN1S1、CSN2、CSN3)的表达水平(P<0.05)(图8C),而CSN2蛋白表达水平与对照组差异不显著(图8B、9B)。
3. 讨论与结论
PRL主要是由垂体前叶分泌的一类蛋白质激素,具有广泛的生物活性。PRL具有显著的分子多态性,包括不同百分比的糖基化形式,这可能是PRL多效性的基础[13]。当前市面上多为原核表达的PRL重组蛋白,缺乏蛋白质翻译后修饰,表达的蛋白既不能进行正确折叠,也不能被糖基化,并且通常由于二硫键错配而以包涵体形式存在[16-17],需要进一步溶解和复性,蛋白质的生物活性难以保证。为了获得与天然猪源PRL更相近的具有活性的重组蛋白,本研究利用CHO细胞真核表达系统体外表达了猪源PRL蛋白。
CHO真核表达系统不仅具有蛋白质翻译后修饰功能,而且表达的蛋白质具有完整的生物学功能[18];糖基化是蛋白质翻译后修饰的最重要形式之一,其直接影响重组蛋白的空间构象、稳定性、聚性和生物学活性以及体内特性(包括药物代谢动力学和免疫原性)[19]。慢病毒载体是以慢病毒的基因组为基础,由目的基因取代部分病毒基因结构构成的一类载体[20],能够携带大片段的外源基因,并且不容易诱发宿主的免疫反应,更重要的是其能够将目的基因高效整合到宿主细胞的染色体中,且不易引起插入突变,理论上能够在细胞内永久表达并在子代中稳定表达[21];而且改造后的慢病毒是假病毒,没有复制能力,具备较好的生物安全性,是一种理想的基因转移载体[22]。因此,我们选用慢病毒载体系统进行构建稳定表达猪源PRL重组蛋白的CHO-K1-PRL细胞系。
目前大多数慢病毒载体根据HIV-1基因组改装而来,慢病毒感染宿主细胞后,能够整合自身基因组到宿主细胞中,从而使外源基因能够在宿主细胞中实现长期稳定地表达。事实上,慢病毒载体已成功用于表达人的PRL重组蛋白[23]。在本研究中,我们通过将猪PRL基因片段插入到pCDH-CMV-MCS-EF1-GFP+Puro慢病毒表达载体,经慢病毒包装获得含有猪PRL基因的PRL−慢病毒液,其感染CHO-K1细胞并经过嘌呤霉素药物筛选后得到稳定的猪PRL表达系统,并利用镍柱亲和层析法纯化猪PRL重组蛋白。Western blot鉴定发现该重组蛋白能与His-Tag抗体结合,条带的相对分子质量约为25 000;此外,对纯化得到的猪PRL重组蛋白进行LC-MS/MS质谱分析,结果显示该重组蛋白与天然猪PRL蛋白对比的覆盖率达94%,可以确定CHO-K1-PRL细胞培养液中含有猪源PRL蛋白。天然猪PRL相对分子质量为23 000,而本研究表达的PRL蛋白携带His-Tag蛋白,PRL-6His相对分子质量约为25 000。信号肽通常位于新合成肽链的N端,一般由15~30个氨基酸残基组成,能够引导细胞内新合成的蛋白质转移到内质网并分泌到细胞;信号肽序列在不同蛋白质之间可以通用,优化蛋白质外源表达可通过选择高效引导蛋白质分泌表达的信号肽序列替换蛋白质本身的信号肽[24]。本研究在构建慢病毒表达载体时,保留了PRL基因中的信号肽序列,在未经浓缩的CHO-K1-PRL阳性细胞的培养液中难以检测到重组蛋白,而纯化得到的PRL重组蛋白质量浓度为50 μg/mL,说明本研究的猪PRL表达系统蛋白表达量低,后续想要获得更高效的PRL表达系统,可以考虑通过替换PRL基因的信号肽序列进行优化。此外,由于在蛋白表达过程中,蛋白携带的His-Tag可能会被包裹在蛋白内部,可能是导致纯化的重组蛋白浓度低的原因之一[25]。
PRL以其促进乳腺的生长发育和泌乳而闻名[26],PRL与PRLR结合后,激活PRL介导的信号转导通路,刺激乳腺发育、乳蛋白的合成与分泌等[27]。PRL与乳腺分泌细胞膜上的PRLR结合,能够促进乳蛋白尤其是酪蛋白mRNA产生[28],乳蛋白的合成与酪蛋白基因的表达水平具有强相关性[29]。本研究中,为验证PRL重组蛋白的生物活性,将其添加到HC11细胞的体外培养体系中,处理24 h后,检测分析细胞相对增值率和细胞中酪蛋白基因(CSN1S1、CSN2、CSN3)mRNA表达及CSN2、Cyclin D1蛋白的表达水平。试验结果表明,添加重组PRL蛋白能够显著促进HC11细胞的增殖,同时也上调了HC11细胞中CSN1S1、CSN2、CSN3 mRNA及CSN2蛋白的表达水平,说明重组PRL对乳腺细胞增殖和酪蛋白的表达具有显著的诱导作用。
综上,本研究利用真核表达系统成功表达了具有生物学活性的猪源PRL重组蛋白,为猪PRL的功能研究和生产应用奠定了基础。
-
表 1 ECA模块融合结果对比
Table 1 Comparison of ECA module fusion results
% 网络模型
Network model精确率
Precision召回率
Recall平均精度
Average precisionYOLOv5l 90.19 88.99 92.02 ECA-YOLOv5l-Backbone 93.58 89.56 94.47 ECA-YOLOv5l-SPP 93.63 90.94 95.05 ECA-YOLOv5l-Neck 92.64 90.46 94.33 表 2 YOLOv5l消融试验1)
Table 2 YOLOv51 ablation experiment
% 注意力
机制
ECA数据增强
Data
augmentation精确率
Precision召回率
Recall平均精度
Average
precision× × 88.32 84.21 88.18 × √ 90.19 88.99 92.02 √ √ 93.63 90.94 95.05 1) “√”和“×”分别表示使用和未使用
1) “√” and “×” indicates used and unused respectively表 3 不同网络模型检测性能对比
Table 3 Comparison of different networks
网络模型
Network
model平均精度/%
Average
precision检测速率/
(帧·ms−1)
Detection rate内存/MB
Memory
sizeFaster R-CNN 86.65 11.72 109.0 SSD 88.43 10.83 100.0 YOLOv5l 92.02 15.30 89.3 YOLOv5x 94.43 18.09 166.0 改进 YOLOv51
Improved YOLOv5195.05 23.50 91.9 -
[1] 郭雷风. 面向农业领域的大数据关键技术研究[D]. 北京: 中国农业科学院, 2016. [2] 章元, 许庆, 邬璟璟. 一个农业人口大国的工业化之路: 中国降低农村贫困的经验[J]. 经济研究, 2012, 47(11): 76-87. [3] 辛良杰, 李鹏辉. 中国居民口粮消费特征变化及安全耕地数量[J]. 农业工程学报, 2017, 33(13): 1-7. [4] 杨仕华, 廖琴. 中国水稻品种试验与审定[M]. 北京: 中国农业科学技术出版社, 2005. [5] IKEDA M, HIROSE Y, TAKASHI T, et al. Analysis of rice panicle traits and detection of QTLs using an image analyzing method[J]. Breeding Science, 2010, 60(1): 55-64. doi: 10.1270/jsbbs.60.55
[6] ZHOU X G. First report of bacterial panicle blight of rice caused by Burkholderia glumae in South Africa[J]. Plant Disease, 2014, 98(4): 566.
[7] ZHANG Y, LIU M, DANNENMANN M, et al. Benefit of using biodegradable film on rice grain yield and N use efficiency in ground cover rice production system[J]. Field Crops Research, 2017, 201: 52-59. doi: 10.1016/j.fcr.2016.10.022
[8] BOIL K, KANG S K, SANG W G, et al. Variation of panicle differentiation stage by leaf growth according to rice cultivars and transplanting time[J]. Korean Journal of Crop Science, 2013, 58(4): 353-361. doi: 10.7740/kjcs.2013.58.4.353
[9] 赵锋, 王克俭, 苑迎春. 基于颜色特征和AdaBoost算法的麦穗识别的研究[J]. 作物杂志, 2014(1): 141-144. doi: 10.3969/j.issn.1001-7283.2014.01.033 [10] 范梦扬, 马钦, 刘峻明, 等. 基于机器视觉的大田环境小麦麦穗计数方法[J]. 农业机械学报, 2015, 46(S1): 234-239. doi: 10.6041/j.issn.1000-1298.2015.S0.038 [11] ZHOU C Q, LIANG D, YANG X D, et al. Wheat ears counting in field conditions based on multi-feature optimization and TWSVM[J]. Frontiers in Plant Science, 2018, 9: 1024. doi: 10.3389/fpls.2018.01024.
[12] DUAN L F, HUANG C L, CHEN G X, et al. Determination of rice panicle numbers during heading by mufti-angle imaging[J]. The Crop Journal, 2015, 3(3): 211-219. doi: 10.1016/j.cj.2015.03.002
[13] LI Q Y, CAI J H, BERGER B, et al. Detecting spikes of wheat plants using neural networks with laws texture energy[J]. Plant methods, 2017, 13(1): 1-13. doi: 10.1186/s13007-016-0152-4
[14] OLSEN P A, RAMAMURTHY K N, RIBERA J, et al. Detecting and counting panicles in sorghum images[C]//2018 IEEE 5th International Conference on Data Science and Advanced Analytics (DSAA). Turin, Italy: IEEE, 2019: 400-409.
[15] 李静, 陈桂芬, 安宇. 基于优化卷积神经网络的玉米螟虫害图像识别[J]. 华南农业大学学报, 2020, 41(3): 110-116. [16] 顾伟, 王巧华, 李庆旭, 等. 基于改进SSD的棉种破损检测[J]. 华中农业大学学报, 2021, 40(3): 278-285. [17] 周云成, 许童羽, 邓寒冰, 等. 基于面向通道分组卷积网络的番茄主要器官实时识别[J]. 农业工程学报, 2018, 34(10): 153-162. [18] KOIRALA A, WALSH K B, WANG Z, et al. Deep learning for real-time fruit detection and orchard fruit load estimation: Benchmarking of ‘MangoYOLO’[J]. Precision Agriculture, 2019, 20(6): 1107-1135. doi: 10.1007/s11119-019-09642-0
[19] TIAN Y N, YANG G D, WANG Z, et al. Apple detection during different growth stages in orchards using the improved YOLO-V3 model[J]. Computers and Electronics in Agriculture, 2019, 157: 417-426. doi: 10.1016/j.compag.2019.01.012
[20] 张领先, 陈运强, 李云霞, 等. 基于卷积神经网络的冬小麦麦穗检测计数系统[J]. 农业机械学报, 2019, 50(3): 144-150. doi: 10.6041/j.issn.1000-1298.2019.03.015 [21] 彭文, 兰玉彬, 岳学军, 等. 基于深度卷积神经网络的水稻田杂草识别研究[J]. 华南农业大学学报, 2020, 41(6): 75-81. [22] 黄小杭, 梁智豪, 何子俊, 等. 基于YOLOv2的莲蓬快速识别研究[J]. 现代农业科技, 2018(13): 164-167. [23] 彭红星, 黄博, 邵园园, 等. 自然环境下多类水果采摘目标识别的通用改进SSD模型[J]. 农业工程学报, 2018, 34(16): 155-162. doi: 10.11975/j.issn.1002-6819.2018.16.020 [24] 张洋. 基于深度学习的水稻种粒缺陷识别及重叠分布千粒重测量研究[D]. 扬州: 扬州大学, 2021. [25] 马志宏, 贡亮, 林可, 等. 基于稻穗几何形态模式识别的在穗籽粒数估测[J]. 上海交通大学学报, 2019, 53(2): 239-246. [26] WANG Q L, WU B G, ZHU P F, et al. ECA-Net: Efficient channel attention for deep convolutional neural networks[C]//2020 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Seattle, USA: IEEE, 2020: 11531-11539.
[27] HU J, SHEN L, ALBANIE S, et al. Squeeze-and-excitation networks[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2020, 42(8): 2011-2023. doi: 10.1109/TPAMI.2019.2913372
-
期刊类型引用(3)
1. 罗倩敏,韦丽,韩佃刚,叶玲玲,杨云庆,董俊,张冲,李静,尹尚莲,李瑶瑶,何昌霖,董仙兰,李丹丹,唐晓萍,艾军. 流行性出血病的全球流行及分布. 中国动物检疫. 2022(11): 96-104 .  百度学术
百度学术
2. 杨振兴,李占鸿,宋子昂,李卓然,李华春,朱建波,廖德芳,杨恒. 新血清型流行性出血病病毒荧光定量qRT-PCR与RT-PCR检测方法的建立及应用. 中国兽医科学. 2021(04): 446-454 .  百度学术
百度学术
3. 李卓然,吴健敏,朱建波,王金萍,吕敏娜,肖雷,杨振兴,廖德芳,李华春,杨恒. 2013—2019年我国流行性出血病病毒血清1型毒株的分离与遗传特征分析. 南方农业学报. 2021(08): 2043-2052 .  百度学术
百度学术
其他类型引用(1)






 下载:
下载: