Regulatory mechanism of autophagy in formation of crop agronomic traits and potential application
-
摘要:
细胞自噬是真核生物在进化过程中高度保守的重要降解途径,通过将受损的蛋白或细胞器包裹到双层膜结构的自噬小泡后,进而转运至溶酶体(动物)或液泡(酵母和植物)中进行降解,最终完成细胞内容物的循环利用。随着自噬在动物和酵母中研究的不断深入,人们也越来越多地关注植物自噬,且相关研究正在从模式植物逐渐扩展到作物。为更好地了解自噬在作物产量、品质和抗逆性等方面的作用,本文综述了近年来作物自噬的研究进展,并对自噬在重要农艺性状形成过程中的调控机制进行了深入探讨,以期为进一步改良作物的农艺性状和提高农业生产效率等提供参考。
Abstract:Autophagy is a highly conserved and important degradation pathway in eukaryotes during evolution. Damaged proteins or organelles are wrapped into autophagic vesicles with bilayer membrane structure, they are then transported to lysosomes(animals) or vacuoles(yeast and plants) for degradation, and finally the recycling of cell contents is completed. With the in-depth study of autophagy in animals and yeast, people are paying more and more attention to plant autophagy, and the related research is gradually expanding from model plants to crops. To better understand the effects of autophagy in crop yield, quality and resistance, etc, we summarized the recent advances in autophagy in crop plants, and discussed the regulatory mechanism of autophagy in the formation of important agronomic traits in depth. This paper will provide references for further improving crop agronomic traits and agricultural production efficiency.
-
Keywords:
- Autophagy /
- Regulatory mechanism /
- Agronomic trait /
- Crop
-
自噬(Autophagy)一词来源于希腊文auto-(自我)和phagein(吞噬),从字面讲就是自我吞噬的意思。这一概念最早在1963年由比利时生物化学和细胞生物学家Christian De Duve在观察到细胞中的自噬现象时提出[1-3]。受试验手段限制,自噬初期的研究难以深入,相关的分子机制更是知之甚少,仅能通过形态学观察来描述细胞自噬。20世纪90年代初,日本遗传学家Yoshinori Ohsumi研究组利用酵母这一经典的模式生物,进行细胞自噬突变体的遗传筛选,最终鉴定到多个参与自噬的关键基因,这些基因被命名为APG(Autophagy)基因,2003年自噬基因统一命名为ATG(Autophagy-related gene)基因[4-5]。1997年,继第1个自噬基因APG1(植物中为ATG1)被克隆后,迄今已有43个ATG基因陆续被克隆和报道[6-7],从而将细胞自噬带入了分子水平研究的时代,极大地推动了自噬的生物学功能研究的深度和广度,尤其是关于自噬与各种重要疾病形成和发生之间的密切关系,自噬成为当前生命科学研究的前沿热点[8]。
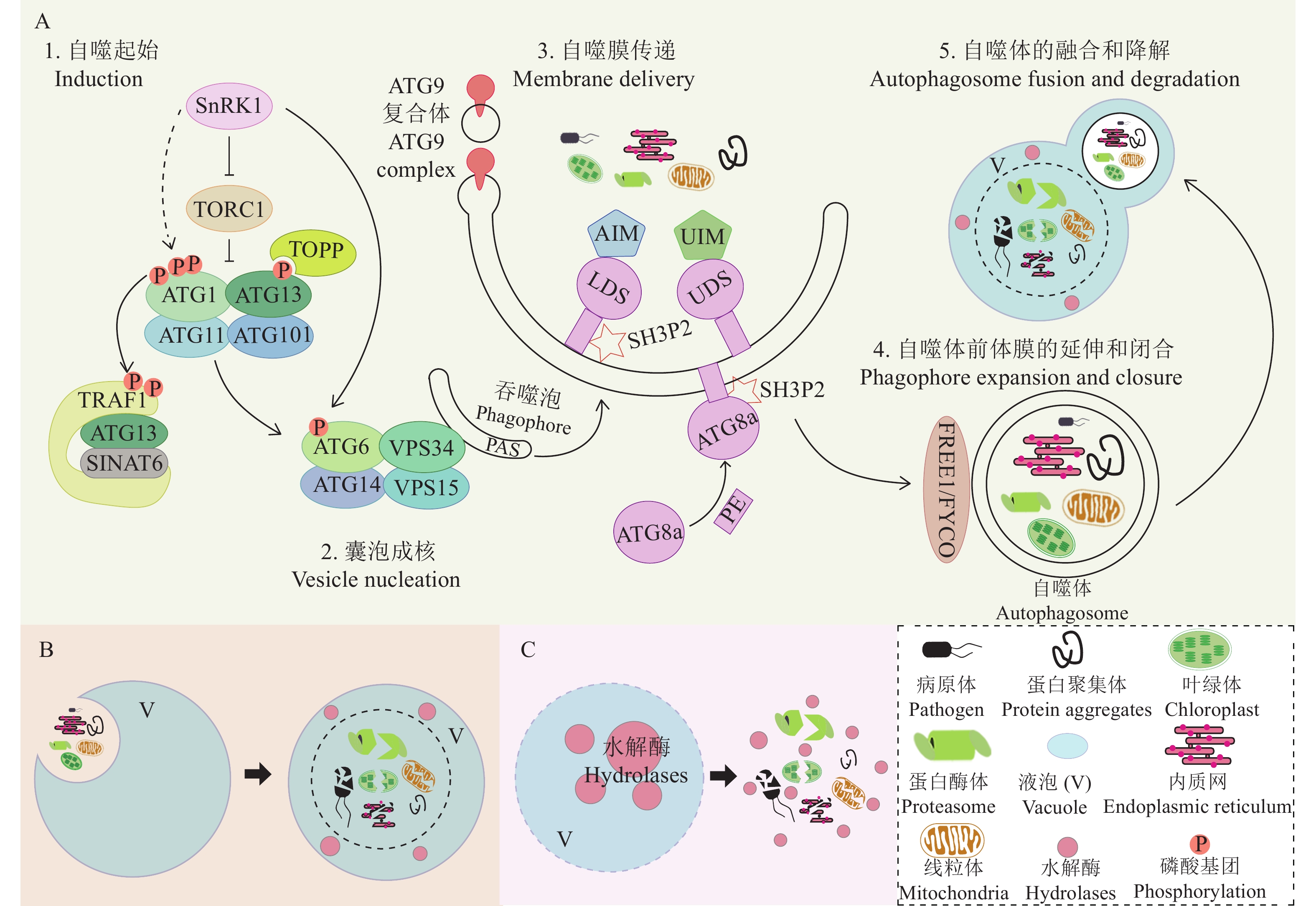
细胞自噬作为一种在进化中高度保守的细胞代谢过程,是真核细胞内清除衰老或受损细胞器、蛋白与蛋白聚集体的主要途径,在维持细胞内稳态和细胞组分再利用方面发挥重要作用[9]。目前已鉴定3种具有差异性的自噬类型,动物和酵母与植物中的类型略有不同[10]。在动物和酵母中,细胞自噬分为微自噬(Micro-autophagy)、巨自噬(Macro-autophagy)和分子伴侣介导的自噬(Chaperone-mediated autophagy, CMA);而在植物中,细胞自噬分为巨自噬、微自噬和超自噬(Mega-autophagy)[11-13](图1)。在巨自噬过程中,自噬体前体包裹吞噬物后形成具有双层膜结构的自噬体,然后自噬体与溶酶体(液泡)融合,自噬体内容物被溶酶体(液泡)内的水解酶降解[14-15](图1A)。在微自噬中,溶酶体膜(液泡膜)凹陷并包裹吞噬物,然后吞噬物在溶酶体(液泡)腔内被降解[16](图1B)。超自噬则是指通过液泡膜渗透或者破裂而实现的一种极端自噬过程,通常发生在植物细胞程序性死亡(Programmed cell death, PCD)的最终阶段[17]。在超自噬过程中,液泡膜通透性变差、破裂,直接向细胞质释放大量液泡水解酶,在细胞质中降解细胞质物质,甚至降解细胞壁,进而诱导PCD的发生[18](图1C)。分子伴侣介导的自噬即分子伴侣与胞质内蛋白质结合并转至溶酶体膜上,通过溶酶体跨膜蛋白把该蛋白质转至溶酶体腔内,再由溶酶体酶进行降解,此种自噬鲜见在植物中报道[19-21]。
近年来,随着研究手段和技术的不断进步,对细胞自噬尤其是巨自噬(也被称为选择性自噬)的认识更深入了。众多植物自噬相关的ATG基因被克隆,其在植物生长调节和胁迫响应中的功能也被逐一发掘。目前,植物自噬的研究已经从传统模式植物扩展到农作物。例如,模式植物拟南芥Arabidopsis thaliana的ATG基因的同源基因已在多个作物中被鉴定。越来越多的研究表明,这些ATG同源基因介导的细胞自噬参与了作物产量、品质和抗逆性的调控。为此,本文总结了植物巨自噬(以下简称自噬)的分子机制及其在作物重要农艺性状形成中的调控机理,并对其在作物遗传改良中的潜在应用进行了探讨。
1. 自噬一般过程和分子机制
目前已知的自噬途径主要包括:自噬起始、自噬前体形成、自噬前体膜延伸和闭合、自噬体转运与降解(图1A)。
1.1 自噬起始
ATG1/ATG13蛋白激酶复合体(包括ATG1、ATG11、ATG13和ATG101)启动自噬体的形成[22],受上游激酶TOR(Target of rapamycin)与SnRK1(Sucrose non-fermenting-1-related protein kinase 1)调控,响应上游调控因子对营养、能量信号的感知。
植物SnRK1与酵母SNF1(Sucrose non-fermenting 1)以及哺乳动物AMPK(AMP-activated kinase)具有较高的同源性,功能保守,作为能量感受器参与环境和细胞胁迫响应[23]。植物体内碳水化合物的水平往往随着发育状态和环境的变化而变化,植物通过感应碳水化合物的水平来调控SnRK1的激酶活性,同时,SnRK1能控制下游与能量相关的关键代谢酶类的酶活,反馈调控植物碳水化合物水平,进而控制植物生长发育以适应周围环境[24]。研究表明,拟南芥的SnRK1在TOR上游激活自噬[25],SnRK1还可能通过影响ATG1蛋白的磷酸化从而正向调控自噬[26]。在长期的饥饿条件下,SnRK1可以直接磷酸化PI3K复合体中的ATG6启动自噬[27]。
TOR是真核生物中高度保守的丝氨酸/苏氨酸蛋白激酶,能整合营养、能量、生长因子及环境信号,协调细胞增殖、生长和代谢等过程,是真核生物生长发育的核心调控因子[28]。TOR以复合体的形式行使功能。真核生物中已鉴定出2个结构和功能截然不同的蛋白复合体,分别为TORC1(TOR complex 1)和TORC2(TOR complex 2)[29]。与哺乳动物和酵母中存在2个TOR复合体不同,植物中只存在TORC1。TORC1主要由TOR、LST8(Lethal with sec thirteen protein 8)和RAPTOR(Regulatory associate protein of TOR)组成。LST8与TOR激酶结构域结合,帮助其选择性结合下游底物。RAPTOR主要维持TORC1二聚体(LST8与TOR形成的二聚体)的稳定性、催化能力和与底物结合的能力。ATG13蛋白结构域上存在一个TOS(TOR signaling)基序,该基序介导ATG13与RAPTOR结合[30-31]。在营养充足的条件下,TOR高度活跃,与其他激酶促进ATG13亚基的磷酸化并抑制ATG1亚基的自磷酸化,高度磷酸化的ATG13与低度磷酸化的ATG1之间的亲和力降低,导致两者之间的相互作用减弱,从而抑制自噬的启动[32];而在营养胁迫的条件下,TOR的活性被抑制,导致ATG13去磷酸化并与高度磷酸化的ATG1紧密结合,ATG1、ATG13、ATG11和ATG101形成紧密的激酶复合体,从而启动自噬。I型蛋白磷酸酶(Type one protein phosphatases, TOPPs)通过影响ATG13a的磷酸化状态调控复合体ATG1-ATG13的形成,超表达TOPP能促进ATG1a的磷酸化,该过程依赖于ATG13a[33]。
最新研究表明,拟南芥ATG1和ATG13的稳定性还受26S蛋白酶体调控。在正常条件下,E3泛素连接酶SINAT1/2(RING-type E3 ubiquitin transferase 1/2)与ATG13互作,促进ATG13降解从而抑制自噬。胁迫条件下,E3泛素连接酶SINAT6被诱导,与SINAT1/2竞争结合位点,其与ATG13结合,干扰SINAT1/2介导的ATG13的泛素化和降解,肿瘤坏死因子受体相关因子1(Tumor necrosis factor receptor-associated 1,TRAF1)作为该过程的适配器,可以与 ATG6、SINAT6 互作从而促进自噬的发生[34-35]。
1.2 自噬前体形成
ATG1/ATG13蛋白激酶复合体通过另外2种蛋白复合体促进自噬体前体形成:一种是磷脂酰肌醇−3−激酶(Phosphatidylinositol 3-kinase, PI3K)复合体,另一种是ATG9复合体。植物PI3K复合体为囊泡蛋白分选相关蛋白34(Vacuolar protein sorting 34, VPS34)形成的复合物,其中VPS34复合物Ⅰ(VPS34、VPS15、ATG6和ATG14)在巨自噬过程中参与成核作用;VPS34复合物Ⅱ(VPS34、VPS15、ATG6和VPS38)参与胞吞[36]。通过VPS34激酶活性,产生磷脂酰肌醇−3−磷酸(Phosphatidylinositol 3-phosphate, PI3P)以修饰自噬囊泡。VPS34复合物I早期被招募到起始膜上,以PI3P富集吞噬细胞膜,作为PI3P结合效应物招募的平台[37]。此外PI3P还能被FYCO1(FYVE and coiled-coil domain autophagy adaptor 1)蛋白中的 FYVE(Fab-1, YGL023,Vps27 and EEA1)结构域识别,在自噬囊泡转运上起着重要作用[38]。植物ATG9复合体包括ATG9、ATG2和ATG18。跨膜蛋白ATG9在ATG1-ATG13激酶复合体激活下,将脂质传递到自噬体前体膜上的组装位点,与膜蛋白ATG2-ATG18复合物互作,辅助自噬体前体膜的延伸[39-41]。
1.3 自噬前体膜的延伸和闭合
自噬前体膜的延伸和闭合需要2条类泛素化途径参与:一条是ATG8-PE结合途径,包括ATG8、ATG4、ATG7和ATG3,另一条则是ATG12-ATG5结合途径,包括ATG12、ATG7、ATG10、ATG5和ATG16。ATG8在与磷脂酰乙醇胺(Phosphatidylethanolamine, PE)结合后方可进入自噬小泡的膜层,ATG8-PE的形成先是半胱氨酸蛋白酶ATG4对ATG8的C端进行剪切,露出甘氨酸残基,接着在具有泛素活化酶E1活性的ATG7作用下,ATG7的半胱氨酸残基和ATG8的甘氨酸残基形成硫脂键,ATG8转移到具有泛素交联酶E2活性的ATG3上,最终与磷脂酰乙醇胺结合形成ATG8-PE复合体。ATG12-ATG5结合途径与ATG8-PE结合途径相似,先是ATG12与ATG7结合,后经具有泛素交联酶E2活性的ATG10转移到靶蛋白ATG5上,形成ATG12-ATG5复合体,该复合体与ATG16结合后形成二聚体。该复合物可以进一步促进ATG8与PE的酯化,并将其锚定在自噬体膜上[42-45]。ATG8与PE的结合是可逆的,可被ATG4切割,使外膜上的ATG8释放并循环利用,而自噬体内膜上的ATG8-PE复合物则在液泡中被降解[46]。大量的ATG8结合到自噬体前体上,促使自噬体前体逐渐扩张形成碗状结构,最后闭合形成完整的自噬体。此外,在拟南芥中存在一种非ATG蛋白SH3P2(SH3 domain-containing protein 2),该蛋白有助于调节自噬体的形成。在自噬诱导之前,SH3P2被招募到吞噬泡组装位点(Phagophore assembly site,PAS)并与PIP(Phosphatidylinositol phosphate)、PI3K复合体及ATG8结合,促进自噬体的形成[47]。另外一种内吞体分选转运复合体组分FREE1(FYVE domain protein required for endosomal sorting 1)蛋白和 SH3P2相互作用,并和PI3K复合体一起调控自噬体与液泡的融合以及自噬体的降解[48-49]。总之,ATG8-PE复合物不断组装在延伸中的自噬前体膜上,并通过招募SH3P2和FREE1来封闭囊泡。
1.4 自噬体转运和降解
最后,密封的ATG8和PI3P修饰的自噬体在FYVE和含有卷曲螺旋结构域(FYCO)蛋白的帮助下通过微管运送到液泡中。随后,SNARE(Soluble N-ethylmaleimide-sensitive factor attachment protein receptor)介导自噬体与液泡融合,该过程依赖于ARP2/3(Actin-related protein 2/3)、NAP1(NCK-associated protein)、ESCRT(Endosomal sorting complexes required for transport)复合物以及外囊蛋白EXO70B1的协助。最后,自噬体被运送到液泡中被液泡水解酶降解,消化后的产物再被液泡释放到细胞质供回收利用[50-51]。
2. 自噬受体识别及类型
巨自噬既可对细胞质内容物进行非选择性的整体降解,也可选择性地对一些可溶蛋白、受损的细胞器、蛋白聚集体和细菌等物质进行特异性降解[52]。这种选择性自噬主要是在脂化的ATG8插入自噬膜时发挥作用,内膜上的ATG8提供对接平台作用,通过与自噬受体(Receptor)和接头蛋白(Adaptor)互作来驱动自噬小泡进行货物选择。自噬受体通常包含一个以上的ATG8相互作用基序(ATG8-interacting motif, AIM/LIR)[53],该基序一般模式为W/F/YXXV/I/L,其中X代表任意氨基酸,可紧密地契合于ATG8上的2个保守疏水区,统称为LIR/AIM对接位点(LIR/AIM docking site, LDS)[54-55]。近年来又鉴定出一个新的保守位点:ATG8高亲和结合的泛素化相互作用基序(Ubiquitin-interacting motif, UIM),与之在ATG8上对应的是UIM对接位点(UIM-docking site, UDS)。一般来说,UIM是20个氨基酸的延伸,具有较大的序列多样性,可能会折叠成螺旋结构[56]。UIM序列为ψ-δ-X-A-ψ-X-X-S,其中ψ代表疏水残基,δ为亲水残基,X为任意氨基酸[57]。
随着研究的不断深入,人们发现植物细胞中存在着ATG8相互作用基序蛋白,其参与各种类型的选择性自噬,可以选择性地进行特定细胞器自噬[58]:病原体自噬(Xenophagy)、聚集体自噬(Aggrephagy)、蛋白酶体自噬(Proteaphagy)、线粒体自噬(Mitophagy)、叶绿体自噬(Chlorophagy)等[59-61]。
3. 自噬的作物生物学功能
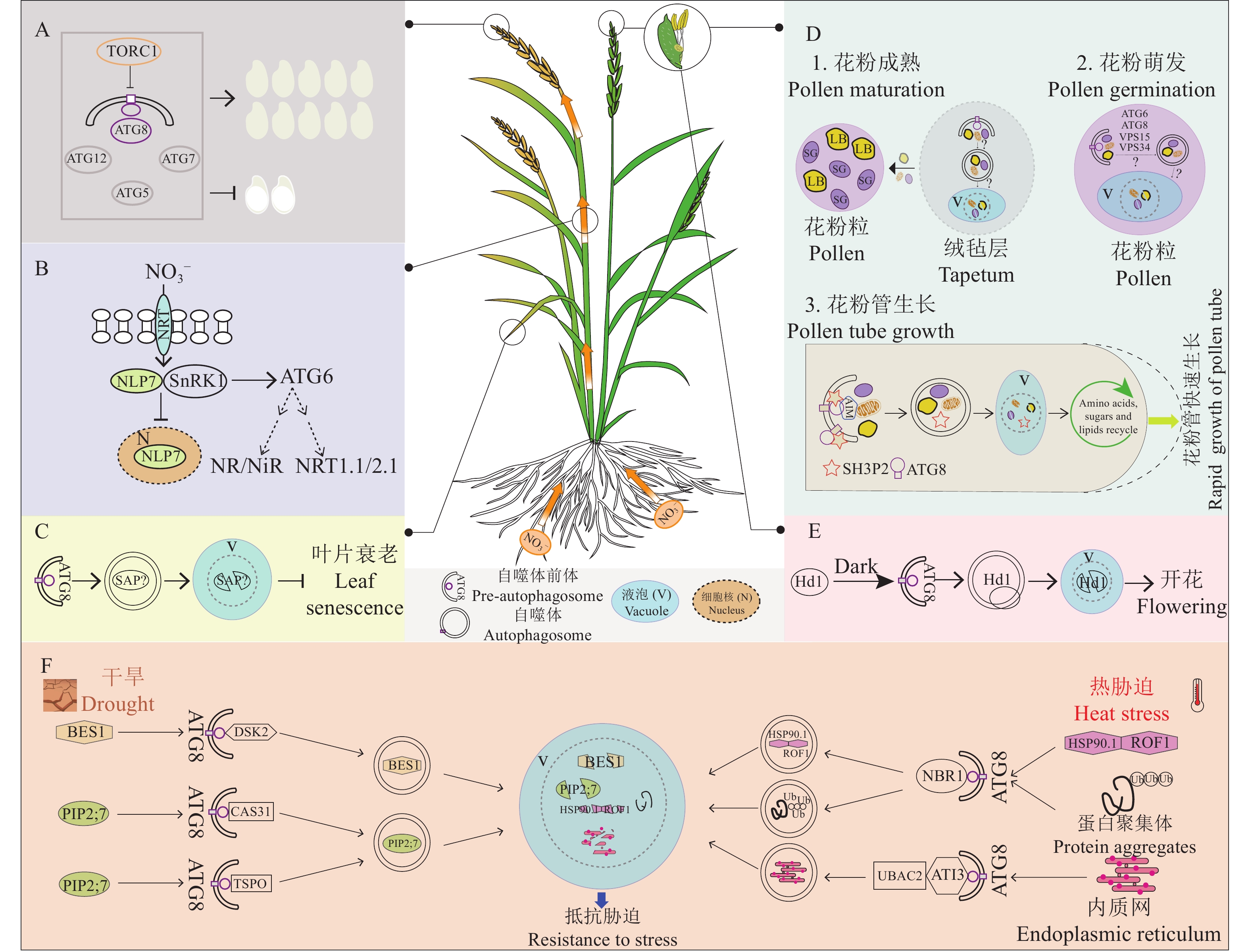
在正常生长条件下,植物细胞的各种发育活动,尤其是某些形态转变过程中,都需要自噬的参与。当环境因素改变时,植物将会大量诱导自噬以平衡生长和抗逆(图2)。
3.1 自噬对作物氮素利用率及低氮耐受性的影响
氮是植物必需营养元素,对作物的生物量和产量起着关键作用。植物对氮素的利用包括吸收、转运、同化和再利用。水稻Oryza sativa自噬突变体osatg7-1的株高、根长、分蘖数和总叶面积显著小于野生型,生物量小,氮素利用率低,氮的再动员受到抑制[62];玉米Zea mays自噬缺陷突变体zmatg12-1和zmatg12-2在低氮土壤条件下,生长受到更严重的胁迫,根系和地上部的生长明显下降,氮素从叶片到果实的分配受到限制[63];番茄Solanum lycopersicum自噬突变体slatg6、slatg10、slatg18a的生物量、氮积累量均较野生型减少[64],表明自噬缺陷突变体由于不能及时进行能量循环利用,造成新发育组织氮的可利用减少,从而导致植株整体叶面积减少,光合能力降低,氮回收能力削弱,从而在全株水平上影响了氮的高效再利用。
在低氮胁迫下,苹果Malus domestica中过表达MdATG8a可通过促进3种高亲和力硝酸盐转运蛋白(Nitrate transporter, NRT):NRT2.1/2.4/2.5表达量上调,进而提高转基因苹果对氮缺乏的耐受性[65]。过表达番茄SlATG6促进了低氮诱导的自噬体的形成,且NRT1.1、NRT2.1在根中的表达亦受到诱导,同时,硝酸还原酶(Nitrate reductase, NR)与亚硝酸还原酶 (Nitrite reductase, NiR)的活性亦提高,进而增加了番茄叶片的氮同化,最终促使根的氮含量以及生物量的同时提高[64]。这些发现表明在氮素缺乏的情况下,高水平的自噬不仅有利于植物氮素的循环利用,而且对植物氮素的吸收也具有正向调节作用。植物主要通过根系从土壤中吸收氮,该过程通过NRT完成,NRT1.1的蛋白水平受ABI2(Abscisic acid-insensitive 2)、CPSF30-L(Cleavage and polyadenylation specifcity factor 30-L)和FIP1[Factor interacting with poly(A) polymerase 1]负调控[66],暗示着自噬途径可能通过降解这些负调控因子从而提高NRT的水平,进而促进氮的高效利用。日后可关注这些负调控因子是否具有AIM序列,是否可与ATG8蛋白结合,进而被自噬选择性降解。最新研究表明SnRK1还能整合植物体内的代谢信号、内源激素以及外源环境刺激来调控硝态氮信号转导的关键转录因子NLP7(NODULE INCEPTION-like protein 7)活性;在低氮情况下,SnRK1与NLP7相互作用,然后磷酸化NLP7使其滞留在细胞质降解,从而阻止NLP7进入细胞核内,进而抑制植物硝态氮信号转导[67],NLP的具体降解途径未知。由于SnRK1是植物自噬的上游调控因子,而NLP7的降解依赖于SnRK1,推测NLP7可能是自噬调控氮响应通路的靶标蛋白之一,而NLP7是否通过自噬途径降解还需进一步研究。
氮收获指数(Nitrogen harvest index, NHI)指收获时经济器官中氮素占整株植物中氮素的比例,NHI越高表明植株积累的氮较多地分配到籽粒,籽粒蛋白质含量和积累量越多,营养品质越佳,种子萌发成苗和生长更好[68]。在低氮条件下,过表达OsATG8b可显著提高NHI,过表达SiATG8a亦可显著促进植株长势[69];过表达GmATG8c能够提高大豆Glycine max及其转基因愈伤组织的低氮耐受性[70];过表达MdATG9、MdATG10、MdATG18a均能提高苹果低氮耐受性,同时氮转运相关基因MdNRT1.1、MdNRT2.5、MdNIA1和MdNIA2的表达量均升高,保持较高的光合作用能力,获得较好的生长[65]。综上表明,过表达自噬相关基因可增强作物低氮耐受性,提高产量,提高籽粒品质,因此,自噬在未来的生物育种中具有重要的开发潜力。
3.2 自噬在延缓叶片衰老中的作用
自噬相关学术论文期刊的封面经常出现东西方古老文明中的图腾形象——衔尾蛇。衔尾蛇在古希腊语中有“自我吞噬”的含义,除此之外,衔尾蛇还象征着更新、重生和无限,与细胞自噬的生理功能不谋而合。人体细胞的自噬能力随着年龄的增加而降低,而细胞自噬能力越强,细胞的自我调节能力越好,但若是细胞自噬能力减弱,细胞就可能会出现不稳定,加快人的衰老速度,跟多种疾病还可能有一定的联系,植物同理。已有研究表明,水稻atg7突变体会出现明显的叶片早衰现象[62],大豆GmATG2沉默植株特异性呈现加速衰老表型,并且活性氧和水杨酸的积累显著增加,致病相关基因1(Pathogenesis-related gene 1, PR1)的表达也显著被诱导[71]。拟南芥自噬突变体atg2、atg4a/4b、atg5、atg7、atg9、atg10和atg18a也出现早衰现象,衰老基因SAG12(Senescence-associated gene 12)转录被大量诱导[72],说明植物自噬参与了植物衰老的过程。
有趣的是,TOR和SnRK激酶调控自噬的机制是通过调节转录因子活性发生的。因此,这些通路的下游靶点极有可能是自噬的直接转录调控因子。SnRK1通过磷酸化激活bZIP(Basic leucine zipper)转录因子,例如bZIP63,在植物中启动低能量反应[73]。其次,SnRK1.1可以与NAC转录因子ATAF1相互作用,过表达后导致早期衰老[74]。相反,TOR激活BZR1(Brassinazole resistant 1),BZR1通过抑制多个NAC转录因子来抑制衰老的起始[75]。当TOR不稳定时,BZR1通过自噬被降解[76]。综上所述,可以预见,作用于TOR和SnRK激酶下游的转录因子是植物自噬的直接转录调控因子,自噬可能参与降解叶片衰老相关蛋白(Senescence associated protein,SAP)从而延缓衰老。
植物的衰老主要表现为绿色光合器官的衰老,特别是细胞器叶绿体的优先降解,叶绿体及其内含物的降解途径与自噬相关:主要包括叶绿体组分的自噬降解以及整个叶绿体的自噬降解。核酮糖−1,5−二磷酸羧化酶/加氧酶(Ribulose-1,5-bisphosphate carboxylase/oxygenase, Rubisco)是光合作用中决定碳同化速率的关键酶,同时也是植物光呼吸的关键酶。在叶片衰老的小麦Triticum aestivum叶片中,存在包含叶绿体基质蛋白Rubisco但不包含类囊体蛋白质的小泡,被称为包含Rubisco小体(Rubisco-containing bodies, RCBs)[77]。RCBs在敲除ATG5或ATG7的突变体中不产生,并且RCBs与自噬体标记物GFP-ATG8共定位,这些结果表明RCBs是一种自噬体,它将部分基质蛋白传递到液泡中[78]。ATI1能与叶绿体蛋白质和自噬结构的核心蛋白ATG8互作介导特异质体蛋白的选择性自噬,通过形成ATI1-PS(ATG8-interacting protein 1 plastid bodies)将靶蛋白转运至液泡中降解[79]。整个受损的叶绿体也被证明可直接被液泡包被吞噬,发生叶绿体微自噬降解[80-81]。
自噬延缓植物衰老与植物激素的调控有关。大豆生殖期衰老叶片中ATG8s、ATG4和ATG9的同源物表达量提高[82]。3个ATG基因BrATG5b、BrATG8e-1和BrATG8h-1在经济作物菜心Brassica campestris ssp.chinensis var.utilis采后叶片衰老过程中的表达显著上调,这些基因受促进衰老的植物激素茉莉酸甲酯和脱落酸诱导,而受延缓衰老的激素细胞分裂素6−苄基腺苷(6-benzylaminopurine, 6-BA)和赤霉素抑制[83]。通过阻断水杨酸(Salicylic acid, SA)的生物合成或信号传导可以缓解这些自噬突变体的叶片早衰现象,如过表达SA羟化酶NahG(Salicylate hydroxylase)可以完全抑制atg2和atg5的早衰表型,而施加SA类似物苯并噻二唑(Benzothiadiazole, BTH)后则会恢复这些突变体的早衰表型[84]。利用玉米优良杂交种Jidan27及其亲本Si-287(早衰)和Si-144(保持绿色)的后代,以及173个玉米自交系,鉴定出的2个控制玉米叶片衰老的新基因座及其候选基因Stg3(ZmATG18b)和Stg7(ZmGH3.8),它们分别被预测为自噬途径和生长素途径的成员。ZmATG18b表达量较低、ZmGH3.8表达量较高的Stg3Si-144/Stg7Si-144等位基因组合能够显著延缓叶片的衰老,增加穗质量;而ZmATG18b表达量较高、ZmGH3.8表达量较低的Stg3Si-287/Stg7Si-287组合则恰恰相反,表明自噬和生长素途径在叶片衰老上可能起着协同调控作用[85]。植物激素与细胞自噬的具体互作机理尚未清楚,我们推测自噬可能通过降解植物激素代谢的负调控因子影响植物的衰老过程。
对于作物生产而言,叶片衰老最终影响作物产量与品质性状的形成。早衰的水稻、小麦和棉花Gossypium hirsutum品种会导致20%~30%的产量损失,而适度晚衰的玉米和小麦品种可以增产20%~30%。理论上,作物叶片衰老启动得愈晚光合总量愈大,衰老进程速率愈快,营养物质被动员和再利用的效率就愈高,对作物产量和品质性状的形成就愈有利,反之则不利[86]。适当的延缓叶片衰老,使作物产生合适的持绿现象是有必要的,过表达AtATG5和AtATG7的叶片衰老发生时间较野生型显著延迟4~7 d,平均寿命比野生型植株延长10%~20%[87]。因此,未来的生物育种可通过对自噬相关基因表达量的精细微调,从而达到适度延缓作物衰老,进而提高产量的目的。
3.3 自噬在调控作物抽穗开花中的作用
水稻抽穗期的重要农艺性状由主效和微效多基因控制,属于数量性状,它间接或直接地影响产量[88]。目前已知的水稻开花调控网络主要有3条:1)以OsGI-Hd1-Hd3a(Rice GIGANTEA、Heading date1和Heading date 3a)为核心进化保守的信号通路[89]。2)水稻特有的以Ghd7/Hd4-Ehd1-Hd3a/RFT1(Grain number、plant height and heading date、Early heading date 1 、Heading date 3a和Rice flowering locus T1)为核心的长日照条件下抑制开花途径[90-92]。3)独立于Hd1和Ehd1路径之外的其他开花调控基因:如DTH2、Se14、OsCO3和OsDOF12等[93-95]。
Hd1是第1个被报道的水稻开花基因,参与光周期诱导的抽穗期调控。Hd1扮演双元角色,在短日照下正调控抽穗期,在长日照下负调控抽穗期[96]。研究发现,osatg5-1突变体的开花时间比野生型晚25 d,自噬的其他关键基因OsATG7和OsATG8的缺失也导致类似的晚花表型,表明自噬途径调节水稻开花。在昼夜转化过程中自噬被激活,自噬核心蛋白ATG8通过相互作用基序AIM1识别细胞核定位的Hd1,进而将其运输到液泡中,完成Hd1蛋白的降解;当自噬功能丧失时,Hd1蛋白显著积累,抑制成花素基因表达,延迟水稻开花[97]。调控水稻开花的另外2条途径是否也与自噬有关系呢?Ehd1蛋白是否含有AIM识别序列?是否也作为自噬的靶蛋白从而影响水稻抽穗开花?将来可对Ehd1的降解途径进行研究。
3.4 自噬对作物生殖发育的影响
植物的生殖生长包括花发育、雌雄配子发育、传粉受精等生物过程。自噬在花粉成熟、自交不亲和性响应、花粉萌发、花粉管伸长和受精等过程中均扮演着重要角色。以下主要讨论自噬在花粉成熟、花粉萌发以及花粉管伸长发育过程中的影响。
花药是被子植物重要的雄性生殖器官,花药壁由外到内分别为表皮层、药室内壁、中层和绒毡层[98]。水稻花药减数分裂后期,绒毡层细胞中积累了双膜类自噬体结构与液泡包围的脂质体;而atg7突变体的绒毡层细胞中没有自噬体,但积累了大量脂质体。花粉成熟过程中,花药绒毡层逐渐消失,但atg7突变体还留有部分绒毡层,花药不开裂,而其花粉里只有少量脂质体与淀粉粒,并且其三酰基甘油与甘油二酯含量均低于野生型,花粉萌发活性降低,导致雄性不育,表明自噬可能通过调控花药绒毡层里脂质与其他营养物质的降解以及绒毡层自身的降解而影响营养物质向花粉传递,调控脂质与淀粉的代谢过程,促进花粉脂质等营养物质的积累,促进生殖发育[99],但具体的调控机制还有待研究。
在拟南芥中,AtATG6主要在成熟花粉中表达,atatg6的突变体抑制了花粉的萌发,进而导致雄性不育[100-101]。囊泡分选相关蛋白VPS34是植物中鉴定出的第1个PI3K囊泡分选蛋白,VPS34缺失突变体的花粉不能正常萌发[102]。此外,atvsp15突变体中花粉萌发也发生异常[103]。
磷脂酰肌醇依赖的磷脂酶C(Phosphoinositide-specific phospholipase C, PI-PLC)途径是花粉管极性生长中的信号转导调控途径之一。花粉管通过降解胞内储存的淀粉、脂质和蛋白质等物质可持续获得细胞生长所需的物质与能量。自噬在拟南芥花粉管生长和雄性育性过程中发挥重要生物学功能。在花粉管中,线粒体自噬与多种形式的自噬共同存在并维系花粉管的快速生长,在拟南芥雄配子体受精过程中发挥重要的调控功能。去极化的线粒体通过AIM对接位点特异性重组并与ATG8结合,实现线粒体自噬,但含有AIM的线粒体蛋白有待进一步研究。此外,自噬还可参与花粉管生长过程中破碎膜的去除和细胞质的缺失。在众多自噬调控因子和效应因子中,SH3P2(含有SH3区域蛋白)和ATG8可能是调控花粉萌发以及花粉管生长过程中自噬体生物发生和功能的关键分子[104]。在作物中可能也有相似调控模式,有待研究。
3.5 自噬对作物产量的影响
自噬影响着植物生长的方方面面,最终协同调控产量性状。水稻osatg8b沉默植株穗粒数和单株产量降低[105-106],玉米自噬缺陷突变体zmatg12-1和zmatg12-2种子产量较对照相比分别显著下降28%和25%,千粒质量也显著下降25%和21%[63]。拟南芥atatg5和atatg7突变体植株,每株莲座鲜质量和成熟种子总质量均降低了50%[87]。在番茄中鉴定到一个与拟南芥高度同源的TOR基因,相似度为86.53%,一致率为79.36%,编码的2470个氨基酸具有保守的FAT、FRB、激酶域和FATC结构域。与拟南芥不同的是,番茄tor突变体没有出现胚胎致死的表型,tor突变体自噬体的数目高于野生型。此外,tor突变体提高了饱和光强下的CO2同化速率、坐果率、单株结果数和果实产量[107],表明自噬过程在作物提高产量上起着不可或缺的作用。
自噬在转录水平上受到激素的调控,而植物激素的相互作用影响着植物发育的各个过程,从而影响产量性状[108-109]。乙烯是一种催熟剂,其在水稻结实过程中所起的作用与籽粒终止灌浆及成熟有关,籽粒中乙烯的活性高峰一般出现在灌浆末期,在作物产量上发挥重要作用。乙烯信号转录因子(Ethylene response factor 5, ERF5)通过与番茄SlATG8d和SlATG18h的启动子结合,进而促进自噬活性[110]。生长素含量与其灌浆过程密切相关,籽粒中生长素主要在水稻灌浆前期起作用,并在很大程度上决定着水稻的维管发育状况、叶片光合能力及籽粒灌浆速率,与水稻产量形成关系密切。生长素可以通过促进TOR活性来抑制细胞自噬[111]。自噬也有可能会通过降解植物激素通路的相关酶或蛋白反作用于影响植物激素信号通路,从而影响作物产量,具体的调控模式有待进一步研究。
在多种植物中过表达ATGs均表现出有利于植株整体生长发育的表型。过表达OsATG8a、OsATG8c株系从分蘖期到成熟期的分蘖数均显著多于对照组,植株的健壮性和旺盛性也均高于对照,单株产量也提高[105-106]。与对照相比,OsATG8b过表达植株穗粒数和单株产量增加,这些数据表明OsATG8b可能参与水稻的发育和影响产量。同时OsATG8b过表达收获指数(Harvest index, HI)显著高于对照,说明自噬在灌浆期起着重要作用[112]。GmATG8c过表达的转基因大豆较受体大豆‘天隆一号’非转基因对照的营养生长长势更加旺盛,其侧枝生长速度明显大于非转基因对照,二级分枝数也显著高于对照。对转基因大豆和非转基因对照的产量性状分析结果显示,转基因大豆的单株总荚数、单株粒数、单株粒质量和百粒质量等指标均显著高于非转基因对照[70, 113]。AtATG5和AtATG7过表达的植株比各自的敲除突变体和野生型植株发育出更多、更高的花序,开花时间更长,每个植株产生更多的种子,产量增加[87]。综上所述,自噬过程促进作物生长发育、增加产量。不仅如此,异源表达ATG8也能提高作物产量。在拟南芥中异源表达水稻OsATG8a、OsATG8b基因,与对照组相比具有明显的生长优势,莲座叶大小和株高显著增加,抽薹开花提前,转基因拟南芥的总果荚数、千粒质量和单株总种子质量显著增加[114-115],异源过表达大豆GmATG8c可以增强转基因拟南芥的单株总荚数和产量,其产量较野生型最高提高了22.2%。未来分子育种可考虑通过过表达ATG8基因从而达到高产的目的。
3.6 自噬对作物品质的影响
细胞自噬对作物品质的影响在粮食作物水稻上的研究较多。垩白是指由于稻米胚乳中组织疏松而形成的白色不透明的部分,是衡量稻米外观品质的主要指标之一[116]。野生型淀粉颗粒在整个胚乳中紧密堆积,形状呈多边形,边缘尖锐,而水稻自噬突变体osatg8b植株的种子颖壳有棕色斑点,含有白垩质胚乳,垩白率高于对照,籽粒存在许多松散堆积的小淀粉粒,种子品质很差。相反,过表达OsATG8b籽粒胚乳淀粉粒较大且紧密[112]。水稻osatg7-1的籽粒也呈白色垩白外观,中心区淀粉颗粒的形状与野生型相似,排列紧密,而侧面的淀粉颗粒排列松散,淀粉颗粒表面经常可见几个小凹坑,籽粒内源淀粉含量较低,可溶性糖含量较高,胚乳中淀粉降解的典型产物麦芽糖也在osatg7-1突变体的垩白颗粒中积累,表明osatg7-1胚乳中淀粉降解的异常激活[117],表明自噬突变体淀粉颗粒呈白垩状的主要原因是淀粉颗粒形态异常所造成的。
水稻α−淀粉酶至少有8 个成员,分为3个亚家族:Amy1、Amy2和Amy3,在储备淀粉降解的第1步起作用。研究发现,AmyI-1(Amy1A)和AmyⅡ-4(Amy3D)在osatg7-1突变体的垩白颗粒中比在野生型表达更强烈。成熟阶段osatg7-1籽粒α−淀粉酶活性也高于野生型。α−淀粉酶如Amy1A和Amy3D的异位过表达,即使在正常情况下也会导致白垩粒发生[118]。Amy1A和Amy3D主要定位于稻米外层,而α−葡萄糖苷酶和AmyII-3主要定位于稻米内层,由于osatg7-1突变体胚乳侧壁区淀粉粒形态异常,在水稻种子成熟过程中,胚乳侧壁区α−淀粉酶的异常表达和激活可能发生在osatg7-1突变体上,说明自噬可能参与降解关键淀粉酶,从而调控淀粉和糖代谢,进而调控水稻品质[119]。
自噬除了影响稻米垩白外,过表达自噬相关基因ATG8b可显著提高稻米的可溶性蛋白含量,提高稻米品质[117]。稻米品质与根系形态指标呈显著或极显著正相关,水稻根系形态的生长发育状况与根系的养分吸收率呈显著的正相关关系,较好的根系形态有利于提高稻米品质[120],但具体自噬如何通过影响根系或者其他与之相关信号通路进而调控稻米品质还需进一步探究。
3.7 自噬在调控作物抗逆中的作用
植物生长在一个开放的系统中,会受到各种不利环境条件如高温、干旱、盐渍等的影响,进而造成细胞器、细胞壁和细胞膜结构的破坏,影响正常生理代谢。在这些过程中不断有异常蛋白的产生,如果这些异常蛋白没有及时被清理掉,那么它们累积到一定的水平会对细胞产生毒害,最终影响植物的正常生长发育甚至导致死亡[121]。
模式植物拟南芥atg5、atg7突变体对高温、干旱胁迫不耐受[122]。在番茄中,沉默ATG5、ATG7基因导致植株对高温胁迫更加敏感[123]。过表达自噬基因通常可增强植物的抗性:番茄异源表达MdATG5能够提高植株对高温干旱胁迫的抗性[124]、过表达MdATG18a可提高苹果植株在高温胁迫下的抗氧化系统活性并减少高温对光合系统的损伤,从而提高基础耐热性[125]。长期干旱胁迫下,过表达MdATG4a苹果植株的长势、气孔开张度、光合作用、叶绿素含量和叶片相对含水量显著优于野生型[126]。辣椒Capsicum annuum自噬相关基因CaATG8c沉默或者过量表达都降低植物对热胁迫和盐胁迫的抗性,热胁迫下基因沉默植株比对照植株有更高的丙二醛含量、更低的叶绿素含量,且对照植株叶片的SOD活性上升,而基因沉默植株的SOD活性下降,说明CaATG8c基因沉默使辣椒植株对热胁迫下产生的活性氧的清除能力下降,从而导致其耐热性降低[121]。植物自噬受体蛋白NBR1(Neighbor of BRCA1)在热胁迫过程中有积累,并且可以调控植物对热胁迫的记忆。在热胁迫恢复过程中,ROF1(Rotamase FKBP1)和休克蛋白(Heat shock protein)HSP90.1结合,经NBR1介导选择性自噬降解,从而清除植物热胁迫的记忆使其迅速恢复到正常生长状态[127]。同时在热胁迫下, 植物细胞中变形或者受损的蛋白所形成的泛素化蛋白聚集体(Ubiquitinated protein aggregates)也由NBR1介导的选择性自噬清除[128]。内质网通路蛋白UBAC2(Ubiquitin-associated domain-containing protein 2)在植物耐热性中发挥重要作用,通过与ATI3(ATG8-interacting 3)互作,进而选择性自噬降解特异的内质网组分,从而在热胁迫中发挥作用[129]。在干旱胁迫中,泛素受体蛋白DSK2(Dominant suppressor of KAR 2)作为自噬受体介导油菜素内酯(Brassinosteroid, BR)信号通路中关键转录因子BES1(BRI1-EMS-suppressor 1)的选择性降解[130]。苜蓿Medicago truncatula中,应答干旱胁迫的正向调控因子脱水素蛋白MtCAS31(Cold acclimation-specific 31)参与自噬降解途径[131],拟南芥TSPO(Tryptophan-rich sensory protein)也可与自噬受体水通道蛋白AtPIP2;7相互作用,并促进AtPIP2;7由质膜向液泡运输,调节细胞透水性以适应干旱胁迫[132]。高温及干旱是全球性气候变化的重要表征,不仅影响植物生长发育、种群分布,而且也是作物产量和品质下降的主要原因。自然界中的植物面对的通常是多种胁迫条件,比如高温、干旱往往同步或相继发生[133]。目前在研究自噬在逆境响应中的作用时, 一般局限于单一胁迫因子,因此今后可关注高温和干旱复合胁迫、交叉胁迫下的自噬功能研究。
4. 总结与展望
自噬途径影响作物农艺性状形成的方方面面,包括提高作物氮的利用效率、延缓叶片衰老、促进开花、调控生殖发育、提高作物抗逆性,最终协同调控作物的产量与品质。近年来,尽管作物自噬相关研究已取得一定进展,如自噬形成机制、货物受体(Cargo receptor)的证实以及选择性自噬的鉴定,但是大部分选择性自噬的靶标蛋白尚不清楚。自噬通过降解哪些靶标蛋白从而调控作物的生长发育仍是未来研究的重点。而基于这些靶标蛋白的分子设计和改造,有望改变自噬的靶向降解进而达到有利目的。例如,对正调控产量性状的靶蛋白的AIM/UIM位点进行编辑突变,促使其逃逸ATG8识别而介导自噬降解,从而延长(或增强)该蛋白的调控作用,最终达到促进产量提升的效应。此外,细胞自噬的核心进程在真核生物中具有高度保守性,但不同物种中自噬的功能和调控网络存在特异性。因此,即使许多ATGs在模式植物拟南芥中的生物学功能已被鉴定,但在作物生长发育过程中的具体功能还需要继续破译,方可开发其潜在的应用潜力用于农业生产。综上所述,针对以上问题的研究必将丰富和完善植物自噬的调控机理,并为农作物的产量和品质、产量和抗性、品质和抗性的协同改良提供更多的理论依据和新的研究思路。
-
-
[1] MIZUSHIMA N. A brief history of autophagy from cell biology to physiology and disease[J]. Nature Cell Biology, 2018, 20(5): 521-527. doi: 10.1038/s41556-018-0092-5
[2] WARBURTON D, BELLUSCI S. Normal lung development needs self-eating[J]. The Journal of Clinical Investigation, 2019, 129(7): 2658-2659. doi: 10.1172/JCI129442
[3] KLIONSKY D J. Autophagy revisited: A conversation with Christian de Duve[J]. Autophagy, 2008, 4(6): 740-743. doi: 10.4161/auto.6398
[4] TAKESHIGE K, BABA M, TSUBOI S, et al. Autophagy in yeast demonstrated with proteinase-deficient mutants and conditions for its induction[J]. The Journal of Cell Biology, 1992, 119(2): 301-311. doi: 10.1083/jcb.119.2.301
[5] KLIONSKY D J, CREGG J M, DUNN W A JR, et al. A unified nomenclature for yeast autophagy-related genes[J]. Developmental Cell, 2003, 5(4): 539-545. doi: 10.1016/S1534-5807(03)00296-X
[6] MATSUURA A, TSUKADA M, WADA Y, et al. Apg1p, a novel protein kinase required for the autophagic process in Saccharomyces cerevisiae[J]. Gene, 1997, 192(2): 245-250. doi: 10.1016/S0378-1119(97)00084-X
[7] FUKUDA T, EBI Y, SAIGUSA T, et al. Atg43 tethers isolation membranes to mitochondria to promote starvation-induced mitophagy in fission yeast[J]. eLife, 2020, 9: e61245. doi: 10.7554/eLife.61245
[8] 杨娇, 胡荣贵. 牺牲局部、成就整体的细胞自噬: 2016年度诺贝尔生理学或医学奖成果简介[J]. 科技导报, 2016, 34(24): 39-43. [9] 陈俊慧, 谷有全, 姚利和, 等. 分子伴侣介导的自噬在阿尔茨海默病中作用的研究进展[J]. 上海交通大学学报(医学版), 2021, 41(11): 1529-1534. [10] REHMAN N U, ZENG P, MO Z, et al. Conserved and diversified mechanism of autophagy between plants and animals upon various stresses[J]. Antioxidants, 2021, 10(11): 1736. doi: 10.3390/antiox10111736
[11] ZHANG Z, YANG X, SONG Y, et al. Autophagy in Alzheimer’s disease pathogenesis: Therapeutic potential and future perspectives[J]. Ageing Research Reviews, 2021, 72: 101464. doi: 10.1016/j.arr.2021.101464
[12] VAN DOORN W G, PAPINI A. Ultrastructure of autophagy in plant cells: A review[J]. Autophagy, 2013, 9(12): 1922-1936. doi: 10.4161/auto.26275
[13] BASSHAM D C, LAPORTE M, MARTY F, et al. Autophagy in development and stress responses of plants[J]. Autophagy, 2006, 2(1): 2-11. doi: 10.4161/auto.2092
[14] OKU M, SAKAI Y. Three distinct types of microautophagy based on membrane dynamics and molecular machineries[J]. Bioessays, 2018, 40(6): 1800008. doi: 10.1002/bies.201800008
[15] 蔡霞, 方晓艾, 田兰婷, 等. 显微镜技术在植物细胞自噬研究中的应用[J]. 电子显微学报, 2016, 35(2): 180-185. doi: 10.3969/j.issn.1000-6281.2016.02.013 [16] SIENKO K, POORMASSALEHGOO A, YAMADA K, et al. Microautophagy in plants: Consideration of its molecular mechanism[J]. Cells, 2020, 9(4): 887. doi: 10.3390/cells9040887
[17] KAUSHIK S, CUERVO A M. The coming of age of chaperone-mediated autophagy[J]. Nature Reviews Molecular Cell Biology, 2018, 19(6): 365-381. doi: 10.1038/s41580-018-0001-6
[18] BU F, YANG M, GUO X, et al. Multiple functions of ATG8 family proteins in plant autophagy[J]. Frontiers in Cell and Developmental Biology, 2020, 8: 466. doi: 10.3389/fcell.2020.00466
[19] BOURDENX M, MARTIN-SEGURA A, SCRIVO A, et al. Chaperone-mediated autophagy prevents collapse of the neuronal metastable proteome[J]. Cell, 2021, 184(10): 2696-2714. doi: 10.1016/j.cell.2021.03.048
[20] CABALLERO B, BOURDENX M, LUENGO E, et al. Acetylated tau inhibits chaperone-mediated autophagy and promotes tau pathology propagation in mice[J]. Nature Communications, 2021, 12(1): 2238. doi: 10.1038/s41467-021-22501-9
[21] VAN DOORN W G, WOLTERING E J. What about the role of autophagy in PCD?[J]. Trends in Plant Science, 2010, 15(7): 361-362. doi: 10.1016/j.tplants.2010.04.009
[22] SUTTANGKAKUL A, LI F, CHUNG T, et al. The ATG1/ATG13 protein kinase complex is both a regulator and a target of autophagic recycling in Arabidopsis[J]. The Plant Cell, 2011, 23(10): 3761-3779. doi: 10.1105/tpc.111.090993
[23] BAENA-GONZALEZ E, ROLLAND F, THEVELEIN J M, et al. A central integrator of transcription networks in plant stress and energy signalling[J]. Nature, 2007, 448(7156): 938-942. doi: 10.1038/nature06069
[24] HAN C, LIU Y, SHI W, et al. KIN10 promotes stomatal development through stabilization of the SPEECHLESS transcription factor[J]. Nature Communications, 2020, 11(1): 4214. doi: 10.1038/s41467-020-18048-w
[25] SOTO-BURGOS J, BASSHAM D C. SnRK1 activates autophagy via the TOR signaling pathway in Arabidopsis thaliana[J]. PLoS One, 2017, 12(8): e0182591. doi: 10.1371/journal.pone.0182591
[26] CHEN L, SU Z Z, HUANG L, et al. The AMP-activated protein kinase KIN10 is involved in the regulation of autophagy in Arabidopsis[J]. Frontiers in Plant Science, 2017, 8: 1201. doi: 10.3389/fpls.2017.01201
[27] HUANG X, ZHENG C, LIU F, et al. Genetic analyses of the Arabidopsis ATG1 kinase complex reveal both kinase-dependent and independent autophagic routes during fixed-carbon starvation[J]. The Plant Cell, 2019, 31(12): 2973-2995. doi: 10.1105/tpc.19.00066
[28] 孟彦彦, 张楠, 熊延. 植物TOR激酶响应上游信号的研究进展[J]. 植物学报, 2022, 57(1): 1-11. doi: 10.11983/CBB21183 [29] ALBERT V, HALL M N. mTOR signaling in cellular and organismal energetics[J]. Current Opinion in Cell Biology, 2015, 33: 55-66. doi: 10.1016/j.ceb.2014.12.001
[30] WANG Q, HOU S. The emerging roles of ATG1/ATG13 kinase complex in plants[J]. Journal of Plant Physiology, 2022, 271: 153653. doi: 10.1016/j.jplph.2022.153653
[31] SON O, KIM S, KIM D, et al. Involvement of TOR signaling motif in the regulation of plant autophagy[J]. Biochemical and Biophysical Research Communications, 2018, 501(3): 643-647. doi: 10.1016/j.bbrc.2018.05.027
[32] KRAFT C, KIJANSKA M, KALIE E, et al. Binding of the Atg1/ULK1 kinase to the ubiquitin-like protein Atg8 regulates autophagy[J]. The EMBO Journal, 2012, 31(18): 3691-3703. doi: 10.1038/emboj.2012.225
[33] WANG Q, QIN Q, SU M, et al. Type one protein phosphatase regulates fixed-carbon starvation-induced autophagy in Arabidopsis[J]. The Plant Cell, 2022: koac251. doi: 10.1093/plcell/koac251
[34] QI H, LI J, XIA F N, et al. Arabidopsis SINAT proteins control autophagy by mediating ubiquitylation and degradation of ATG13[J]. The Plant Cell, 2020, 32(1): 263-284. doi: 10.1105/tpc.19.00413
[35] QI H, XIA F N, XIE L J, et al. TRAF family proteins regulate autophagy dynamics by modulating AUTOPHAGY PROTEIN6 stability in Arabidopsis[J]. The Plant Cell, 2017, 29(4): 890-911. doi: 10.1105/tpc.17.00056
[36] BHATI K K, LUONG A M, BATOKO H. VPS34 complexes in plants: Untangled enough?[J]. Trends in Plant Science, 2021, 26(4): 303-305. doi: 10.1016/j.tplants.2021.02.001
[37] HURLEY J H, SCHULMAN B A. Atomistic autophagy: The structures of cellular self-digestion[J]. Cell, 2014, 157(2): 300-311. doi: 10.1016/j.cell.2014.01.070
[38] PANKIV S, ALEMU E A, BRECH A, et al. FYCO1 is a Rab7 effector that binds to LC3 and PI3P to mediate microtubule plus end-directed vesicle transport[J]. The Journal of Cell Biology, 2010, 188(2): 253-269. doi: 10.1083/jcb.200907015
[39] XIONG Y, CONTENTO A L, BASSHAM D C. AtATG18a is required for the formation of autophagosomes during nutrient stress and senescence in Arabidopsis thaliana[J]. The Plant Journal, 2005, 42(4): 535-546. doi: 10.1111/j.1365-313X.2005.02397.x
[40] ZHUANG X, CHUNG K P, CUI Y, et al. ATG9 regulates autophagosome progression from the endoplasmic reticulum in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(3): E426-E435.
[41] LAI L, YU C, WONG J, et al. Subnanometer resolution cryo-EM structure of Arabidopsis thaliana ATG9[J]. Autophagy, 2020, 16(3): 575-583. doi: 10.1080/15548627.2019.1639300
[42] 王燕, 刘玉乐. 植物细胞自噬研究进展[J]. 中国细胞生物学学报, 2010, 32(5): 677-689. [43] CHUNG T, PHILLIPS A R, VIERSTRA R D. ATG8 lipidation and ATG8-mediated autophagy in Arabidopsis require ATG12 expressed from the differentially controlled ATG12A and ATG12B loci[J]. The Plant Journal, 2010, 62(3): 483-493. doi: 10.1111/j.1365-313X.2010.04166.x
[44] HANADA T, NODA N N, SATOMI Y, et al. The Atg12-Atg5 conjugate has a novel E3-like activity for protein lipidation in autophagy[J]. The Journal of Biological Chemistry, 2007, 282(52): 37298-37302. doi: 10.1074/jbc.C700195200
[45] ROMANOV J, WALCZAK M, IBIRICU I, et al. Mechanism and functions of membrane binding by the Atg5-Atg12/Atg16 complex during autophagosome formation[J]. The EMBO Journal, 2012, 31(22): 4304-4317. doi: 10.1038/emboj.2012.278
[46] KIRISAKO T, ICHIMURA Y, OKADA H, et al. The reversible modification regulates the membrane-binding state of Apg8/Aut7 essential for autophagy and the cytoplasm to vacuole targeting pathway[J]. The Journal of Cell Biology, 2000, 151(2): 263-276. doi: 10.1083/jcb.151.2.263
[47] ZHUANG X, WANG H, LAM S K, et al. A BAR-domain protein SH3P2, which binds to phosphatidylinositol 3-phosphate and ATG8, regulates autophagosome formation in Arabidopsis[J]. The Plant Cell, 2013, 25(11): 4596-4615. doi: 10.1105/tpc.113.118307
[48] GAO C, ZHUANG X, CUI Y, et al. Dual roles of an Arabidopsis ESCRT component FREE1 in regulating vacuolar protein transport and autophagic degradation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(6): 1886-1891. doi: 10.1073/pnas.1421271112
[49] SUN S, FENG L, CHUNG K P, et al. Mechanistic insights into an atypical interaction between ATG8 and SH3P2 in Arabidopsis thaliana[J]. Autophagy, 2022, 18(6): 1350-1366. doi: 10.1080/15548627.2021.1976965
[50] SURPIN M, ZHENG H, MORITA M T, et al. The VTI family of SNARE proteins is necessary for plant viability and mediates different protein transport pathways[J]. The Plant Cell, 2003, 15(12): 2885-2899. doi: 10.1105/tpc.016121
[51] KATSIARIMPA A, KALINOWSKA K, ANZENBERGER F, et al. The deubiquitinating enzyme AMSH1 and the ESCRT-III subunit VPS2.1 are required for autophagic degradation in Arabidopsis[J]. The Plant Cell, 2013, 25(6): 2236-2252. doi: 10.1105/tpc.113.113399
[52] SVENNING S, LAMARK T, KRAUSE K, et al. Plant NBR1 is a selective autophagy substrate and a functional hybrid of the mammalian autophagic adapters NBR1 and p62/SQSTM1[J]. Autophagy, 2011, 7(9): 993-1010. doi: 10.4161/auto.7.9.16389
[53] XIE Q, TZFADIA O, LEVY M, et al. hfAIM: A reliable bioinformatics approach for in silico genome-wide identification of autophagy-associated Atg8-interacting motifs in various organisms[J]. Autophagy, 2016, 12(5): 876-887. doi: 10.1080/15548627.2016.1147668
[54] BEHRENDS C, SOWA M E, GYGI S P, et al. Network organization of the human autophagy system[J]. Nature, 2010, 466(7302): 68-76. doi: 10.1038/nature09204
[55] NODA N N, OHSUMI Y, INAGAKI F. Atg8-family interacting motif crucial for selective autophagy[J]. FEBS Letters, 2010, 584(7): 1379-1385. doi: 10.1016/j.febslet.2010.01.018
[56] HOFMANN K, FALQUET L. A ubiquitin-interacting motif conserved in components of the proteasomal and lysosomal protein degradation systems[J]. Trends in Biochemical Sciences, 2001, 26(6): 347-350. doi: 10.1016/S0968-0004(01)01835-7
[57] MARSHALL R S, HUA Z, MALI S, et al. ATG8-binding UIM proteins define a new class of autophagy adaptors and receptors[J]. Cell, 2019, 177(3): 766-781. doi: 10.1016/j.cell.2019.02.009
[58] FLOYD B E, MORRISS S C, MACINTOSH G C, et al. What to eat: Evidence for selective autophagy in plants[J]. Journal of Integrative Plant Biology, 2012, 54(11): 907-920.
[59] 曹嘉健, 周杰. 植物自噬的功能及其农业应用展望[J/OL]. [2022-08-25] 中国科学: 生命科学, 2022: 1-18. https://kns.cnki.net/kcms/detail/11.5840.Q.20220629.1007.002.html [60] 杨小龙, 李漾漾, 刘玉凤, 等. 植物细胞选择性自噬研究进展[J]. 园艺学报, 2017, 44(10): 2015-2028. [61] LIU W, LIU Z, MO Z, et al. ATG8-interacting motif: Evolution and function in selective autophagy of targeting biological processes[J]. Frontiers in Plant Science, 2021, 12: 783881. doi: 10.3389/fpls.2021.783881
[62] WADA S, HAYASHIDA Y, IZUMI M, et al. Autophagy supports biomass production and nitrogen use efficiency at the vegetative stage in rice[J]. Plant Physiology, 2015, 168(1): 60-73. doi: 10.1104/pp.15.00242
[63] LI F Q, CHUNG T, PENNINGTON J G, et al. Autophagic recycling plays a central role in maize nitrogen remobilization[J]. The Plant Cell, 2015, 27(5): 1389-1408. doi: 10.1105/tpc.15.00158
[64] CAO J J, ZHENG X L, XIE D L, et al. Autophagic pathway contributes to low-nitrogen tolerance by optimizing nitrogen uptake and utilization in tomato[J]. Horticulture Research, 2022, 9: uhac068. doi: 10.1093/hr/uhac068
[65] SUN X, JIA X, HUO L, et al. MdATG18a overexpression improves tolerance to nitrogen deficiency and regulates anthocyanin accumulation through increased autophagy in transgenic apple[J]. Plant Cell and Environment, 2018, 41(2): 469-480. doi: 10.1111/pce.13110
[66] MAGHIAOUI A, GOJON A, BACH L. NRT1.1-centered nitrate signaling in plants[J]. Journal of Experimental Botany, 2020, 71(20): 6226-6237. doi: 10.1093/jxb/eraa361
[67] WANG H, HAN C, WANG J G, et al. Regulatory functions of cellular energy sensor SnRK1 for nitrate signalling through NLP7 repression[J]. Nature Plants, 2021, 8(9): 1094-1107.
[68] 程建峰, 戴廷波, 曹卫星, 等. 不同氮收获指数水稻基因型的氮代谢特征[J]. 作物学报, 2007, 33(3): 497-502. doi: 10.3321/j.issn:0496-3490.2007.03.022 [69] 李微微. 谷子自噬相关基因SiATG8a调控植物低氮胁迫响应的功能分析[D]. 哈尔滨: 哈尔滨师范大学, 2017. [70] 王丹, 王宁宁. 大豆自噬关键基因GmATG8c在氮高效、高产转基因新品种培育中的应用研究[J]. 大豆科技, 2019(5): 44-47. doi: 10.3969/j.issn.1674-3547.2019.05.011 [71] HASHIMI S M, WU N N, RAN J, et al. Silencing autophagy-related gene 2 (ATG2) results in accelerated senescence and enhanced immunity in soybean[J]. International Journal of Molecular Sciences, 2021, 22(21): 11749. doi: 10.3390/ijms222111749
[72] WANG H, SCHIPPERS J. The role and regulation of autophagy and the proteasome during aging and senescence in plants[J]. Genes, 2019, 10(4): 267. doi: 10.3390/genes10040267
[73] MAIR A, PEDROTTI L, WURZINGER B, et al. SnRK1-triggered switch of bZIP63 dimerization mediates the low-energy response in plants[J]. eLife, 2015, 4: e05828. doi: 10.7554/eLife.05828
[74] KLEINOW T, HIMBERT S, KRENZ B, et al. NAC domain transcription factor ATAF1 interacts with SNF1-related kinases and silencing of its subfamily causes severe developmental defects in Arabidopsis[J]. Plant Science, 2009, 177(4): 360-370. doi: 10.1016/j.plantsci.2009.06.011
[75] LOZANO-DURAN R, MACHO A P, BOUTROT F, et al. The transcriptional regulator BZR1 mediates trade-off between plant innate immunity and growth[J]. eLife, 2013, 2 e00983.
[76] ZHANG Z Z, ZHU J Y, ROH J, et al. TOR signaling promotes accumulation of BZR1 to balance growth with carbon availability in Arabidopsis[J]. Current Biology, 2016, 26(14): 1854-1860. doi: 10.1016/j.cub.2016.05.005
[77] CHIBA A, ISHIDA H, NISHIZAWA N K, et al. Exclusion of ribulose-1, 5-bisphosphate carboxylase/oxygenase from chloroplasts by specific bodies in naturally senescing leaves of wheat[J]. Plant and Cell Physiology, 2003, 44(9): 914-921. doi: 10.1093/pcp/pcg118
[78] ISHIDA H, YOSHIMOTO K, IZUMI M, et al. Mobilization of rubisco and stroma-localized fluorescent proteins of chloroplasts to the vacuole by an ATG gene-dependent autophagic process[J]. Plant Physiology, 2008, 148(1): 142-155. doi: 10.1104/pp.108.122770
[79] MICHAELI S, HONIG A, LEVANONY H, et al. Arabidopsis ATG8-interacting protein 1 is involved in autophagy-dependent vesicular trafficking of plastid proteins to the vacuole[J]. The Plant Cell, 2014, 26(10): 4084-4101. doi: 10.1105/tpc.114.129999
[80] OTEGUI M S. Vacuolar degradation of chloroplast components: Autophagy and beyond[J]. Journal of Experimental Botany, 2018, 69(4): 741-750. doi: 10.1093/jxb/erx234
[81] XIE Q J, MICHAELI S, PELED-ZEHAVI H, et al. Chloroplast degradation: One organelle, multiple degradation pathways[J]. Trends in Plant Science, 2015, 20(5): 264-265. doi: 10.1016/j.tplants.2015.03.013
[82] HTWE N, YUASA T, ISHIBASHI Y, et al. Leaf senescence of soybean at reproductive stage is associated with induction of autophagy-related genes, GmATG8c, GmATG8i and GmATG4[J]. Plant Production Science, 2011, 14(2): 141-147. doi: 10.1626/pps.14.141
[83] ZENG Z X, WANG C M, ZHAO Y T, et al. Molecular characterization of leaf senescence-associated autophagy genes in postharvest Chinese flowering cabbage and identifying their transcriptional activator BrMYB108[J]. Postharvest Biology and Technology, 2022, 185: 111785. doi: 10.1016/j.postharvbio.2021.111785
[84] RIGAULT M, CITERNE S, MASCLAUX-DAUBRESSE C, et al. Salicylic acid is a key player of Arabidopsis autophagy mutant susceptibility to the necrotrophic bacterium Dickeya dadantii[J]. Scientific Reports, 2021, 11(1): 3624. doi: 10.1038/s41598-021-83067-6
[85] FENG X, LIU L, LI Z, et al. Potential interaction between autophagy and auxin during maize leaf senescence[J]. Journal of Experimental Botany, 2021, 72(10): 3554-3568. doi: 10.1093/jxb/erab094
[86] 蒯本科. 植物衰老关乎器官发育和作物产量与品质性状的形成[J]. 植物生理学报, 2014, 50(9): 1265-1266. doi: 10.13592/j.cnki.ppj.2014.1026 [87] MININA E A, MOSCHOU P N, VETUKURI R R, et al. Transcriptional stimulation of rate-limiting components of the autophagic pathway improves plant fitness[J]. Journal of Experimental Botany, 2018, 69(6): 1415-1432. doi: 10.1093/jxb/ery010
[88] 王凌志. 水稻产量构成因子及抽穗期QTL定位[D]. 贵阳: 贵州大学, 2021. [89] HAYAMA R, YOKOI S, TAMAKI S, et al. Adaptation of photoperiodic control pathways produces short-day flowering in rice[J]. Nature, 2003, 422(6933): 719-722. doi: 10.1038/nature01549
[90] DOI K, IZAWA T, FUSE T, et al. Ehd1, a B-type response regulator in rice, confers short-day promotion of flowering and controls FT-like gene expression independently of Hd1[J]. Genes and Development, 2004, 18(8): 926-936. doi: 10.1101/gad.1189604
[91] SHIM J S, JANG G. Environmental signal-dependent regulation of flowering time in rice[J]. International Journal of Molecular Sciences, 2020, 21(17): E6155. doi: 10.3390/ijms21176155
[92] ZHOU S, ZHU S, CUI S, et al. Transcriptional and post-transcriptional regulation of heading date in rice[J]. The New Phytologist, 2021, 230(3): 943-956. doi: 10.1111/nph.17158
[93] WU W, ZHENG X M, LU G, et al. Association of functional nucleotide polymorphisms at DTH2 with the northward expansion of rice cultivation in Asia[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(8): 2775-2780. doi: 10.1073/pnas.1213962110
[94] 张硕. RID1招募组蛋白修饰因子调控水稻成花转换的分子机理研究[D]. 武汉: 华中农业大学, 2021. [95] 王婧莹, 赵广欣, 邱冠凯, 等. 水稻抽穗期途径基因的磷酸化、泛素化研究进展[J]. 中国水稻科学, 2022, 36(3): 215-226. [96] YANO M, KATAYOSE Y, ASHIKARI M, et al. Hd1, a major photoperiod sensitivity quantitative trait locus in rice, is closely related to the Arabidopsis flowering time gene CONSTANS[J]. The Plant Cell, 2000, 12(12): 2473-2484. doi: 10.1105/tpc.12.12.2473
[97] HU Z, YANG Z, ZHANG Y, et al. Autophagy targets Hd1 for vacuolar degradation to regulate rice flowering[J]. Molecular Plant, 2022, 15(7): 1137-1156. doi: 10.1016/j.molp.2022.05.006
[98] 宋露, 唐佳琦, 田晓杰, 等. UDT1/TDR基因过表达对水稻育性的影响[J]. 土壤与作物, 2022, 11(2): 150-158. doi: 10.11689/j.issn.2095-2961.2022.02.004 [99] KURUSU T, KOYANO T, HANAMATA S, et al. OsATG7 is required for autophagy-dependent lipid metabolism in rice postmeiotic anther development[J]. Autophagy, 2014, 10(5): 878-888. doi: 10.4161/auto.28279
[100] FUJIKI Y, YOSHIMOTO K, OHSUMI Y. An Arabidopsis homolog of yeast ATG6/VPS30 is essential for pollen germination[J]. Plant Physiology, 2007, 143(3): 1132-1139. doi: 10.1104/pp.106.093864
[101] QIN G, MA Z, ZHANG L, et al. Arabidopsis AtBECLIN 1/AtAtg6/AtVps30 is essential for pollen germination and plant development[J]. Cell Research, 2007, 17(3): 249-263. doi: 10.1038/cr.2007.7
[102] LEE Y, KIM E S, CHOI Y, et al. The Arabidopsis phosphatidylinositol 3-kinase is important for pollen development[J]. Plant Physiology, 2008, 147(4): 1886-1897. doi: 10.1104/pp.108.121590
[103] XU N, GAO X Q, ZHAO X Y, et al. Arabidopsis AtVPS15 is essential for pollen development and germination through modulating phosphatidylinositol 3-phosphate formation[J]. Plant Molecular Biology, 2011, 77(3): 251-260. doi: 10.1007/s11103-011-9806-9
[104] YAN H, ZHUANG M, XU X, et al. Autophagy and its mediated mitochondrial quality control maintain pollen tube growth and male fertility in Arabidopsis[J]. Autophagy, 2022. doi: 10.1080/15548627.2022.2095838.
[105] YU J, ZHEN X, LI X, et al. Increased autophagy of rice can increase yield and nitrogen use efficiency (NUE)[J]. Frontiers in Plant Science, 2019, 10: 584. doi: 10.3389/fpls.2019.00584
[106] ZHEN X, LI X, YU J, et al. OsATG8c-mediated increased autophagy regulates the yield and nitrogen use efficiency in rice[J]. International Journal of Molecular Sciences, 2019, 20(19): 4956. doi: 10.3390/ijms20194956
[107] 王玉. 番茄生长发育和逆境响应中自噬的作用及其调控机制[D]. 杭州: 浙江大学, 2017. [108] GOU W, LI X, GUO S, et al. Autophagy in plant: A new orchestrator in the regulation of the phytohormones homeostasis[J]. International Journal of Molecular Sciences, 2019, 20(12): 2900. doi: 10.3390/ijms20122900
[109] LI X, WU P, LU Y, et al. Synergistic interaction of phytohormones in determining leaf angle in crops[J]. International Journal of Molecular Sciences, 2020, 21(14): 5052. doi: 10.3390/ijms21145052
[110] ZHU T, ZOU L, LI Y, et al. Mitochondrial alternative oxidase-dependent autophagy involved in ethylene-mediated drought tolerance in Solanum lycopersicum[J]. Plant Biotechnology Journal, 2018, 16(12): 2063-2076. doi: 10.1111/pbi.12939
[111] PU Y, LUO X, BASSHAM D C. TOR-dependent and -independent pathways regulate autophagy in Arabidopsis thaliana[J]. Frontiers in Plant Science, 2017, 8: 1204. doi: 10.3389/fpls.2017.01204
[112] FAN T, YANG W, ZENG X, et al. A rice autophagy gene OsATG8b is involved in nitrogen remobilization and control of grain quality[J]. Frontiers in Plant Science, 2020, 11: 588. doi: 10.3389/fpls.2020.00588
[113] XIA T, XIAO D, LIU D, et al. Heterologous expression of ATG8c from soybean confers tolerance to nitrogen deficiency and increases yield in Arabidopsis[J]. PLoS One, 2012, 7(5): e37217. doi: 10.1371/journal.pone.0037217
[114] 李鑫, 甄晓溪, 于金磊, 等. 过表达OsATG8a基因提高转基因拟南芥对低氮的耐受性和产量[J]. 植物生理学报, 2019, 55(1): 69-79. doi: 10.13592/j.cnki.ppj.2018.0309 [115] 甄晓溪, 刘浩然, 李鑫, 等. 异源过表达OsATG8b基因提高转基因拟南芥的氮/碳胁迫耐受性和产量[J]. 植物学报, 2019, 54(1): 23-36. doi: 10.11983/CBB18064 [116] 朱爱科. 水稻垩白粒率QTL qPGWC-1的遗传分析及定位[D]. 北京: 中国农业科学院, 2018. [117] FITZGERALD M A, MCCOUCH S R, HALL R D. Not just a grain of rice: The quest for quality[J]. Trends in Plant Science, 2009, 14(3): 133-139. doi: 10.1016/j.tplants.2008.12.004
[118] NAKATA M, FUKAMATSU Y, MIYASHITA T, et al. High temperature-induced expression of rice α-amylases in developing endosperm produces chalky grains[J]. Frontiers in Plant Science, 2017, 8: 2089. doi: 10.3389/fpls.2017.02089
[119] SERA Y, HANAMATA S, SAKAMOTO S, et al. Essential roles of autophagy in metabolic regulation in endosperm development during rice seed maturation[J]. Scientific Reports, 2019, 9(1): 18544. doi: 10.1038/s41598-019-54361-1
[120] YAN F J, SUN Y J, XU H, et al. Effects of wheat straw mulch application and nitrogen management on rice root growth, dry matter accumulation and rice quality in soils of different fertility[J]. Paddy and Water Environment, 2018, 16(3): 507-518. doi: 10.1007/s10333-018-0643-1
[121] 翟于菲. 辣椒CaATG8c基因抗逆功能研究与自噬相关基因的全基因组鉴定及表达分析[D]. 杨凌: 西北农林科技大学, 2016. [122] ZHOU J, WANG J, CHENG Y, et al. NBR1-mediated selective autophagy targets insoluble ubiquitinated protein aggregates in plant stress responses[J]. PLoS Genetics, 2013, 9(1): e1003196. doi: 10.1371/journal.pgen.1003196
[123] ZHOU J, WANG J, YU J Q, et al. Role and regulation of autophagy in heat stress responses of tomato plants[J]. Frontiers in Plant Science, 2014, 5: 174.
[124] 贾昕. 苹果自噬相关基因MdATG5-1和MdATG5-2在干旱、高温逆境中的功能分析[D]. 杨凌: 西北农林科技大学, 2018. [125] HUO L, SUN X, GUO Z, et al. MdATG18a overexpression improves basal thermotolerance in transgenic apple by decreasing damage to chloroplasts[J]. Horticulture Research, 2020, 7: 21. doi: 10.1038/s41438-020-0243-2
[126] 王琪. 自噬基因MdATG4a在苹果响应干旱和盐胁迫中的功能分析[D]. 杨凌: 西北农林科技大学, 2021. [127] THIRUMALAIKUMAR V P, GORKA M, SCHULZ K, et al. Selective autophagy regulates heat stress memory in Arabidopsis by NBR1-mediated targeting of HSP90 and ROF1[J]. Autophagy, 2021, 17(9): 2184-2199. doi: 10.1080/15548627.2020.1820778
[128] JUNG H, LEE H N, MARSHALL R S, et al. Arabidopsis cargo receptor NBR1 mediates selective autophagy of defective proteins[J]. Journal of Experimental Botany, 2020, 71(1): 73-89. doi: 10.1093/jxb/erz404
[129] ZHOU J, WANG Z, WANG X T, et al. Dicot-specific ATG8-interacting ATI3 proteins interact with conserved UBAC2 proteins and play critical roles in plant stress responses[J]. Autophagy, 2018, 14(3): 487-504. doi: 10.1080/15548627.2017.1422856
[130] NOLAN T M, BRENNAN B, YANG M R, et al. Selective autophagy of BES1 mediated by DSK2 balances plant growth and survival[J]. Developmental Cell, 2017, 41(1): 33-46. doi: 10.1016/j.devcel.2017.03.013
[131] LI X, LIU Q W, FENG H, et al. Dehydrin MtCAS31 promotes autophagic degradation under drought stress[J]. Autophagy, 2020, 16(5): 862-877. doi: 10.1080/15548627.2019.1643656
[132] HACHEZ C, VELJANOVSKI V, REINHARDT H, et al. The Arabidopsis abiotic stress-induced tspo-related protein reduces cell-surface expression of the aquaporin pip2;7 through protein-protein interactions and autophagic degradation[J]. The Plant Cell, 2014, 26(12): 4974-4990. doi: 10.1105/tpc.114.134080
[133] 林思琪, 刘沁松. 细胞自噬在植物高温及干旱胁迫响应中的作用机制[J]. 植物生理学报, 2021, 57(5): 1031-1038. -
期刊类型引用(0)
其他类型引用(2)



 下载:
下载: