Morphological characterization and genetic mapping of shrunken endosperm mutant sh2021 in maize
-
摘要:目的
分析玉米籽粒皱缩突变体的表型特征并进行籽粒相关基因的精细定位,为揭示该基因调控玉米籽粒发育的分子机制奠定基础。
方法以玉米自交系‘B73’种植过程中籽粒自发突变个体为材料,命名为shank2021(sh2021),对其形态学和细胞学特征进行观察;构建分离群体,通过混合群体分离分析法(Bulked segregant analysis,BSA)对基因进行初步定位,筛选交换单株进一步缩小定位区间,最后结合转录组测序及基因功能注释推测控制籽粒缺陷性状的候选基因。
结果与野生型相比,sh2021籽粒凹陷皱缩、颜色加深、籽粒排列不规则,且百粒质量降低。扫描电镜观察发现,与野生型相比,sh2021胚乳细胞和淀粉粒均显著变小,且淀粉粒大小不均匀。遗传分析结果表明,sh2021是由单个隐性基因突变所致。利用BSA分析方法将目的基因定位在3号染色体末端约13.25 Mb区域。进一步扩大分离群体筛选交换单株,将目的基因定位在标记ID5与ID9之间的529.60 kb范围。通过sh2021和野生型的转录组测序,结合基因功能注释结果,预测Zm00001d044119、Zm00001d044120、Zm00001d044122、Zm00001d044129、Zm00001d044142这5个基因可能是影响玉米籽粒发育的重要候选基因。
结论sh2021的发现与鉴定为进一步深入解析玉米籽粒发育的遗传机制提供了丰富的试验材料,同时也为后续相关基因的克隆和功能解析奠定了基础。
Abstract:ObjectiveTo analyze the phenotypic characteristic of maize shrunk endosperm mutant and fine mapping of related genes, and lay a foundation for further understanding molecular mechanism of maize kernel development.
MethodThe spontaneous shrunk endosperm mutant shank2021(sh2021) was isolated from maize inbred line B73. Morphological and cytological characteristics were observed. A segregating population was developed, and the bulked segregant analysis (BSA) was used for preliminary gene mapping. The recombinant plants were screened for further narrowing the mapping interval. Finally the candidate genes controlling grain defect traits were speculated by transcriptome sequencing combined with gene function annotation analysis.
ResultCompared with wild type, sh2021 displayed sunken and shrunken kernels, darker color, irregular grain arrangement, and lower 100-grain weight. The scanning electron microscope observation revealed that sh2021 had smaller endosperm cells and starch granules, and the starch granules were varied in size. The genetic analysis results indicated that sh2021 was caused by a single recessive gene mutation. The BSA indicated that the target gene was located on a 13.25 Mb fragment at the end of chromosome 3. By further expanding the segregating population and screening recombinant plants, the target gene was narrowed down to an interval of 529.60 kb between markers ID5 and ID9. Transcriptome sequencing and gene annotation of sh2021 and wild type indicated that Zm00001d044119, Zm00001d044120, Zm00001d044122, Zm00001d044129, and Zm00001d044142 mighted be candidate genes controlling maize kernel development.
ConclusionThe identification of sh2021 provides abundant experimental materials for the study of kernel development, and lays a foundation for further map-based cloning and functional analysis of sh2021.
-
Keywords:
- Maize /
- Shrunken endosperm mutant /
- Genetic analysis /
- Fine mapping
-
氟苯尼考(Florfenicol,FF)属于酰胺醇类动物专用广谱抗生素,广泛用于防治畜禽消化道和呼吸道感染性疾病[1],氟苯尼考经动物摄入后随排泄物进入环境中,造成潜在的环境生态风险[2-3]。呼秀智等[4]研究表明,氟苯尼考对土壤微生物的抑制作用随浓度增加而增强。硫酸铜(CuSO4)作为一种促生长、防腹泻的添加剂用于养殖业,并发挥细胞电子传递、氧化还原等重要生理作用[5],但动物摄入后随排泄物进入环境中的高含量Cu可影响土壤微生物丰度、群落结构及功能多样性,导致土壤生态结构破坏[6]。许多报道指出金属与抗生素联用后存在协同或拮抗作用,如:与单一处理相比,土霉素与铅的交互作用使土壤细菌蔗糖酶活性明显下降,表现为协同效应[7];Fe(II)通过与土霉素形成复合物,降低其生物利用度,从而降低水稻对土霉素的吸收,表现为拮抗作用[8]。二者联用效果与相关酶活性变化、是否形成复合物以及目标物对重金属耐受性差异等因素均相关。Wang等[9]报道,动物粪便中氟苯尼考和Cu残留不仅阻碍环境中细菌正常代谢,还可能增加细菌耐药基因(Antibiotic resistance genes,ARGs)传播到人类的风险。
土壤中固氮菌介导的生物固氮作用是生态系统中氮元素输入土壤的主要过程,是全球氮循环至关重要的一步,兽用抗生素和重金属残留可能对固氮菌产生潜在影响[10]。本文对优势固氮菌RpEC2071进行氟苯尼考联合Cu胁迫处理,研究胁迫条件下对固氮菌胞外多糖产生及生物膜形成的影响,并分析固氮酶结构基因、氮代谢调控基因以及生物膜基因mRNA表达水平,将为评估兽药及添加剂的生态风险提供依据。
1. 材料与方法
1.1 供试材料
固氮菌从华南农业大学广东生物防治教育部工程研究中心无抗生素污染的花生根部附近土壤中分离纯化后得到。
氟苯尼考原料药购自大连美伦生物技术有限公司,纯度99.5%; LB肉汤、LB琼脂均购自广州环凯微生物有限公司;硫酸铜、异丙醇、NaOH、浓硫酸、葡萄糖、无水乙醇等均购自广州普智生物仪器有限公司,纯度均为分析纯;革兰氏染色液购自广州翔博生物科技有限公司。
1.2 固氮菌分离鉴定与纯化
1.2.1 花生根围土中固氮菌的分离培养
称5.0 g花生根围土壤,加入50 mL无菌水振荡摇匀,取10 mL悬浊液进行梯度稀释后分别取10−3、10−4、10−5 g/mL土壤悬液0.1 mL均匀涂布于LB琼脂培养基,28 ℃培养5~7 d后进行纯化,将纯化菌株加入φ为10%的甘油水中,−20 ℃条件下保存。
1.2.2 固氮菌镜检与16S rDNA菌种鉴定
制备固氮菌革兰氏染色涂片,油镜(100×)观察染色后细菌形态。提取固氮菌DNA模板,采用细菌16S rDNA通用引物27F/1492R进行PCR扩增,产物经15 g/L琼脂糖凝胶电泳检测后,送样广州擎科生物技术有限公司进行DNA测序。
1.3 固氮菌生物膜形成能力测试
供试菌活化:取保存的固氮菌划线接种LB板,经28 ℃培养48 h后纯化2次备用。
生物膜生长情况观察:挑取固氮菌单菌落于5 mL改良LB肉汤,设置3个重复,28 ℃培养,记录0、12 、24 、36 、72 、120 、132 、144 、156 和168 h时的试管生物膜生长情况。
1.4 固氮菌生物膜形成变化
96孔板内设置4组处理:空白组(CK)、氟苯尼考组(FF)、铜组(Cu)及混合组(FF+Cu),各处理组组分添加量见表1,28 ℃条件下孵育120 、132 、144 、156和168 h进行采样。样品采用结晶紫法进行处理[11],每组3个重复,使用酶标仪测定孔中溶液D590 nm。另外,以未接种供试菌的改良肉汤作为阴性对照,以阴性对照D590 nm(Dc)的2倍作为界限值,根据D590 nm进行生物膜形成能力结果判断:生物膜形成力强(D590 nm>2Dc);生物膜形成力弱(Dc<D590 nm≤2Dc);没有生物膜形成(D590 nm≤Dc)。
表 1 96孔板中处理组各组分添加量Table 1. Addition of each component in a 96-well plate of treatment groupμL 组别 Group 改良肉汤 Improved broth 菌液 Bacterial solution 甲醇 Methanol 氟苯尼考1)Florfenicol CuSO42) 空白组 Blank(CK) 170 20 10 0 0 氟苯尼考组 Florfenicol(FF) 170 20 0 10 0 铜组 Cu(Cu) 160 20 10 0 10 混合组 Mixed(FF+Cu) 160 20 0 10 10 1) ρ (氟苯尼考) =0.8 mg/mL;2) ρ (CuSO4) = 4 mg/mL 1)ρ (Florfenicol) = 0.8 mg/mL; 2) ρ (CuSO4) = 4 mg/mL 1.5 固氮菌胁迫模型构建
设置4组处理:空白组(CK)、氟苯尼考组(FF)、铜组(Cu)及混合组(FF+Cu),各处理组组分添加量见表2,每组3个重复,于给药后0、12、24、36、72、120、132、144、156和168 h测定各组菌液的D600 nm,并采集后5个时间点的细菌沉淀和上清液于–80 ℃保存,用于后续测定固氮菌胞外多糖含量。
表 2 胁迫模型组各组分添加量Table 2. Addition of each component in stress model groupsmL 组别 Group 改良肉汤 Improved broth 菌液 Bacterial solution 甲醇 Methanol 氟苯尼考1)Florfenicol CuSO42) 空白组 Blank(CK) 40.1 0.1 0.1 0 0 氟苯尼考组 Florfenicol(FF) 40.1 0.1 0 0.1 0 铜组 Cu(Cu) 40.0 0.1 0.1 0 0.1 混合组 Mixed(FF+Cu) 40.0 0.1 0 0.1 0.1 1) ρ (氟苯尼考)=16 mg/mL;2) ρ (CuSO4)= 80 mg/mL 1) ρ (Florfenicol) = 16 mg/mL; 2) ρ (CuSO4) = 80 mg/mL 1.6 固氮菌胞外多糖含量测定
分别取“1.5”各样品上清液2 mL至离心管中,加入乙醇,4 ℃条件下静置24 h后离心,洗涤沉淀后60 ℃干燥至恒质量。将各组干燥样品溶于2 mL蒸馏水中,另以蒸馏水作为阴性对照,使用苯酚硫酸法及紫外分光光度计估算胞外多糖浓度[12]。
1.7 实时荧光定量PCR检测
采用TRIzol法[13]抽提固氮菌样品RNA后反转录合成cDNA。参考全基因组测序结果设计引物,引物序列见表3,引物由生工生物工程(上海)股份有限公司合成。以获得的cDNA为模板,16S rDNA为内参基因,采用Bio-Rad CFX96检测系统进行实时荧光定量PCR (Quantitative real-time PCR,qRT-PCR)试验,所得数据运用2−△△Ct法进行分析计算。
表 3 引物序列Table 3. Primer sequences基因名称 Gene name 引物序列(5′→3′) Primer sequence 基因名称 Gene name 引物序列(5′→3′) Primer sequence flaF F: GCGAGCGACAGGCGTTGA R: TGATTATCCGGCTGCTTGAGATC ntrX F: ACTTGTCGGTGCGTCACTTGC R: GATGGGCTTCTTCCAGTGCG fliL F: AAAACGAACAGGCAGAGGGC R: GGGAAACGGTGCGGACATAG glnK F: TGACCGTGACCGAAGTAAAGGG R: TGCCGTCGCCGATCTGCC flhA F: ACCACCAGTCATTTCCTTGCCC R: CCGCCGTCGGACCCTCAT nnrR F: GCTGGACGGATTGCTGACCC R: GCCACCCGACGCTCTACCTC fliQ F: ATCGTCGGTGTCGCCATCG R: CGTCATTTCCTGAACCTGCGTC nifH F: CGGATTATCGCAGTAGCAAACC R: TCTGTCGTCTCCATCGCTTCAC ntrY F: CGCTGACACCAAGTTCACGACG R: GCATCATGGAGTTCCAGATACCC Rp16S-1 F: AGATGCTCTACCTTGATGTCCCTG R: AGATGCGTTGCGCCACCT 1.8 试验数据处理
用Excel对数据进行初步整理,用SPSS 25.0软件进行数据分析和统计,GraphPad Prism 7.0软件绘图,各指标用One-way ANOVA进行显著性分析,结果采用平均数±SD表示,P <0.05为差异显著性标准。所有数据均为3次或3次以上试验的结果。
2. 结果与分析
2.1 革兰氏染色与菌种鉴定
2.1.1 革兰氏染色镜检
经革兰氏染色后,染色结果如图1,在100倍光学显微镜下镜检细菌呈红色、棒状,判定该菌为革兰阴性菌。
2.1.2 16S rDNA菌种鉴定
将分离得到的菌株序列上传至NCBI数据库进行比对,发现其与固氮菌的匹配度最高,将该菌命名为RpEC2071。绘制系统发育树(图2),发现固氮菌RpEC2071与根瘤菌NRCPB10相似度高达99%,进一步鉴定其属于根瘤菌。
2.2 RpEC2071生物膜形成能力表型分析
根据生物膜试管形成试验结果(图3),发现0~36 h未能明显观察到生物膜,表明分离菌株在前36 h生物膜形成能力弱;培养至72 h时开始出现较为明显的生物膜,且生物膜形成能力随培养时长增加而逐渐增强。
2.3 RpEC2071生物膜形成能力分析
采集4种胁迫条件下RpEC2071菌液,测定D600 nm得到生长曲线,如图4所示,4组模型均表现为前期生长较为缓慢,在120 h处生长速度明显加快,之后逐渐趋于稳定,最终呈现衰退现象,故选取后面5个时间点做进一步分析。
96孔板内生物膜形成能力定量测定结果如图5所示。在156 h时,FF+Cu组的生物膜形成能力最低,CK组的生物膜形成能力最强,是FF+Cu组的3.1倍;在168 h时,FF+Cu组生物膜形成能力最低,FF组生物膜形成能力最强,是FF+Cu组的3.1倍。进一步分析发现,FF组和CK组生物膜形成能力均在156 h时急剧增强,Cu组在168 h时急剧增强,而FF+Cu组变化稳定且生物膜形成能力最低。综上生物膜的形成能力:FF组 > Cu组 > CK组 > FF+Cu组。
2.4 胞外多糖含量测定
根据“2.3”生长曲线选择的5个时间点对胞外多糖含量进行定量测定,结果如图6所示。由图6可得,4个试验组在5个时间点的胞外多糖平均分泌量表现为:FF组 > CK组 > Cu组 > FF+Cu组。表明Cu的添加对固氮菌存在抑制作用,且氟苯尼考能促进Cu的抑制作用。
2.5 相关性分析
基于全基因组测序结果,定量测定固氮基因nifH、4个氮代谢调控基因ntrY、ntrX、glnK、nnrR及7个生物膜相关功能基因flaF、fliL、flhA、fliP、fliQ、fliR、flbT,并进行相关性分析。结果显示,flaF、fliL、flhA、fliQ这4种生物膜相关基因分别与固氮基因nifH、氮代谢调控基因ntrX呈现正相关(r=0.548~0.832,P<0.05);另外发现nifH基因的表达可能受到ntrY、ntrX、glnK、nnrR这4种调控因子的调控(r =0.590~0.827,P<0.01)。
2.6 基因表达差异分析
基因荧光定量表达差异分析比较发现,生物膜相关基因表达受Cu的影响不大,然而其中的fliQ基因在混合或者Cu单独施压作用下,与氟苯尼考单独施压作用下存在显著的差异(图7 D),但均与CK组无明显差异。而在氮代谢相关基因的分析中,发现氟苯尼考与Cu混合后显著促进了nnrR基因的表达(图8 D),表明Cu与氟苯尼考对该调控基因存在协同作用,然而它们对ntrX存在拮抗作用。
对固氮菌中重要固氮酶的代表基因nifH进行差异性分析,结果如图9所示。其中Cu的添加显著升高了nifH基因的表达,而Cu与氟苯尼考联用时又减弱该过程。
3. 讨论与结论
生物膜有助于细菌承受饥饿、干燥等恶劣环境条件,使其能够适应变化的条件,在其面对环境压力、免疫反应和抗生素时提供保护性环境[14]。本研究结果表明,4种不同处理对生物膜的影响不同,FF组生物膜形成能力最强,Cu组次之,说明在氟苯尼考和Cu单独加入时,均对固氮菌产生压力,且氟苯尼考对固氮菌的胁迫压力较大,使固氮菌在胁迫环境下形成生物膜以保护自身,与冯世文等[15]的研究结果相符,而在氟苯尼考和Cu混合添加时则会抑制固氮菌生物膜的形成,其原因本试验没有阐明,可以做进一步的深入研究。与此同时,结果显示随胁迫时间增加固氮菌生物膜形成能力也逐步增强,说明细菌在暴露于压力条件下会产生更多的生物膜。
在4种不同处理下,固氮菌胞外多糖的分泌量明显不同,氟苯尼考组最高,混合组最低,与生物膜测定结果基本吻合,亦与杜心恬等[16]的研究结果相符,说明胞外多糖是生物膜重要组成部分[17],并与生物膜形成紧密相关。进一步分析Cu组发现,由于胞外多糖形成是细菌的主要防御机制之一[18],固氮菌在Cu的单一胁迫下会对胞外多糖的合成产生轻微抑制作用,但不对生物膜的形成能力产生抑制作用,此现象可从另一个方面说明,胞外多糖的分泌会影响生物膜的形成,但不是其决定性因素,可能与载体表面性质及环境因素等因素相关。
对4种不同处理下不同类型基因的表达进行相关性分析,结果表明,生物膜与胞外多糖无明显的相关性,再次验证胞外多糖对生物膜的形成有影响作用,但不是其决定性因素这一观点。进一步分析发现固氮酶基因nifH的表达可能受到ntrY、ntrX、glnK、nnrR这4种调控因子的调控,并与flaF、fliL、flhA、fliQ这4种生物膜相关基因呈现正相关。此外,4种生物膜相关基因还与氮代谢调控基因ntrX呈现显著正相关,这与Li等[19]的研究结果相符。说明氮循环功能跟生物膜形成之间有明显的相关性,故评价氮循环的功能过程不仅应关注其遗传潜力,还应关注其生物膜的生理活性。
差异性分析中,与Ⅲ型分泌系统中鞭毛蛋白转运有关的生物膜相关基因fliQ[20],在氟苯尼考和Cu混合作用或者Cu单独作用下,与氟苯尼考单独作用存在显著的差异,说明氟苯尼考与Cu对该基因表达的影响差异较大,且对其表达存在明显拮抗作用。Cu2+可能使固氮菌鞭毛组成紊乱,进而削弱RpoN2因子功能,导致fliQ基因表达下降[21];而氟苯尼考对固氮菌的胁迫使得其鞭毛运动增强,进而使得fliQ基因表达上升。同理,二者对参与控制鞭毛和胞外多糖的ntrX基因的表达同样存在拮抗作用[22]。nifH是负责编码固氮酶铁蛋白的基因[23],Cu的添加显著提高了nifH的表达,而氟苯尼考会削弱这一过程,存在拮抗作用。Cu蓄积对固氮菌产生不利影响,脱毒过程会增加能源消耗和氮需求,从而可能通过其共生作用促进nifH表达;而氟苯尼考可能与Cu形成络合物,降低Cu浓度,故相较于Cu组,FF+Cu组nifH表达减弱。与之相反,分析氮代谢相关基因发现,氟苯尼考与Cu混合后显著促进了反硝化作用中负责催化NO转化为N2O的反硝化酶的调控基因nnrR的表达,表明Cu与氟苯尼考对该调控基因存在协同作用,可能是由于在氟苯尼考联合Cu的双重胁迫下迫使固氮菌需要更加快速地进行反硝化作用以保护自身,进而使得nnrR的表达增加[24-25]。
本文通过从花生根围土中分离出一株优势固氮菌,以养殖业中被广泛使用的广谱类抗菌药物氟苯尼考和饲料添加剂Cu作为胁迫因子,分别从表型水平和基因水平探究二者单独或联合处理对优势固氮菌产生的影响以及生物固氮主要功能基因分子响应特征。发现二者单独使用均会使固氮菌生物膜形成能力增强,但联用时产生拮抗作用;二者对胞外多糖形成的影响与生物膜测定结果基本一致;二者单独或联用均改变相关功能基因表达水平,且对fliQ、nifH基因产生拮抗作用、对nnrR基因产生协同作用。氟苯尼考及Cu均影响着固氮菌生物膜的形成,说明二者的存在威胁着土壤中固氮菌的生存,长期以往将破坏环境土壤生态系统。
-
图 1 sh2021突变体表型鉴定(F2分离群体)
A:完熟期玉米果穗;B:籽粒表型;C:籽粒百粒质量;“**”表示sh201与野生型在0.01水平差异显著(t检验)
Figure 1. Phenotypic identification of sh2021 mutants (F2 segregating population)
A: Mature ear; B: Grain phenotype; C: 100-kernel weight; “**” indicate significant difference between wild type and sh2021 at the 0.01 level (t test)
图 4 SNP-index的全基因组分布图
散点为原始值,黑色曲线为拟合值;蓝色阈值线和红色阈值线分别表示各位点以及窗口的ΔSNP-index 95%和99%置信区间;横坐标为染色体的分布,每个点代表ΔSNP-index值
Figure 4. Genome-wide distribution of SNP-index
The scatter points are the original values, and the black curve is the fitted value; Blue threshold line and red threshold line indicate the ΔSNP-index 95% and 99% confidence intervals, respectively; The horizontal coordinate is the distribution of chromosomes, and each dot represents the ΔSNP-index value
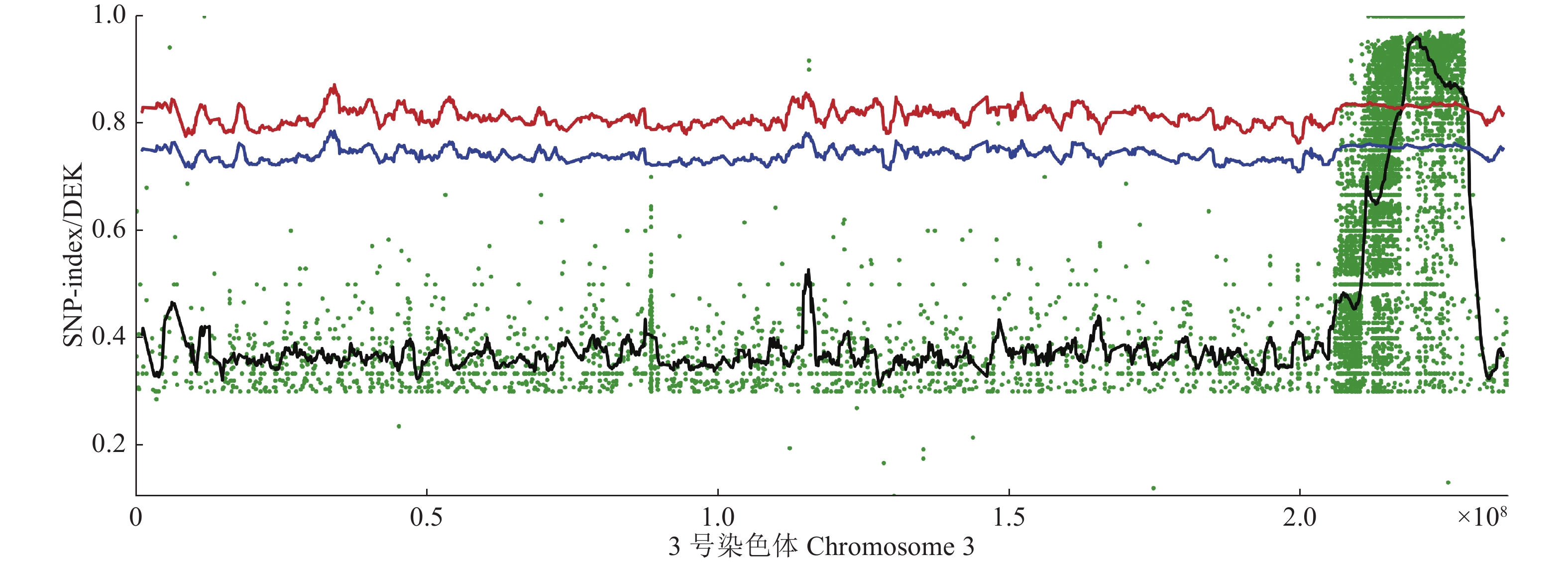
图 5 SNP-index在3号染色体上的分布
散点图为原始值,黑色曲线为拟合值,其他横线为指定阈值位置;横坐标为3号染色体的分布,每个点代表的ΔSNP-index值
Figure 5. Distribution of SNP-index on Chromosome 3
The scatter plot is the original value, the black curve is the fitted value, and the other horizontal lines are the specified threshold positions; The horizontal coordinate is the distribution of Chr 3, and each dot represents the ΔSNP-index value
表 1 sh2021 基因定位引物
Table 1 Primers used for gene mapping of sh2021
引物名称
Primer name序列(5′→3′)
SequenceID1 F:TACAGCTTGTAAAAATACAGGGCC
R:ATGAGAGTGCATGGTCCGTGID2 F:GGCAGCAGGATCAGAAGAGA
R:GGAACTTGTTGTGCCAAGGID3 F:TCTCGAATCAAGAACCAGCA
R:GGAGGGATTGGGTGAAGATTID4 F:GTCTACAACCGGCTCCTCAA
R;GTGGAAGTGTCGTCCGTTCTID5 F:GCCATCTACAGTGTCAGCCA
R:GTCAACGAGATTCTCAGGGCID9 F:CTTCCCGTCGAGATCCACG
R:GTTGACAGCTTGTGGGGTCTID10 F:GCTAGAGATGGCTATTAGGTGCA
R:AGCGTGTCTAACCTCACTTATAACAID11 F:TAACTGTCTCGCTGGGCTTT
R:GGCGTCCGTCCAAATAAAID12 F:CCATGAGGAGGCATGTGTGG
R:CAGCTGGTTCACGCCACATCID13 F:TGCACTTCGAGAGGTTTGTT
R:TCATAATAGTCTCGTATCCAGCCTID14 F:ACCCAAGGACCCCAAAGAGA
R:GCCCTATGCTTGGGGAGTGID15 F:GCCTGTCTAGCATATGATGTGAA
R:GTCTCATGCCAAACGAGGATC3-102 F:CTAGCCAGCCCCCATTCTTC
R:GCAAGGAGTAGGGAGGACGTG表 2 qRT-PCR定量验证引物
Table 2 qRT-PCR quantitative verification primers
引物名称
Primer name序列(5′→3′)
SequenceHX1 F:TTTGAGCGAAACTCCAGTGC
R:ATGTTCATGTGCTGCAGGACHX2 F:GAGTCGGAGATCAGCATCGG
R:AAAGTGTCGGCGAAGAACCTHX3 F:TTGGGAAGACGAAGCCATTG
R:AGGTCGGCTTTCCATATGGACHX4 F:TCACTGAGCAGCCTTCCAAG
R:GACACGTGAGCAGACTCCAAHX5 F:GCCACGGGATCATGAAGGAG
R:TCTCCTCCAACAGACCCTCC -
[1] NEUFFER M G, SHERIDAN W F. Defective kernel mutants of maize: I: Genetic and lethality studies[J]. Genetics, 1980, 95(4): 929-944. doi: 10.1093/genetics/95.4.929
[2] BRUNELLE D C, CLARK J K, SHERIDAN W F. Genetic screening for EMS-induced maize embryo-specific mutants altered in embryo morphogenesis[J]. G3-Genes Genomics Genetics, 2017, 7(11): 3559-3570.
[3] WU Y R, MESSING J. Proteome balancing of the maize seed for higher nutritional value[J]. Frontiers in Plant Science, 2014, 5: 240.
[4] SCHMIDT R J, BURR F A, AUKERMAN M J, et al. Maize regulatory gene opaque-2 encodes a protein with a “leucine-zipper” motif that binds to zein DNA[J]. Proceedings of the National Academy of Sciences of the United States of America, 1990, 87(1): 46-50. doi: 10.1073/pnas.87.1.46
[5] AZEVEDO R A, LEA P J, DAMERVAL C, et al. Regulation of lysine metabolism and endosperm protein synthesis by the opaque-5 and opaque-7 maize mutations[J]. Journal of Agricultural and Food Chemistry, 2004, 52(15): 4865-4871. doi: 10.1021/jf035422h
[6] MYERS A M, JAMES M G, LIN Q. Maize opaque5 encodes monogalactosyldiacylglycerol synthase and specifically affects galactolipids necessary for amyloplast and chloroplast function[J]. Plant Cell, 2011, 23(6): 2331-2347. doi: 10.1105/tpc.111.087205
[7] FU S N, SCANLON M J. Clonal mosaic analysis of empty pericarp2 reveals nonredundant functions of the duplicated heat shock factor binding proteins during maize shoot development[J]. Genetics, 2004, 167(3): 1381-1394. doi: 10.1534/genetics.104.026575
[8] CHETTOOR A M, YI G, GOMEZ E, et al. A putative plant organelle RNA recognition protein gene is essential for maize kernel development[J]. Journal of Integrative Plant Biology, 2015, 57(3): 236-246. doi: 10.1111/jipb.12234
[9] YANG Y Z, DING S, WANG Y, et al. Small kernel2 encodes a glutaminase in vitamin B6 biosynthesis essential for maize seed development[J]. Plant Physiology, 2017, 174(2): 1127-1138. doi: 10.1104/pp.16.01295
[10] 薛慧, 张国治, 吕飞杰, 等. 抗性淀粉测定方法的研究[J]. 河南工业大学学报(自然科学版), 2012, 33(4): 57-60. doi: 10.16433/j.cnki.issn1673-2383.2012.04.017 [11] DICKINSON D B, PREISS J. Presence of ADP-glucose pyrophosphorylase in shrunken-2 and brittle-2 mutants of maize endosperm[J]. Plant Physiology, 1969, 44(7): 1058-1062. doi: 10.1104/pp.44.7.1058
[12] 周瑞颐, 青芸, 李道杨, 等. 玉米ZmBT1研究进展[J]. 分子植物育种, 2020, 18(20): 6702-6706. doi: 10.13271/j.mpb.018.006702 [13] MAGALIE C, PIERRE C, SYLVIE M, et al. Transcriptional and metabolic adjustments in ADP-glucose pyrophosphorylase-deficient bt2 maize kernels[J]. Plant Physiology, 2008, 146(4): 1553-1570. doi: 10.1104/pp.107.112698
[14] 宋欣冉, 胡书婷, 张凯, 等. 玉米籽粒突变体dek101的表型分析和精细定位[J]. 作物学报, 2020, 46(12): 1831-1838. [15] 石慧敏, 蒋成功, 王红武, 等. 玉米籽粒突变体dek48的表型鉴定与基因定位[J]. 作物学报, 2020, 46(9): 1359-1367. [16] HE Y H, WANG J G, QI W W, et al. Maize Dek15 encodes the cohesin-loading complex subunit SCC4 and is essential for chromosome segregation and kernel development[J]. The Plant Cell, 2019, 31(2): 465-485. doi: 10.1105/tpc.18.00921
[17] WANG G F, WANG F, WANG G, et al. Opaque1 encodes a myosin XI motor protein that is required for endoplasmic reticulum motility and protein body formation in maize endosperm[J]. The Plant Cell, 2012, 24(8): 3447-3462. doi: 10.1105/tpc.112.101360
[18] WANG G, QI W W, WU Q, et al. Identification and characterization of maize floury4 as a novel semidominant opaque mutant that disrupts protein body assembly[J]. Plant Physiology, 2014, 165(2): 582-594. doi: 10.1104/pp.114.238030
[19] GILLIKIN J W, ZHANG F, COLEMAN C E, et al. A defective signal peptide tethers the floury-2 zein to the endoplasmic reticulum membrane[J]. Plant Physiology, 1997, 114(1): 345-352. doi: 10.1104/pp.114.1.345
[20] KIM C S, GIBBON B C, GILLIKIN J W, et al. The maize Mucronate mutation is a deletion in the 16-kDa γ-zein gene that induces the unfolded protein response1[J]. The Plant Journal, 2006, 48(3): 440-451. doi: 10.1111/j.1365-313X.2006.02884.x
[21] KUSHWAHA H R, SINGH A K, SOPORY S K, et al. Genome wide expression analysis of CBS domain containing proteins in Arabidopsis thaliana (L. ) Heynh and Oryza sativa L. reveals their developmental and stress regulation[J]. BMC Genomics, 2009, 10: 200. doi: 10.1186/1471-2164-10-200
[22] 王立成. 玉米抗旱高通量FOX文库的构建与验证[D]. 杨凌: 西北农林科技大学, 2019. [23] 崔会婷, 蒋旭, 张铁军, 等. 植物CYP450家族研究进展[J]. 中国草地学报, 2020, 42(5): 173-180. doi: 10.16742/j.zgcdxb.20190182 [24] 刘薇, 张彦威, 李伟, 等. 大豆细胞色素P450基因GmCYP78A69的克隆和生物信息学分析[J]. 分子植物育种, 2020, 18(14): 4523-4531. [25] CHEN X, LIU W, HUANG X, et al. Arg-type dihydroflavonol 4-reductase genes from the fern Dryopteris erythrosora play important roles in the biosynthesis of anthocyanins[J]. PLoS One, 2020, 15(5): e232090. doi: 10.1371/journal.pone.0232090.
[26] GU Z Y, CHEN H, YANG R N, et al. Identification of DFR as a promoter of anthocyanin accumulation in poinsettia (Euphorbia pulcherrima, Willd. ex Klotzsch) bracts under short-day conditions[J]. Scientia Horticulturae, 2018, 236: 158-165. doi: 10.1016/j.scienta.2018.03.032
[27] DEIKMAN J, HAMMER P E. Induction of anthocyanin accumulation by cytokinins in Arabidopsis thaliana[J]. Plant Physiology, 1995, 108(1): 47-57. doi: 10.1104/pp.108.1.47
[28] BAI M Y, FAN M, OH E, et al. A triple helix-loop-helix/basic helix-loop-helix cascade controls cell elongation downstream of multiple hormonal and environmental signaling pathways in Arabidopsis[J]. The Plant Cell, 2012, 24(12): 4917-4929.
[29] 刘浩. 转录因子ABP7在玉米籽粒发育过程中的功能及其分子机理分析[D]. 北京: 中国农业大学, 2017. -
期刊类型引用(10)
1. 邓永涛,尹苗,王聪,陈希文. 猪舍环境影响因子预测模型的研究进展. 家畜生态学报. 2025(02): 122-128 .  百度学术
百度学术
2. 刘艳昌,吴延昌,郭颖轩,李冬阳,张志霞,左现刚,李国厚. 生猪生长环境舒适度实时监测系统设计与实现. 家畜生态学报. 2024(06): 76-81+96 .  百度学术
百度学术
3. 刘丙杰,侯慕馨,冀海燕. 基于支持向量机集成的船舶舱室温湿度预测. 海军工程大学学报. 2024(03): 21-25+32 .  百度学术
百度学术
4. 朱佳明,孙彬,蒲施桦,潘学民,徐顺来,胡彬,齐仁立. 基于多环境因素分析的猪舍温湿度预测模型. 华南农业大学学报. 2024(05): 709-721 .  本站查看
本站查看
5. 杨晓平,李雪寒,聂鹏程. 设施生猪环境调控现状研究和未来展望. 中国农业信息. 2024(02): 83-96 .  百度学术
百度学术
6. 尤德安,邬锡权,陆畅,董钊杰,曾志雄,吕恩利. 数字孪生技术在现代化猪场生产应用的研究进展. 现代农业装备. 2023(04): 2-9+70 .  百度学术
百度学术
7. 翟子鹤,陈小文,高莉平,张天柱. 基于梯度提升算法的温室黄瓜株高生长模拟. 中国农业大学学报. 2022(05): 134-145 .  百度学术
百度学术
8. 杨东轩,吴叶兰,张刚刚,刘硕. 基于边缘计算与深度学习的禽舍监测系统设计. 江苏农业科学. 2022(09): 219-225 .  百度学术
百度学术
9. 陈谦,杨涵,王宝刚,李文生,钱建平. 基于GRU神经网络模型的冷链运输温度时序预测. 农业大数据学报. 2022(01): 82-88 .  百度学术
百度学术
10. 刘炫文,刘仁鑫. 猪舍小气候环境模拟预测技术研究综述. 江西农业学报. 2022(08): 122-126 .  百度学术
百度学术
其他类型引用(7)



 下载:
下载: