Biofilm-forming capacity of soil nitrogen-fixing bacteria and related gene expression under florfenicol combined with copper environmental stress
-
摘要:目的
研究氟苯尼考和铜(Cu)残留对土壤固氮菌的生态毒性效应,为评价兽药的环境风险提供依据。
方法对从花生根际土壤中分离出的一株优势固氮菌RpEC2071进行氟苯尼考和铜胁迫处理,试验设置空白组(0 μg/mL)、氟苯尼考添加组(40 μg/mL)、Cu添加组(200 μg/mL)和混合组(氟苯尼考40 μg/mL+Cu 200 μg/mL),并于给药后采样。采用苯酚硫酸法和96孔法研究氟苯尼考和Cu单独或联合处理对固氮菌胞外多糖产生及生物膜形成能力的影响。采用RT-qPCR测定分析固氮菌生物膜及固氮相关基因的mRNA表达水平。
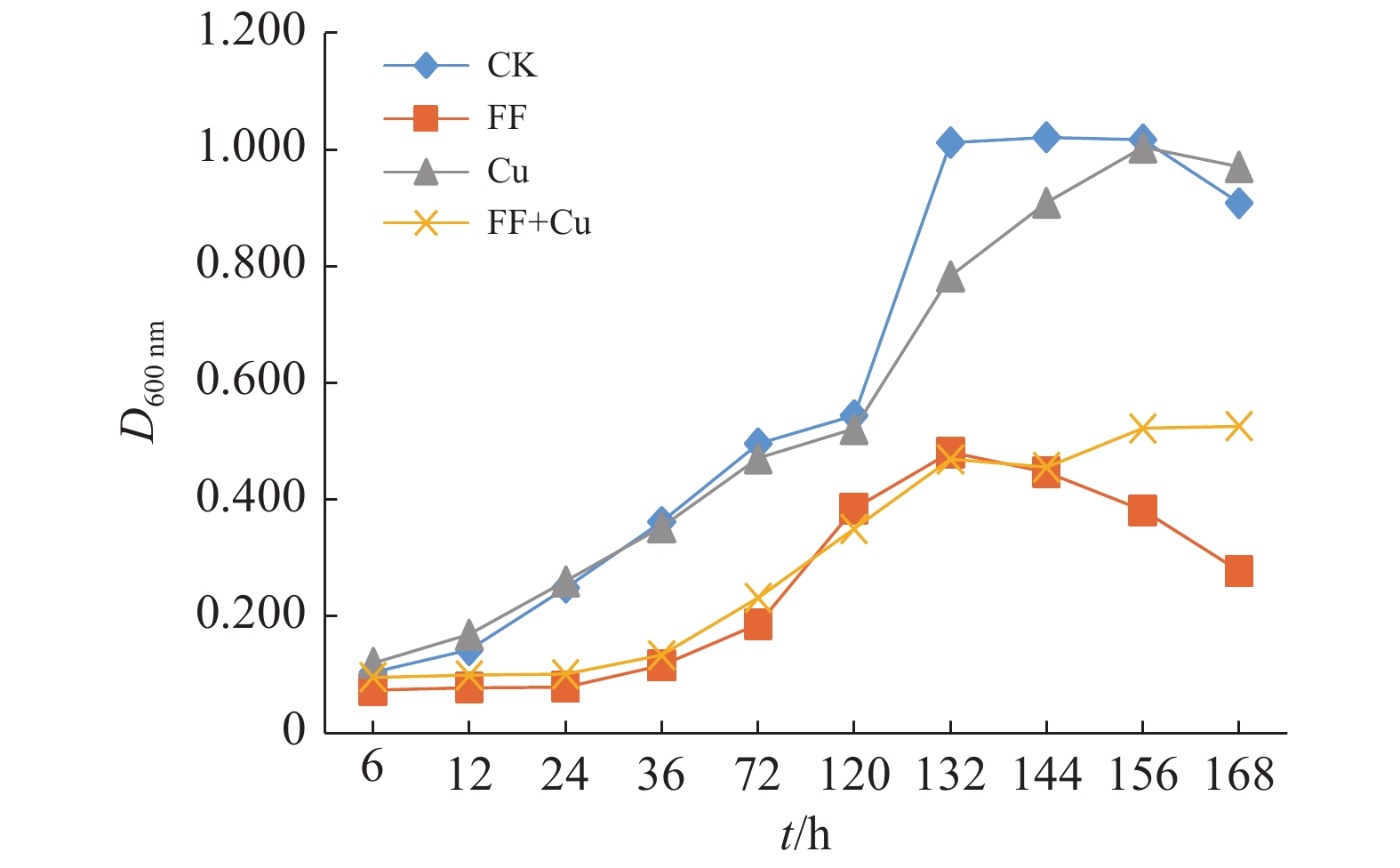
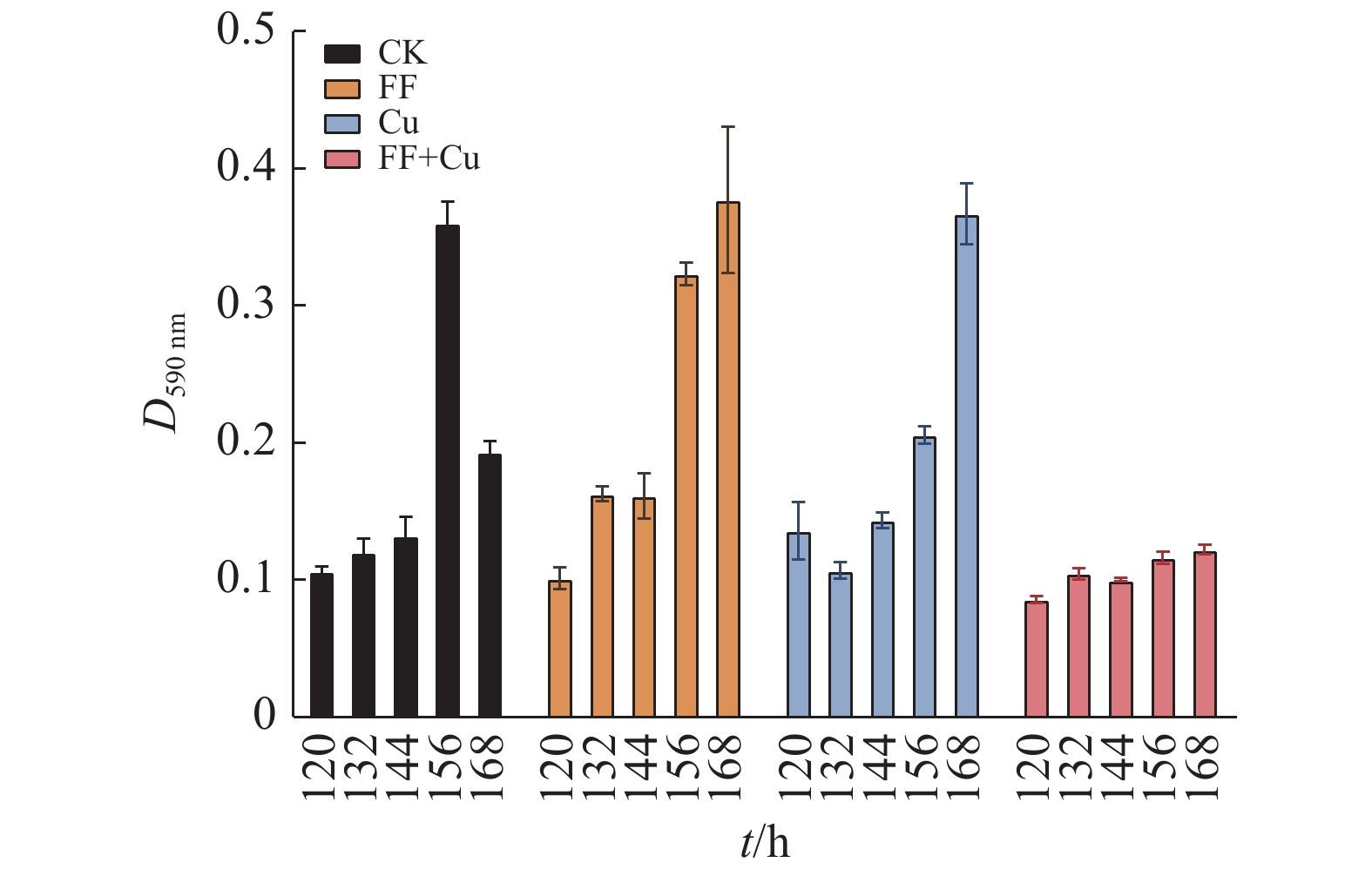
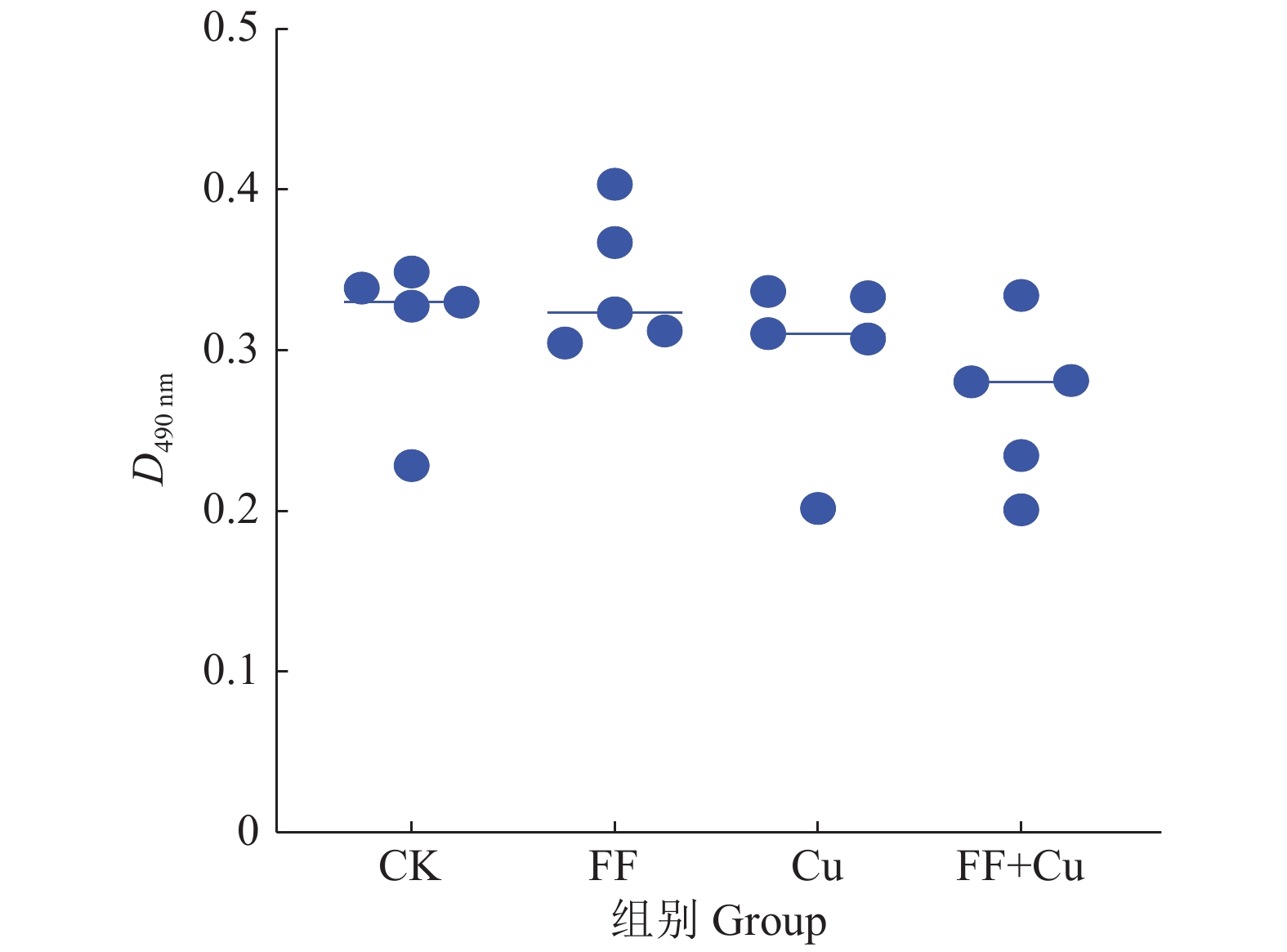
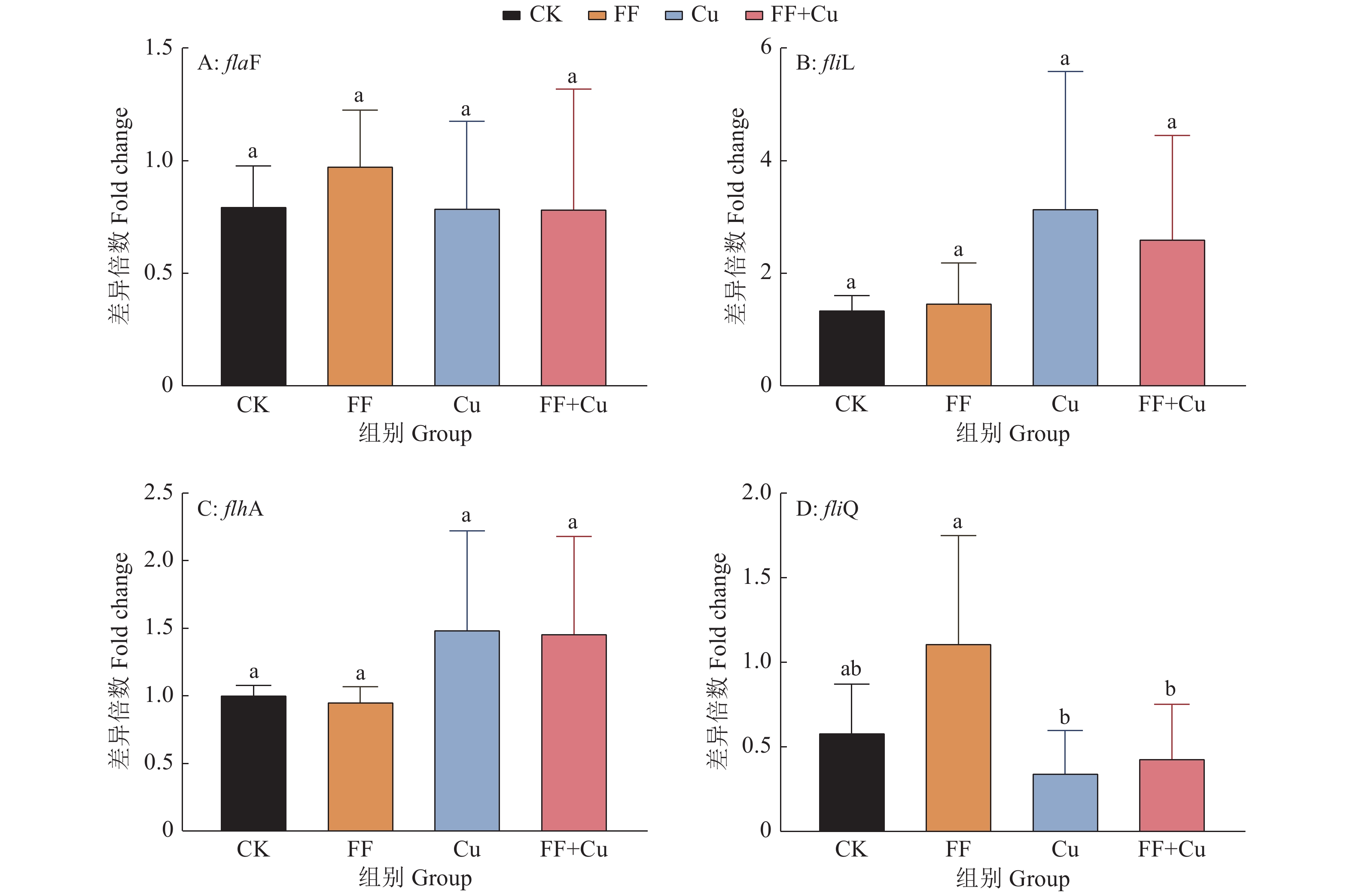
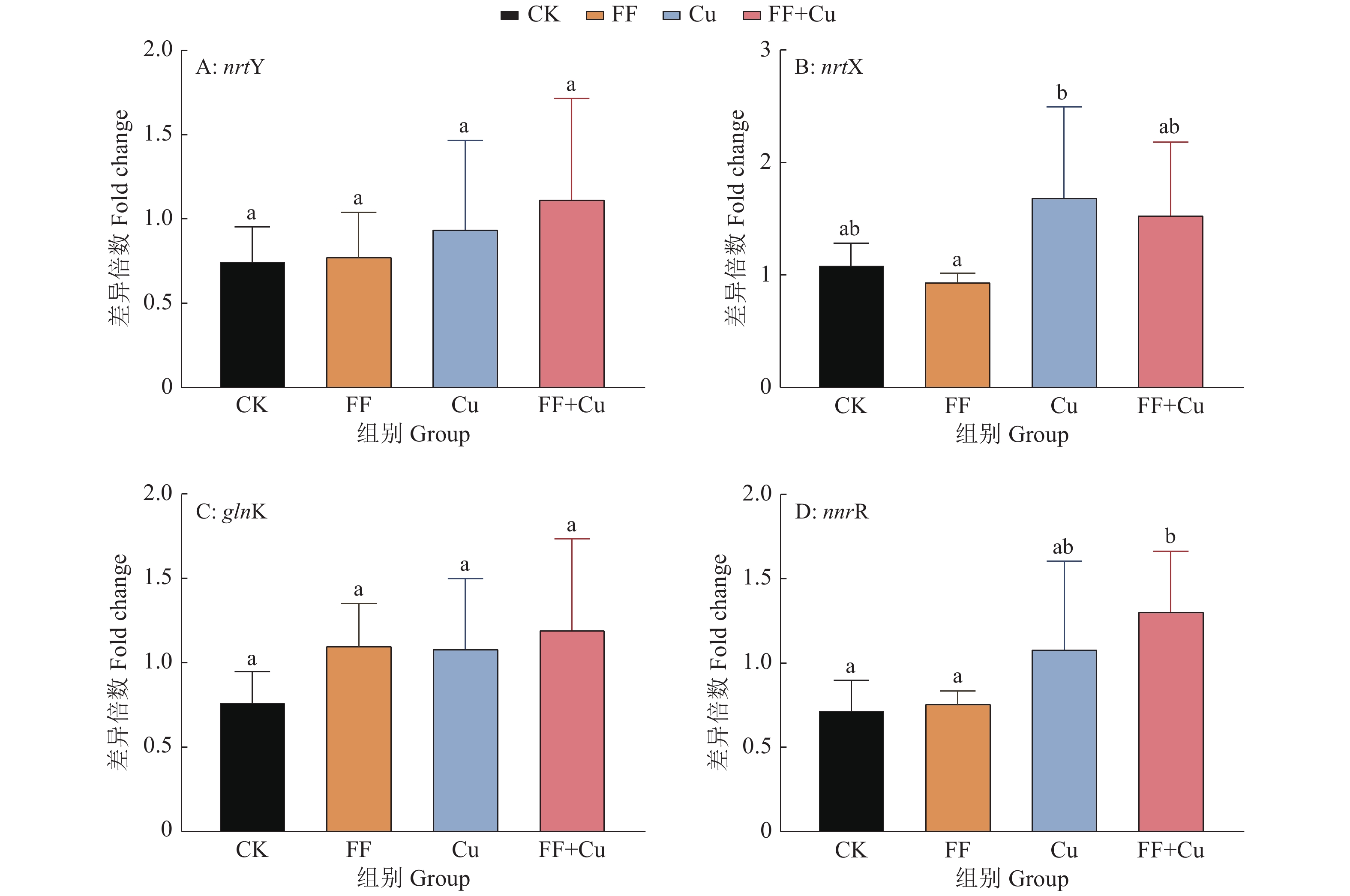
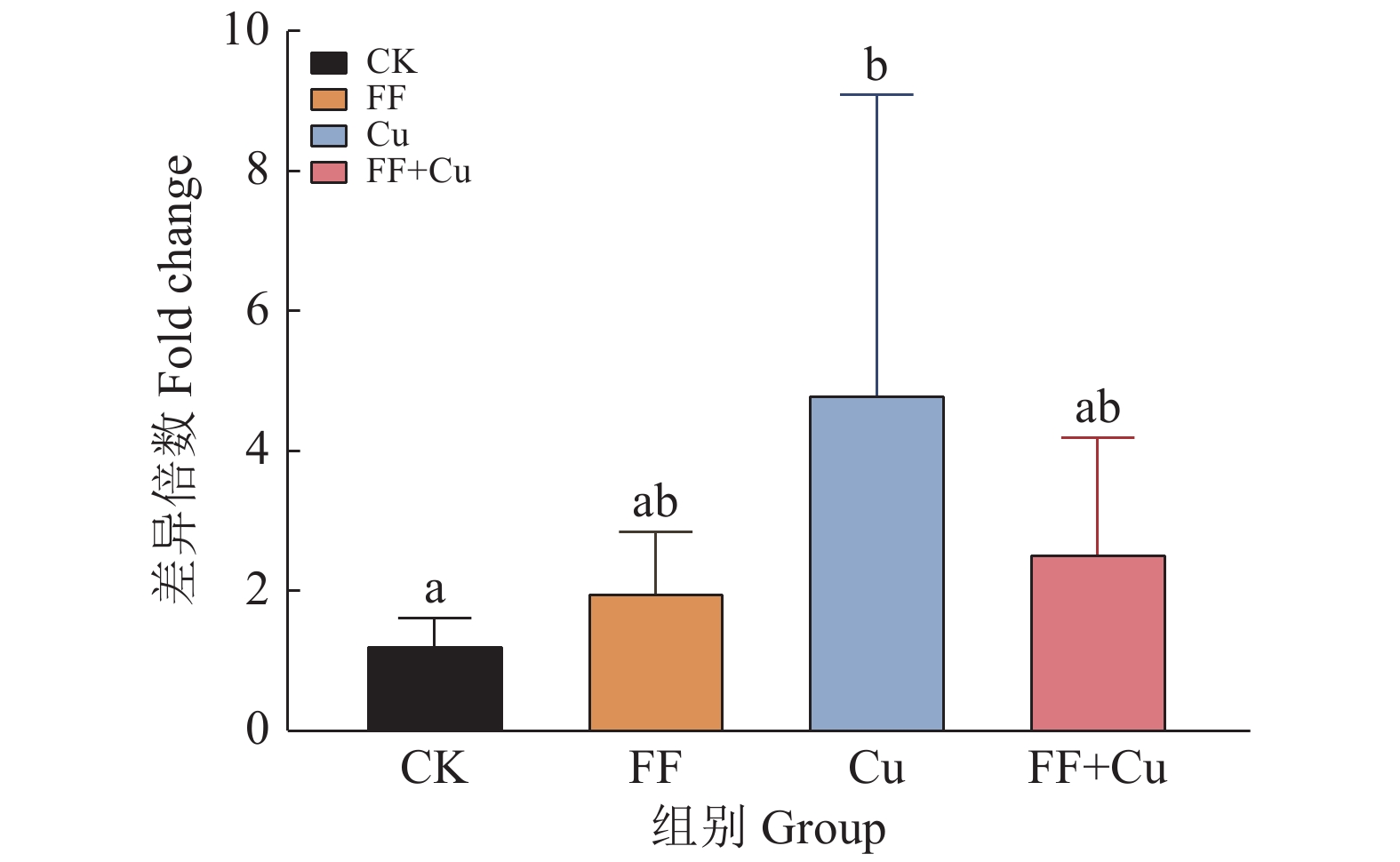
结果氟苯尼考和Cu单独胁迫会促进生物膜的形成,二者生物膜形成能力约为空白组的2倍;混合胁迫会抑制生物膜的形成,空白组生物膜形成能力是其3.1倍。胞外多糖分泌结果与生物膜形成能力测定结果基本一致。生物膜相关基因与氮代谢调控基因呈显著正相关,氟苯尼考和Cu的添加改变了fliQ、nifH和nnrR等基因的表达水平,且二者联合处理后会对其表达分别产生协同或拮抗作用。
结论氟苯尼考与Cu的单独或联合胁迫影响固氮菌RpEC2071中生物膜相关基因表达,进而对其生物膜形成能力产生影响。预示了以氟苯尼考和Cu为例的兽药及重金属残留胁迫会对土壤固氮菌产生潜在的生态毒性效应,从而破坏土壤中固氮生态系统。
Abstract:ObjectiveThe ecotoxic effects of florfenicol and copper (Cu) residues on soil nitrogen-fixing bacteria were studied to provide a basis for evaluating the environmental risks of veterinary drugs.
MethodA dominant nitrogen-fixing bacterium RpEC2071 was isolated from peanut root enclosure and treated under florfenicol and Cu stress. We set the blank group (0 μg/mL), florfenicol group (40 μg/mL), Cu group (200 μg/mL) and mixed group (flufenicol 40 μg/mL, Cu 200 μg/mL), and collected samples at multiple time points after dosing. The phenol-sulfuric acid method and 96-well microplate method were used to study the effects of florfenicol and Cu alone or in combination on the production of extracellular polysaccharides and biofilm formation of nitrogen-fixing bacterium. RT-qPCR was used to determine nitrogen-fixing bacterium biofilm formation and the mRNA expression levels of nitrogen fixation-related genes.
ResultFlorfenicol and Cu alone promoted the formation of biofilms, and the biofilm formation capacity of both was about twice that of the blank group. Under mixed stress, the biofilm formation was inhibited, and the biofilm formation capacity of the blank group was 3.1 times that of it. The results of extracellular polysaccharide secretion were basically consistent with the results of the determination of biofilm formation ability. Biofilm-related genes were significantly positively correlated with nitrogen metabolism regulatory genes, and the addition of florfenicol and Cu changed the expression levels of genes such as fliQ,ntrX andnnrR, and it would produce synergistic or antagonistic effects after the combination treatment of florfenicol and Cu.
ConclusionThe individual or combined stress of florfenicol and Cu affects the expression of biofilm-related genes in nitrogen-fixing bacteria RpEC2071, which in turn affects its ability to form biofilm. This research reveals the ecotoxic potential of veterinary drugs and heavy metal residue stresses on soil nitrogen-fixing bacteria, using florfenicol and Cu as examples. As a result, this can lead to the impairment of the nitrogen-fixing ecosystem in soil.
-
Keywords:
- Florfenicol /
- Copper /
- Nitrogen-fixing bacteria /
- Extracellular polysaccharide /
- Biofilm /
- Ecotoxicity
-
桑树Morus alba Linn.属桑科Moraceae桑属Morus,原产我国中部,是一种具有重要经济价值的落叶乔木。种桑养蚕是我国的传统产业,我国已有约4 000的桑树栽培历史[1]。随着“东桑西移”发展战略的实施,广西逐步成为我国蚕茧生产第一大省区和世界重要原料蚕茧生产基地,2012年,广西桑树种植面积达1.69×105 hm2,家蚕饲养量为655万张,蚕茧产量达2.56×105 t[2]。由可可毛色二孢Lasiodiplodia theobromae Pat.Griffon & Maubl桑树的新病害,2014年,Xie等[3]曾报道该病害在我国广西横县发生,发病面积达2 400 hm2,其后在广西其他蚕桑种植区均发现该病害的存在。
桑根腐病是一种根部新病害,目前生产上鲜见有防治效果好的化学药剂,且化学防治易对桑树和家蚕产生药害,因此,针对该病害开展生物防治的研究具有重要意义,而获得有显著拮抗效果的菌株是病害生物防治的基础。本研究采集健康桑树根际土壤,筛选能显著抑制L. theobromae的拮抗细菌,以期为桑根腐病的生物防治奠定基础。
1. 材料与方法
1.1 材料
Ezup柱式细菌基因组DNA抽提试剂盒、DNA Ladder Min Marker(SM0337)、Dream Taq Green PCR MasterMix(2×)、PCR引物等均购自上海生物工程技术服务有限公司;PDA粉(广东环凯微生物科技有限公司);快速革兰氏染色试剂盒(济南百博生物科技有限责任公司);孔雀石绿(天津市大茂化学试剂厂)。
NA培养基:牛肉浸膏3.0 g、蛋白胨5 g、葡萄糖2.5 g,琼脂粉15 g,用少量蒸馏水加热溶解后,补充蒸馏水至1 L。
可可毛色二孢Lasiodiplodia theobromae菌株由广西大学农学院植保系实验室分离保存。
1.2 方法
从广西自治区南宁市、河池市和来宾市的桑园中,采集健康桑树根际土壤用于分离拮抗细菌。参考孙正祥等[4]的方法分离和纯化细菌菌株。
1.2.1 拮抗细菌对L. theobromae菌丝生长的影响
采用平板对峙法[5]测定拮抗细菌对菌丝生长的影响。将保存的病原菌菌株和细菌菌株分别在PDA培养基和NA培养基上活化。用灭菌打孔器(直径6 mm)取L. theobromae菌饼接种至PDA培养基平板中央,其两侧2 cm处对称放置细菌菌落块,放置于28 ℃培养箱中培养。待对照处理的菌落长满平板后,测量各处理菌落的直径,参照谢晨昭等[6]的方法计算生长抑制率,并在显微镜下观察抑菌带菌丝的形态特征。各处理重复3次。从参试的细菌菌株中筛选出拮抗效果最好的菌株进行后续试验。
1.2.2 细菌培养液对L. theobromae菌丝生长的影响
将筛选出的细菌菌落块接种到150 mL液体NA培养基中,于振荡培养箱中(28 ℃,150 r·min-1)培养3 d。培养液经0.22 μm的微孔滤膜过滤后,备用。取活化好的L. theobromae菌饼接种到PDA培养基中央,在其周围对称放置3片灭菌滤纸片(直径5 mm),再用移液枪吸取10 μL细菌上清液滴在滤纸片上。重复3次,计算生长抑制率。
1.2.3 细菌培养液对L. theobromae孢子萌发的影响
采用载玻片孢子萌发法[5]:利用过滤的细菌培养液配制孢子悬浮液(约1×106个·mL-1),以清水为对照, 将500 μL孢子悬浮液滴在干净的载玻片上,再将载玻片放入垫有湿润滤纸的培养皿中,然后放置于28 ℃培养箱中培养20 h后观察孢子萌发情况,计算孢子萌发率。以芽管长度超过孢子长度的一半作为萌发标准。
1.2.4 拮抗菌株的鉴定
将活化好的拮抗菌株菌落划线接种在NA培养基平板上,放入28 ℃培养箱中培养2 d后,观察菌落特征,并进行革兰氏染色和芽孢染色观察形态特征[7]。参照《常见细菌系统鉴定手册》[7]的方法测定拮抗细菌的生理生化特征。试剂盒提取菌株总DNA,保存于-20 ℃备用。参考并利用Chun等[8]的引物扩增gyrA基因。PCR反应体系:Dream Taq Green PCR MasterMix(2×)25 μL,引物各2 μL,DNA模板2 μL,加ddH2O至50 μL。反应条件:95 ℃预变性3 min;95 ℃ 35 s,62 ℃ 1 min,72 ℃ 2 min,重复30次;72 ℃ 10 min。gyrB基因引物为UP1和UP2r[9]。除引物不同外,PCR反应体系中各成分的量和反应条件与gyrA基因序列扩增试验相同。
PCR扩增完成后,取PCR产物在10 g·L-1琼脂糖凝胶中电泳(150 V、100 mA、20 min)检测产物的特异性并委托上海生工测序。将测得的菌株序列在NCBI上进行BLAST比对,确定菌株的分类地位,同时下载NCBI中与参试菌株序列相似性高的序列用Mega 5软件进行同源性分析,构建系统发育树。
1.2.5 YZ14-3大田防治桑根腐病效果
在广西自治区横县石塘镇选择树龄为4~6年的桑园作为试验田,试验田随机设置3个小区,每小区65~70棵桑树。施用生防菌剂前,先调查记录好各小区的病死桑树。将YZ14-3培养液9 L(4.8×108 CFU·mL-1)兑水18 L后均匀浇灌于各小区桑树根部土壤。试验期限为2014年4月至2015年5月,前5个月,每月施用培养液2次,其后每月施用1次。对照用等量的清水代替YZ14-3培养液。最后1次施药1个月后,调查各处理小区及对照的病死株数,参照赖传雅的方法[10]并稍作改进计算防治效果:
防治效果=[(对照区校正病死率-处理区校正病死率)/对照区校正病死率]×100%,
校正病死率=用药后病死率-用药前病死率。
2. 结果与分析
2.1 拮抗菌株分离及拮抗效果测定
从采集的45份土样中分离到菌落形态有明显差异的菌株22个,经筛选得到对L. theobromae有显著抑制效果的8个菌株,其中菌株YZ14-3的抑菌效果最好(图 1)。YZ14-3菌落与L. theobromae对峙培养时,抑菌圈半径为12 mm,对L. theobromae的生长抑制率为73.3%。因此,选定菌株YZ14-3进行后续试验。研究发现,菌株YZ14-3的培养液能显著抑制L. theobromae的生长,抑菌圈半径为8 mm,抑制率为55.6%。观察发现抑菌圈外缘的L. theobromae菌丝变黑(图 1),挑取变黑的菌丝在显微镜下观察,发现菌丝膨大、畸形、易断裂(图 2)。
2.2 菌株YZ14-3培养液对L. theobromae孢子萌发的影响
采用载玻片孢子萌发法测定了YZ14-3培养液对L. theobromae分生孢子萌发的影响,培养20 h后,在显微镜下观察孢子的萌发情况。每个视野随机观测500个孢子,观察3个视野,共1 500个孢子。观察发现,L. theobromae分生孢子在YZ14-3的培养液中不能萌发且YZ14-3的培养液能降解L. theobromae分生孢子的细胞壁,使分生孢子解体。
2.3 菌株YZ14-3的鉴定
2.3.1 形态特征
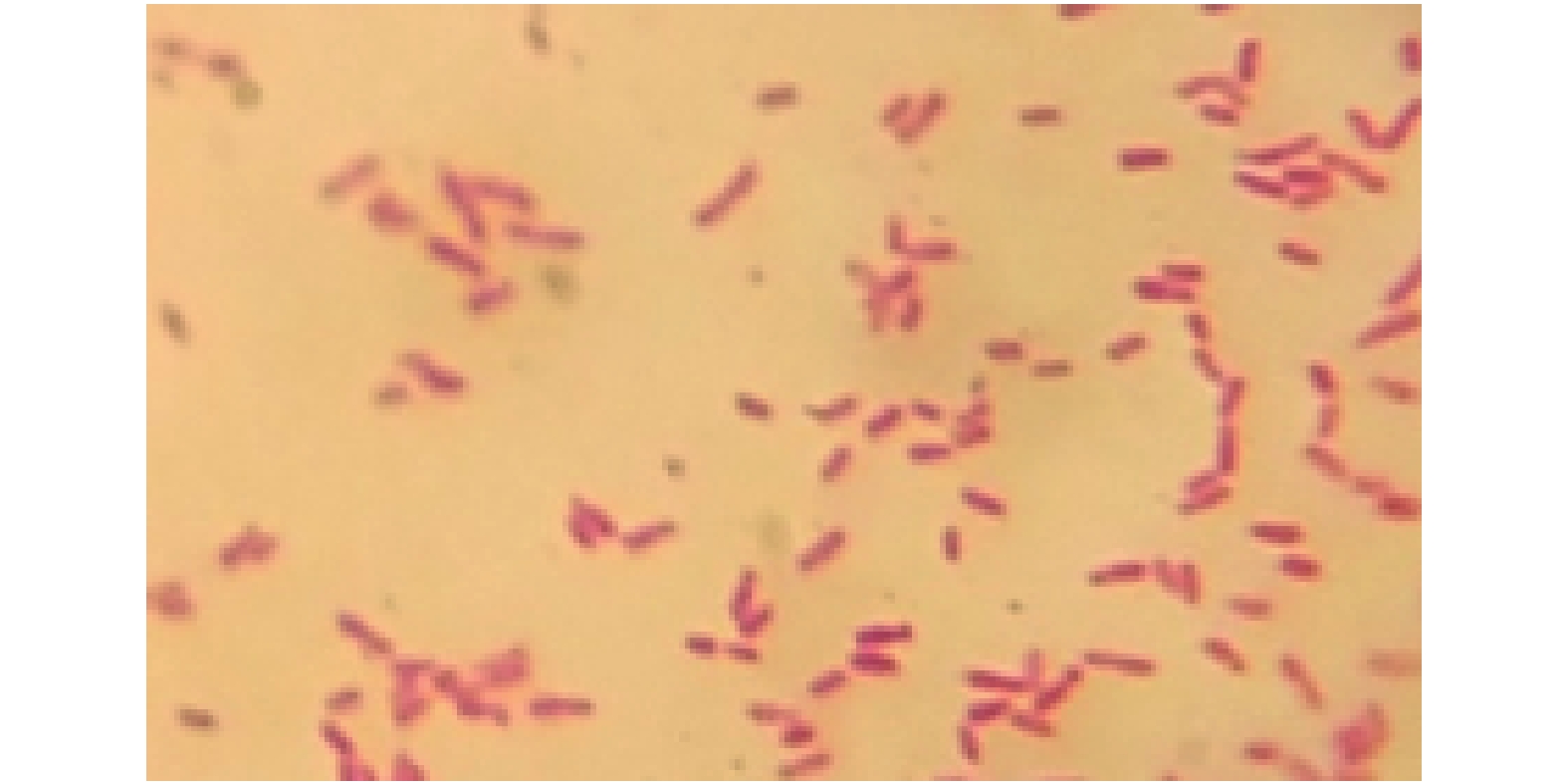
菌株YZ14-3在NA培养基的主要培养特征(图 3A)如下:菌落乳白色,边缘不整齐,表面有皱褶,隆起不透明,菌落干爽无光泽;液体静止培养时形成乳白色菌膜。显微镜下观察发现,YZ14-3革兰氏染色阳性,呈紫色(图 3B),菌体短杆状,直或略弯,菌体单个或呈短链状排列,芽孢顶生或生于菌体中部(图 3C)。根据形态特征,初步鉴定YZ14-3为芽孢杆菌Bacillus sp.。
2.3.2 生理生化特性
本研究测定了YZ14-3的部分生理生化特征(表 1)。将本文测定结果与已发表的B. amyloliquefaciens相关菌株生理生化特征测定结果[11-12]相比较,并参考《常见细菌系统鉴定手册》[7],YZ14-3被初步鉴定为解淀粉芽孢杆菌Bacillus amyloliquefaciens。
表 1 菌株YZ14-3的生理生化特性1)Table 1. Physiological and biochemical characteristics of YZ14-3
2.3.3 gyrA和gyrB序列分析鉴定
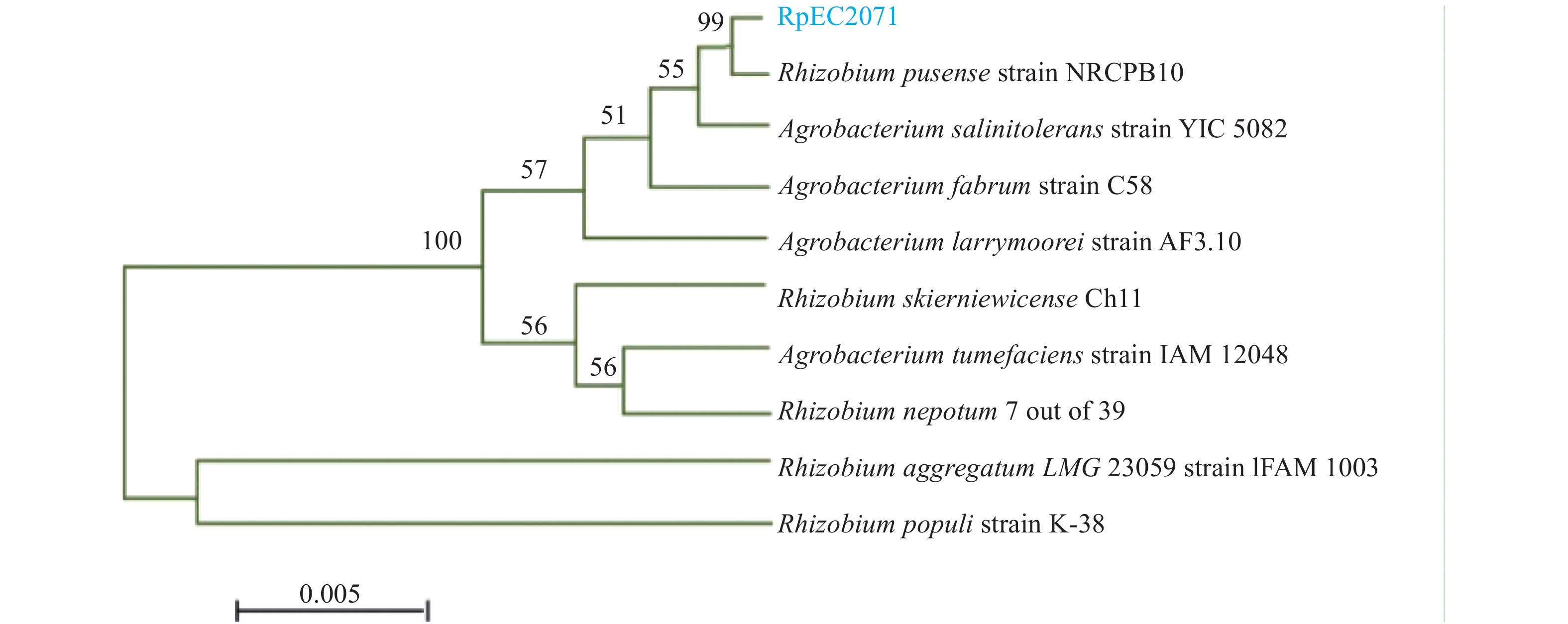
菌株YZ14-3的gyrA基因扩增后,获得1个长度为1 013 bp的片段。PCR产物纯化、测序后,将该基因序列在NCBI上进行BLAST相似性分析,比对结果表明,YZ14-3的gyrA序列与GenBank中的B. amyloliquefaciens的gyrA序列相似性均高于98%。从GenBank中下载与YZ14-3的gyrA序列相似性较高的B. amyloliquefaciens gyrA序列11个、B. subtilis序列8个、B. licheniformis序列6个,以大肠埃希菌Escherichia coli菌株的gyrA序列(登录号:DQ447131)为外群构建系统发育树(图 4A)。由系统发育树可知,YZ14-3与11株B. amyloliquefaciens聚在一群,并与其他菌株分离,表明YZ14-3与B. amyloliquefaciens具有很高的遗传相似性。
菌株YZ14-3的gyrB基因扩增后,获得1个长度为1 243 bp的片段。PCR产物纯化、测序后,将该基因序列在NCBI上进行BLAST相似性分析,结果发现,YZ14-3的gyrB序列与GenBank中的B. amyloliquefaciens的gyrB序列相似性均高于99%。下载GenBank中与YZ14-3的gyrB序列相似性较高的7个B. amyloliquefaciens菌株序列、7个B. subtilis序列、3个B. licheniformis序列、3个B. thuringiensis序列,以E.coli菌株的gyrB序列(登录号:AB083949)为外群构建系统发育树(图 4B)。由系统发育树可知,YZ14-3与7株B. amyloliquefaciens聚在一群,并与7株B. subtilis分离,表明YZ14-3与B. amyloliquefaciens的遗传相似性很高。
由gyrA和gyrB序列构建的系统发育树可鉴定菌株YZ14-3为B. amyloliquefaciens。
2.3.4 菌株YZ14-3的田间防治效果
施用YZ14-3培养液前后各调查1次桑树病死情况,施用YZ14-3培养液用药前和用药后桑树病死率分别为14.44%和18.89%,用药后的桑树病死率显著地低于对照的(29.44%)。生防菌剂处理的校正病死率约为4.45%,显著高于对照的校正病死率13.88%。由生防菌剂处理和对照的校正病死率得YZ14-3的田间防治效果为67.94%。
3. 讨论与结论
B. amyloliquefaciens与B. subtilis亲缘性很高,能分泌一系列抑制真菌、细菌、病毒和支原体生长发育的抗菌脂肽[13]。王奕文等[14]从甜瓜果实表面分离到1株B. amyloliquefaciens,该菌株对灰葡萄孢Botrytis cinerea、链格孢Alternaria sp.、尖孢镰刀菌Fusarium. oxysporum等病原真菌的拮抗作用显著。陈妍等[15]从土壤中分离出1株对棉花黄萎病菌Verticillium dahliae有良好拮抗作用的B. amyloliquefaciens菌株。陈成等[16]从土壤中分离到1株B. amyloliquefaciens,其对黑曲霉Aspergillus niger、稻瘟病菌Magnaporthe oryzae和水稻纹枯病菌Rhizoctonia solani等植物病原真菌有很强的抑制作用。B. amyloliquefaciens菌株DFE16及其发酵液不仅能抑制病菌生长,而且能诱导油菜对黑胫病产生抗性[17]。B. amyloliquefaciens的发酵液对油茶炭疽病具有很强的抑制作用,进一步研究发现发酵液中的抑菌物质为脂肽,该物质能使病菌菌丝畸形[18]。B. amyloliquefaciens分泌的胞外非蛋白类物质能有效抑制鱼腥藻的生长[19]。枯草芽孢杆菌不同种群在植物病害的生物防治中有广泛的应用,其作用机制主要有竞争、抗生、溶菌等[20]。本研究从健康桑树的根际土壤中分离出1株B. amyloliquefaciens YZ14-3,其对桑根腐病菌L. theobromae的抑制效果显著,YZ14-3的无菌培养液能完全抑制病菌分生孢子的萌发,降解分生孢子的细胞壁,从而使孢子解体,说明YZ14-3的培养液中含有溶菌物质。溶菌作用是B. amyloliquefaciens菌株YZ14-3抑制病原菌生长的一种机制。田间防治试验结果表明,YZ14-3的生防效果较显著达67.94%,具有一定的应用前景。本研究结果为菌株YZ14-3在桑根腐病生物防治中的应用奠定了基础。
16S rDNA/RNA基因序列被广泛应用于细菌鉴定或研究细菌的系统进化关系,但由于16S rDNA/RNA基因序列过于保守,在亲缘关系很近的分类类群间,由于序列间的相似度太高而无法区分近缘种[21]。Wang等[22]研究认为16S rDNA/RNA基因序列不能有效区分枯草芽孢杆菌的菌株,而gyrA和gyrB基因序列可以用于枯草芽孢杆菌的鉴定。gyrA和gyrB基因的分子进化速率比16S rDNA/RNA基因大,可以弥补16S rDNA/RNA基因的不足[22-23]。菌株SWB16的16S rRNA序列与B. subtilis、B. amyloliquefaciens、B. licheniformis和贝莱斯芽孢杆菌B. velezensis有99%的相似性,但利用gyrA序列构建的系统发育树显示SWB16与B. amyloliquefaciens聚为一群[24]。本文利用gyrA和gyrB基因序列构建了系统发育树,2株系统发育树均能将B. amyloliquefaciens与B. subtilis及其他芽孢杆菌近缘种区分开。目前,gyrA和gyrB基因已经应用到许多细菌近缘种的鉴别中,如枯草芽孢杆菌组(B. subtilis group)[21, 25-26]、假单孢菌属Pseudomonas[27]、气单孢菌属Aeromonas[28]、分枝杆菌属Mycobacterium[29]等。
-
表 1 96孔板中处理组各组分添加量
Table 1 Addition of each component in a 96-well plate of treatment group
μL 组别 Group 改良肉汤 Improved broth 菌液 Bacterial solution 甲醇 Methanol 氟苯尼考1)Florfenicol CuSO42) 空白组 Blank(CK) 170 20 10 0 0 氟苯尼考组 Florfenicol(FF) 170 20 0 10 0 铜组 Cu(Cu) 160 20 10 0 10 混合组 Mixed(FF+Cu) 160 20 0 10 10 1) ρ (氟苯尼考) =0.8 mg/mL;2) ρ (CuSO4) = 4 mg/mL 1)ρ (Florfenicol) = 0.8 mg/mL; 2) ρ (CuSO4) = 4 mg/mL 表 2 胁迫模型组各组分添加量
Table 2 Addition of each component in stress model groups
mL 组别 Group 改良肉汤 Improved broth 菌液 Bacterial solution 甲醇 Methanol 氟苯尼考1)Florfenicol CuSO42) 空白组 Blank(CK) 40.1 0.1 0.1 0 0 氟苯尼考组 Florfenicol(FF) 40.1 0.1 0 0.1 0 铜组 Cu(Cu) 40.0 0.1 0.1 0 0.1 混合组 Mixed(FF+Cu) 40.0 0.1 0 0.1 0.1 1) ρ (氟苯尼考)=16 mg/mL;2) ρ (CuSO4)= 80 mg/mL 1) ρ (Florfenicol) = 16 mg/mL; 2) ρ (CuSO4) = 80 mg/mL 表 3 引物序列
Table 3 Primer sequences
基因名称 Gene name 引物序列(5′→3′) Primer sequence 基因名称 Gene name 引物序列(5′→3′) Primer sequence flaF F: GCGAGCGACAGGCGTTGA R: TGATTATCCGGCTGCTTGAGATC ntrX F: ACTTGTCGGTGCGTCACTTGC R: GATGGGCTTCTTCCAGTGCG fliL F: AAAACGAACAGGCAGAGGGC R: GGGAAACGGTGCGGACATAG glnK F: TGACCGTGACCGAAGTAAAGGG R: TGCCGTCGCCGATCTGCC flhA F: ACCACCAGTCATTTCCTTGCCC R: CCGCCGTCGGACCCTCAT nnrR F: GCTGGACGGATTGCTGACCC R: GCCACCCGACGCTCTACCTC fliQ F: ATCGTCGGTGTCGCCATCG R: CGTCATTTCCTGAACCTGCGTC nifH F: CGGATTATCGCAGTAGCAAACC R: TCTGTCGTCTCCATCGCTTCAC ntrY F: CGCTGACACCAAGTTCACGACG R: GCATCATGGAGTTCCAGATACCC Rp16S-1 F: AGATGCTCTACCTTGATGTCCCTG R: AGATGCGTTGCGCCACCT -
[1] 牛金利, 彭金菊, 明月月, 等. 氟苯尼考胁迫对土壤细菌耐药性的影响[J]. 中国兽医杂志, 2019, 55(4): 95-98. [2] CHOI J Y, KIM Y, KO E A, et al. Acinetobacter species isolates from a range of environments: Species survey and observations of antimicrobial resistance[J]. Diagnostic Microbiology and Infectious Disease, 2012, 74(2): 177-180. doi: 10.1016/j.diagmicrobio.2012.06.023
[3] 马驿, 彭金菊, 王芸, 等. 环丙沙星对土壤微生物量碳和土壤微生物群落碳代谢多样性的影响[J]. 生态学报, 2013, 33(5): 1506-1512. [4] 呼秀智, 薛占永, 王绥华, 等. 氟苯尼考对土壤微生物活动影响研究[J]. 山东畜牧兽医, 2011, 32(2): 3-5. [5] CAI L M, WANG Q S, LUO J, et al. Heavy metal contamination and health risk assessment for children near a large Cu-smelter in central China[J]. Science of the Total Environment, 2019, 650: 725-733. doi: 10.1016/j.scitotenv.2018.09.081
[6] 施昊坤, 吴次芳, 张茂鑫, 等. 土地整治对工业区周边土壤微生物多样性和群落结构影响分析[J]. 环境科学学报, 2020, 40(1): 212-223. [7] GAO M L, SONG W H, ZHOU Q, et al. Interactive effect of oxytetracycline and lead on soil enzymatic activity and microbial biomass[J]. Environmental Toxicology and Pharmacology, 2013, 36(2): 667-674. doi: 10.1016/j.etap.2013.07.003
[8] BAO Y Y, CHEN Q, MA W, et al. Influence of Fe addition on the accumulation of oxytetracycline in rice seedlings (Oryza sativa L. ) growing in hydroponic and soil culture[J]. Journal of Soils and Sediments, 2018, 18(5): 1958-1970. doi: 10.1007/s11368-018-1920-8
[9] WANG M, WU J, ZHOU T, et al. Effects of copper and florfenicol on nirS- and nirK-type denitrifier communities and related antibiotic resistance in vegetable soils[J]. Ecotoxicology and Environmental Safety, 2021, 213: 112011. doi: 10.1016/j.ecoenv.2021.112011
[10] FALKOWSKI P G. Volution of the nitrogen cycle and its influence on the biological sequestration of CO2 in the ocean[J]. Nature, 1997, 387(6630): 272-275. doi: 10.1038/387272a0
[11] 鲁文茹, 刘广锋, 郭志勋, 等. 金银花、连翘对溶珊瑚弧菌及其生物膜活性的影响[J]. 广东药学院学报, 2014, 30(3): 297-300. [12] 徐光域, 颜军, 郭晓强, 等. 硫酸−苯酚定糖法的改进与初步应用[J]. 食品科学, 2005, 26(8): 342-346. [13] 邹晓蕾, 刘礼崔, 罗立新. 细菌总RNA提取方法的比较[J]. 现代食品科技, 2013, 29(8): 1948-1954. [14] KARATAN E, WATNICK P. Signals, regulatory networks, and materials that build and break bacterial biofilms[J]. Microbiology and Molecular Biology Reviews, 2009, 73(2): 310-347. doi: 10.1128/MMBR.00041-08
[15] 冯世文, 曾芸, 李军, 等. 细菌对氟苯尼考的耐药机制研究[J]. 黑龙江畜牧兽医, 2014(5): 52-54. doi: 10.13881/j.cnki.hljxmsy.2014.0015 [16] 杜心恬, 宋馨, 刘欣欣, 等. 细菌胞外多糖生物合成转录调控因子研究进展[J]. 微生物学通报, 2021, 48(2): 573-581. [17] BOGINO P C, DE LAS MERCEDES OLIVA M, SORROCHE F G, et al. The role of bacterial biofilms and surface components in plant-bacterial associations[J]. International Journal of Molecular Sciences, 2013, 14(8): 15838-15859. doi: 10.3390/ijms140815838
[18] WANG D, XU A M, ELMERICH C, et al. Biofilm formation enables free-living nitrogen-fixing rhizobacteria to fix nitrogen under aerobic conditions[J]. The ISME Journal, 2017, 11(7): 1602-1613. doi: 10.1038/ismej.2017.30
[19] LI S, PENG C, CHENG T, et al. Nitrogen-cycling microbial community functional potential and enzyme activities in cultured biofilms with response to inorganic nitrogen availability[J]. Journal of Environmental Sciences, 2019, 76: 89-99. doi: 10.1016/j.jes.2018.03.029
[20] YANG Z M, HAN Y L, MA Y, et al. Global investigation of an engineered nitrogen-fixing Escherichia coli strain reveals regulatory coupling between host and heterologous nitrogen-fixation genes[J]. Scientific Reports, 2018, 8(1): 10913-10928. doi: 10.1038/s41598-018-29241-9
[21] LI K H, WU G C, LIAO Y L, et al. RpoN1 and RpoN2 play different regulatory roles in virulence traits, flagellar biosynthesis, and basal metabolism in Xanthomonas campestris[J]. Molecular Plant Pathology, 2020, 21(7): 907-922. doi: 10.1111/mpp.12938
[22] CALATRAVA M, NOGALES J, AMEZTOY K, et al. The NtrY/NtrX system of Sinorhizobium meliloti GR4 regulates motility, EPS I production, and nitrogen metabolism but is dispensable for symbiotic nitrogen fixation[J]. Molecular Plant-Microbe Interactions, 2017, 30(7): 566-577. doi: 10.1094/MPMI-01-17-0021-R
[23] HAO J, FENG Y, WANG X, et al. Soil microbial nitrogen-cycling gene abundances in response to crop diversification: A meta-analysis[J]. Science of the Total Environment, 2022, 838(4): 156621. doi: 10.1016/j.scitotenv.2022.156621.
[24] BUENO E, ROBLES E, TORRES M, et al. Disparate response to microoxia and nitrogen oxides of the Bradyrhizobium japonicum napEDABC, nirK and norCBQD denitrification genes[J]. Nitric Oxide, 2017, 68: 137-149. doi: 10.1016/j.niox.2017.02.002
[25] BUENO E, MANIA D, MESA S, et al. Regulation of the emissions of the greenhouse gas nitrous oxide by the soybean endosymbiont Bradyrhizobium diazoefficiens[J]. International Journal of Molecular Sciences, 2022, 23(3): 1486. doi: 10.3390/ijms23031486.




 下载:
下载: