Genome-wide identification and expression analysis of the β-amylase gene family in Ipomoea batatas
-
摘要:目的
挖掘甘薯Ipomoea batatas基因组中β−淀粉酶(Beta-amylase)基因家族序列信息,分析结构与功能信息。
方法基于甘薯栽培种‘泰中6号’全基因组测序数据,利用生物信息学分析方法对鉴定到的12个甘薯β−淀粉酶家族成员进行结构域保守性分析、染色体定位、潜在重复基因筛查、保守基序分析和系统进化树构建,利用转录组数据进行低温胁迫下相关基因的表达分析。
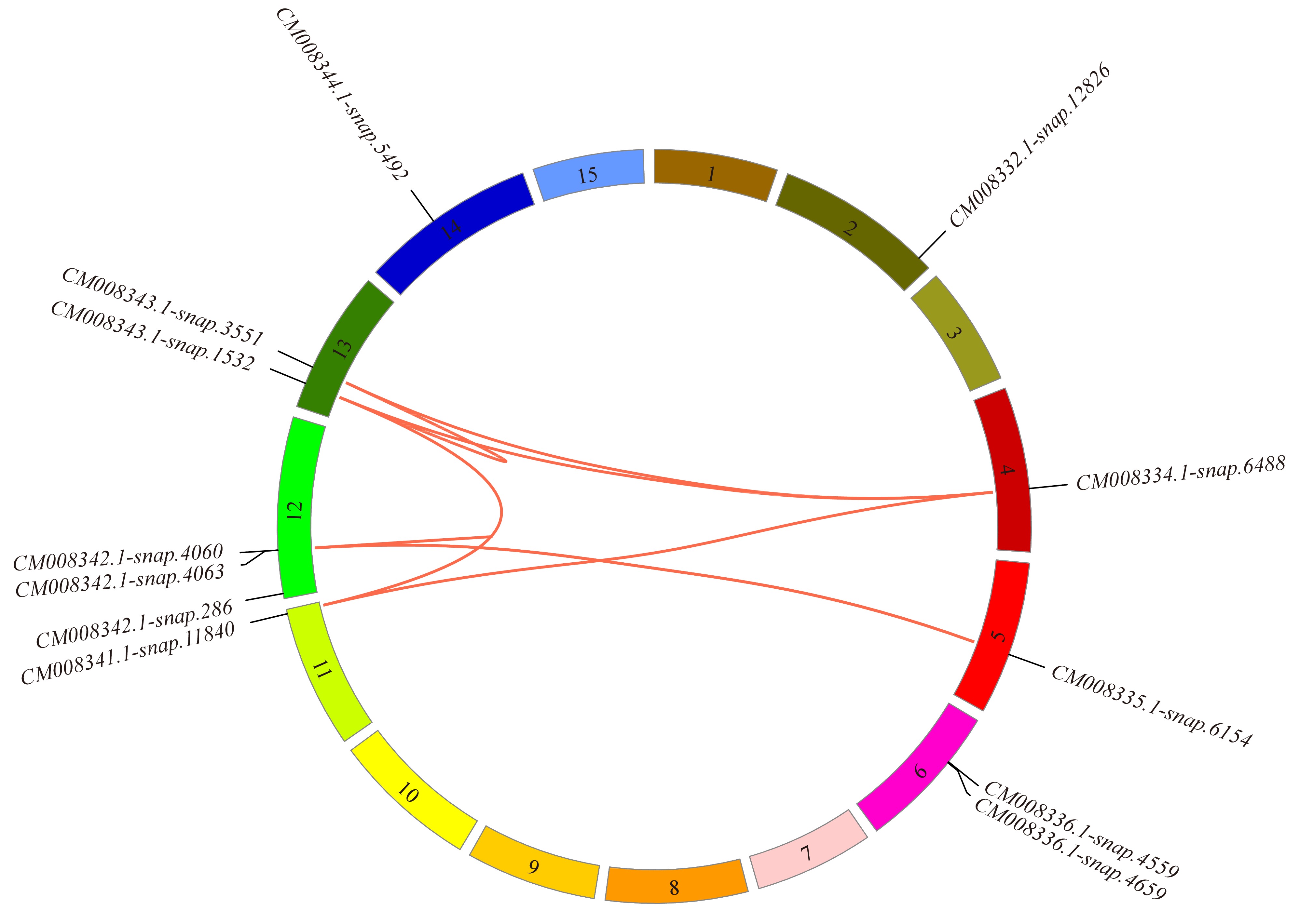
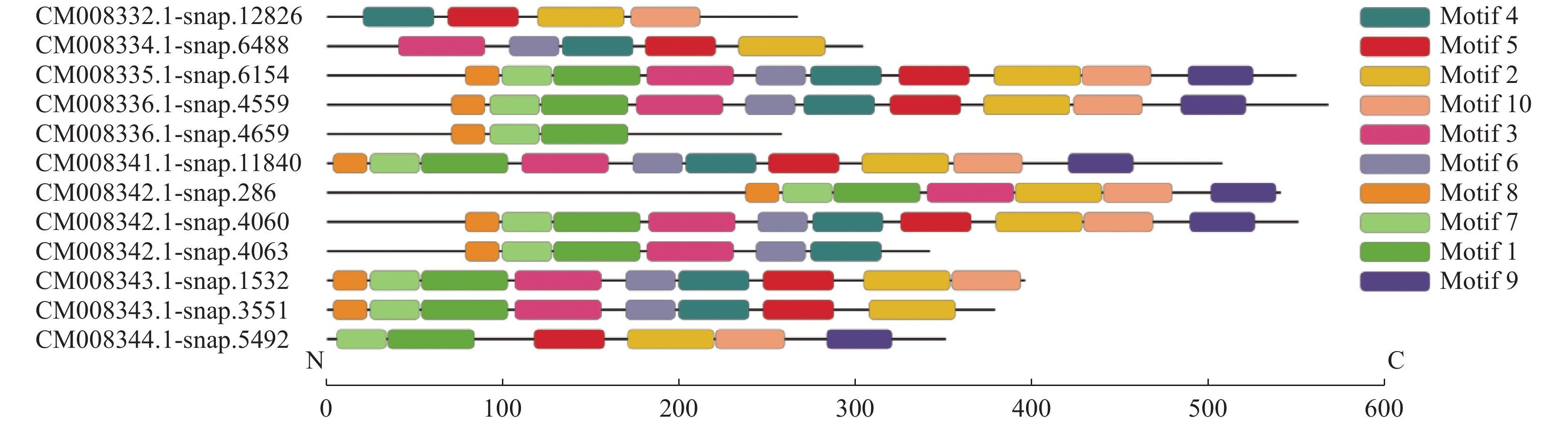
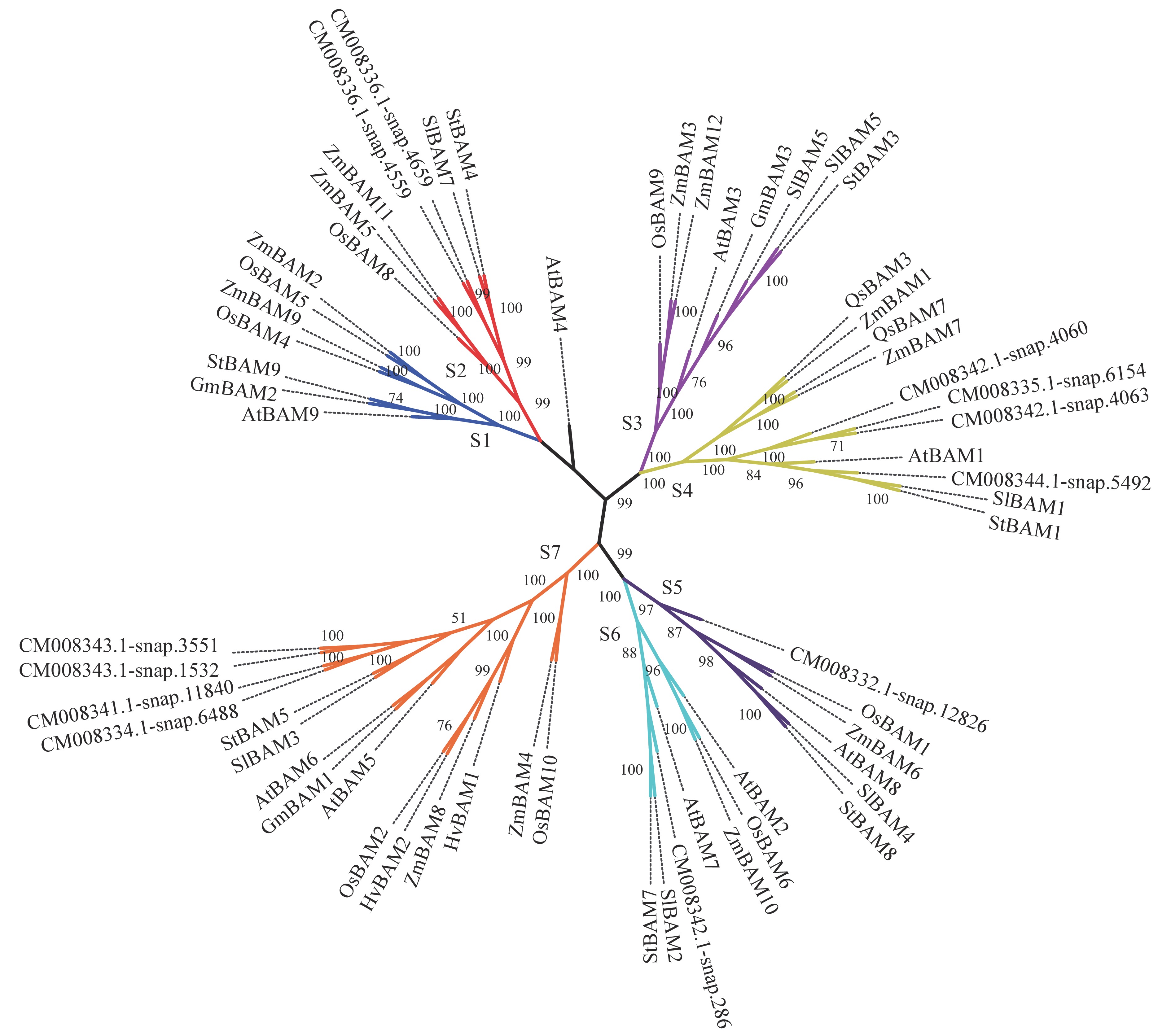
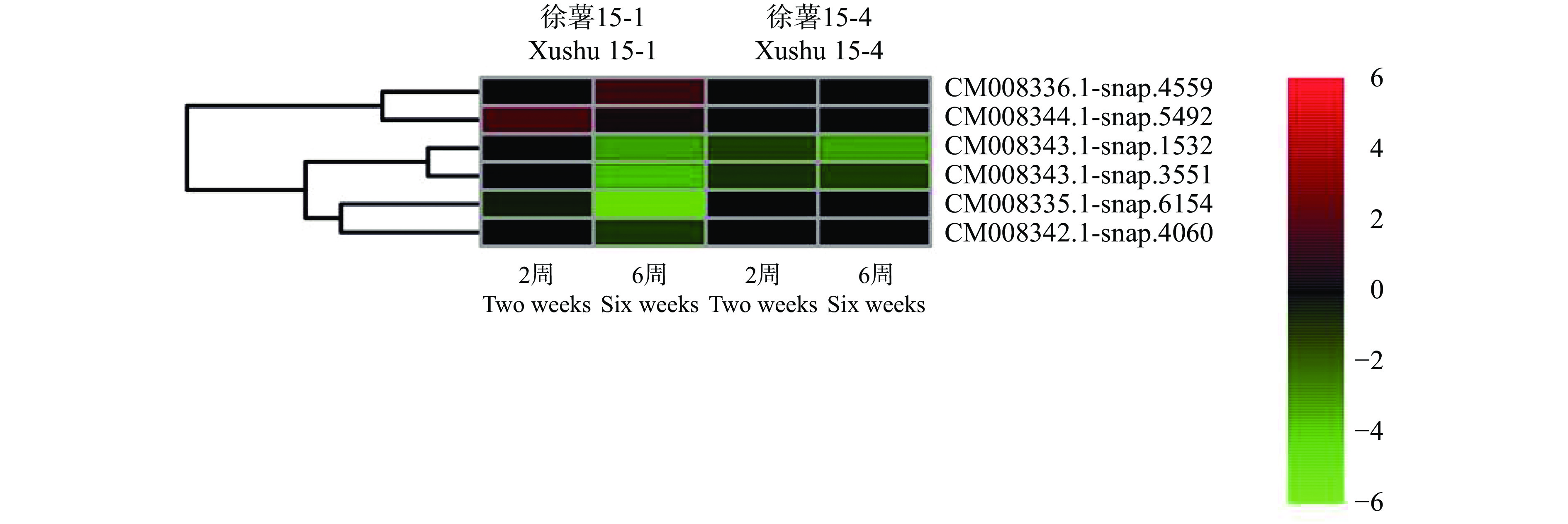
结果12个β−淀粉酶基因分布于甘薯第2、4、5、6、11、12、13和14号染色体上,含有8个具有潜在重复关系的基因对。多重比对和功能结构域搜索结果显示,甘薯β−淀粉酶家族的氨基酸序列中含有3个保守性较高的区域和10个保守基序。甘薯与其他物种β−淀粉酶蛋白的系统进化分析结果显示,62个β−淀粉酶家族成员被分为S1~S7等7个亚组,甘薯β−淀粉酶家族成员主要分布在S2、S4、S5、S6以及S7亚组中,且大多与拟南芥、马铃薯以及番茄的β−淀粉酶为同一分支。转录组测序数据分析结果显示,在低温贮藏的过程中有6个甘薯β−淀粉酶基因表达量出现变化,其中‘徐薯15-1’有2个基因上调表达、4个基因下调表达,‘徐薯15-4’仅有2个基因下调表达。
结论β−淀粉酶是一类关键的淀粉水解酶,在甘薯生长发育和薯块贮藏阶段淀粉降解为还原糖的过程中发挥着重要作用,本研究鉴定得到的12个甘薯β−淀粉酶基因序列信息为进一步探讨甘薯β−淀粉酶基因家族的生物学功能提供数据参考。
Abstract:ObjectiveTo mine the sequence information of the β-amylase gene family of Ipomoea batatas genome, and analyze the structure and function of genes.
MethodBased on the whole genome sequence data of I. batatas cultivar ‘Taizhong 6’, the bioinformatic methods were applied to analyze the identified 12 members of the β-amylase gene family and conduct the domain conservation analysis, chromosomal localization, screening of potential duplication genes, conservative motif analysis, phylogenetic tree construction. The gene expression under low temperature stress was analyzed using the transcriptomics data.
ResultTwelve β-amylase genes were located on chromosomes No. 2, 4, 5, 6, 11, 12, 13 and 14 of I. batatas, and eight pairs showed potential duplication relationship. Multiple sequence alignment and functional domain search indicated that there were three highly conserved domains and 10 conservative motifs in the amino acid sequences of I. batatas β-amylase family. Phylogenetic trees of β-amylase proteins in I. batatas and other species showed that 62 β-amylase family members were divided into seven subgroups of S1−S7. The β-amylases of I. batatas were mainly distributed in the subgroups of S2, S4, S5, S6 and S7, most of which belonged to the same branches with Arabidopsis thaliana, Solanum tuberosum and S. lycopersicum. The results of transcriptomics data showed that six β-amylase genes expressed differentially during the low temperature storage period, of which two were up-regulated and four were down-regulated in ‘Xushu 15-1’, while only two genes were down-regulated in ‘Xushu 15-4’.
ConclusionThe β-amylases are a key class of starch hydrolyzing enzymes that play important roles in the degradation of starch into reducing sugars during the process of I. batatas growth, development and tuber storage stages. The sequences of the identified 12 sweet potato β-amylase genes provide data reference for further study on the biological functions of I. batatas β-amylase gene family.
-
Keywords:
- Ipomoea batatas /
- β-amylase /
- gene family /
- systematic evolution /
- differential expression analysis
-
随着农业机械化水平的不断提高,越来越多的新技术应用于农业。针对农作物病虫害防治,利用农业机器人实现自主作业,可以解决从事农业人口比例逐年降低、劳动者工作强度大等问题,提高作业效率和质量,降低生产成本[1-2]。自动导航是农业机器人实现自主作业重要的一步,目前应用于农业的自动导航技术主要有RTK-GPS导航、机器视觉导航、地磁导航和无线电导航等,但GPS导航容易受到农田作物遮挡影响,信号不稳定,而多传感器融合导航具有实时性好、价格低廉、适用范围广等优点,因此相机与激光雷达融合导航已经逐步成为国内外研究热点。
Søgaard等[3]通过计算图像中玉米的重心代替分割步骤,从而减少了图像处理的耗时;杨洋等[24]利用动态感兴趣区域去除图像中干扰部分,采用最小二乘法直线拟合获取了玉米导航线。常昕等[4]以激光雷达点云数据修正跟踪,解决图像中受光照、阴影和背景干扰的问题。胡丹丹等[5]以玉米收割机器人为载体,采用机器视觉获取玉米植株离散点特征,基于Hough变换进行直线拟合得到导航基准线。Marchant等[6]通过植物的颜色特征和相机标定对作物行进行Hough变换处理,提高了检测效率。梁习卉子等[7]对区域内各色域分量权值进行调整,强调绿色像素而淡化红色和蓝色像素,去除枯草、阴影对检测结果造成的干扰。
目前农业机器人在田间行走主要依靠单一传感器识别,利用相机获取图像数据,将植株绿色像素投影曲线峰值特性,通过算法拟合峰值点可以获得导航基准线。俞毓锋等[8]结合激光雷达的深度信息和图像的颜色纹理信息,构建在时序帧间的特征点匹配关系,终将定位问题转化为特征点对的加权重投影误差优化问题。但实际上存在光照和地面起伏等因素,在信息融合过程中容易出现信息不匹配问题,降低识别准确率。宋宇等[9]提出特征点处植株绿色特征列像素累加值大于左右相邻像素累加值,通过设定峰值点距离阈值来获取准确根茎定位点,最终利用拟合定位点来获取导航线。但实际上玉米田中普遍存在缺苗、一穴多株现象,并且相机拍摄角度或焦距成像不同,在设定距离阈值进行筛选根茎定位点时会出现错误,降低拟合导航线的准确率。冯娟等[10]利用特征点附近像素值梯度变化程度,通过对形态学处理后的图像进行扫描获取土壤与根茎交界线。该方法在根茎附近有杂草干扰的情况时,会在相邻根茎定位点之间产生伪特征点,对导航线拟合产生严重影响。
本文针对单一传感器无法有效识别导航线问题,提出一种基于离散因子的多传感器特征检测和数据融合方法,离散因子是由激光雷达和相机采集到的孤立的信息点集,因此构建多传感器目标识别的相对一致性和加权一致性函数,根据多传感器特征检测的权重以及相关的一致性函数,建立多传感器特征识别的数据融合支持度数学模型以获取特征点。最后利用正确的玉米特征中心点进行拟合,可以获得导航基准线,为农业机械在玉米田间行走提供导航基准。
1. 数据采集和预处理
1.1 图像采集与特征提取
视觉系统采集到的3~5叶期玉米植株图片为RGB彩色图像(图1),图像分辨率为640像素×480像素。测试环境为阴天,环境中有石子、树枝及行人等干扰因素,用
$ 2G - B - R $ 可以提取绿色特征分量[11],采用超绿化算法处理后,灰度图像仍为三维向量数据组,因此需要对图像进行二值化处理,可以有效减少计算量,提高效率。使用最大类间方差法[12-14](OTSU)以及形态学滤波中的开运算[15-17]进行二值化处理,处理效果如图2所示,该算法有效屏蔽了干扰因素并提取植物特征,同时对每个玉米植株连通域计算其重心位置并保存备用。1.2 点云数据采集与聚类分析
采用Velodyne Lidar VLP-16PUCK的激光雷达对视觉采集的同一目标进行数据扫描。激光雷达扫描的数据以点云形式存储,每个扫描点包含该点的三维坐标和反射率等信息。但由于复杂环境存在干扰以及设备存在精度的缺陷,原始点云数据存在伪特征点。针对原始数据中的伪特征点,采用基于半径滤波去噪的方法进行剔除,即设置规定半径范围内点云的最小数量,当给定点周围点云数量大于给定值时保留该点,反之则剔除该点,依序迭代可获得最密集的点,从而去除原始数据中的伪特征点,处理后的点云俯视图如图3所示。
预处理后的玉米植株点云数据依然存在模糊、分散等问题,因此进行部分聚类操作,导出最大密度相连的样本集合,即为最终聚类的一个类别,从而加强玉米植株的特征点云数据。采用DBSCAN[18-19]聚类算法进行点云数据处理,通过将紧密相连的样本划为一类,这样就得到一个聚类类别,通过将所有各组紧密相连的样本划为各个不同的类别,则可以获得最终所有聚类类别的结果。经过DBSCAN聚类操作处理的点云数据如图4所示,玉米植株的特征被划分得更加明显,解决了点云数据杂乱、噪音多等问题。
2. 多传感器数据融合
基于相机和激光雷达的融合导航识别算法是将预处理后点云数据的反射率信息加入到预处理后图像数据中,降低光照不足、遮挡等干扰对可通行区域提取的影响,提高识别精度,算法流程如图5所示。
2.1 相机与激光雷达联合标定
为达到数据融合的目的,首先要将相机和激光雷达数据进行空间匹配,因此需要对相机和激光雷达进行联合标定[20-21],即得到两者之间的数据转换关系,找到同一时刻激光雷达点云数据对应图像中的像素点。
建立激光雷达坐标系
${O_{\rm{L}}} - {X_{\rm{L}}},{Y_{\rm{L}}},{Z_{\rm{L}}}$ ;相机坐标系${O_{\text{C}}} - {X_{\rm{C}}},{Y_{\rm{C}}},{Z_{\rm{C}}}$ ;图像坐标系$ O - x,y $ ;像素坐标系$ O - u,v $ ,如图6所示。假设$ {\theta _X},{\theta _Y},{\theta _Z} $ 为激光雷达坐标系相对于相机坐标系在$ x,y,z $ 坐标轴方向的转角,转动矩阵为R;$ {\boldsymbol{T}}({t_1},{t_2},{t_3}) $ 为相机坐标系到激光雷达坐标系的平移向量;$ ({u_0},{v_0}) $ 为图像坐标系原点在像素坐标系下的位置。传感器融合的关键就是将物体在激光雷达下的坐标转化为像素坐标,从而达到激光点云与图像融合的目的。因此,假设空间有一点P在激光雷达坐标系下的坐标为
$({X_{\rm{L}}},{Y_{\rm{L}}},{Z_{\rm{L}}})$ ,在相机坐标系下的坐标为$({X_{\rm{C}}},{Y_{\rm{C}}},{Z_{\rm{C}}})$ ,在图像坐标系下的坐标为$ (x,y) $ ,在像素坐标系下的坐标为$ (u,v) $ ,t1,t2,t3分别为相机到激光雷达$ x,y,z $ 方向的距离。具体过程为:将P点在激光雷达坐标系下的坐标转为相机坐标,再从相机坐标转为图像坐标,最后从图像坐标转为像素坐标。1) 从激光雷达坐标系到相机的坐标系转化为:
$$ \left[ {\begin{array}{*{20}{c}} {{X_{\rm{C}}}} \\ {{Y_{\rm{C}}}} \\ {{Z_{\rm{C}}}} \end{array}} \right] = {\boldsymbol{R}}\left[ {\begin{array}{*{20}{c}} {{X_{\rm{L}}}} \\ {{Y_{\rm{L}}}} \\ {{Z_{\rm{L}}}} \end{array}} \right] + \left[ {\begin{array}{*{20}{c}} {{t_1}} \\ {{t_2}} \\ {{t_3}} \end{array}} \right] ,$$ (1) 式中,
$$ \begin{split} &{\boldsymbol{R}} = {{\boldsymbol{R}}_x}{{\boldsymbol{R}}_y}{{\boldsymbol{R}}_z} = \left[ {\begin{array}{*{20}{c}} 1&0&0 \\ 0&{\cos {\theta _x}}&{ - \sin {\theta _x}} \\ 0&{\sin {\theta _x}}&{\cos {\theta _x}} \end{array}} \right]\\ &\quad\left[ {\begin{array}{*{20}{c}} {\cos {\theta _y}}&0&{\sin {\theta _y}} \\ 0&1&0 \\ {\sin {\theta _y}}&0&{\cos {\theta _y}} \end{array}} \right]\left[ {\begin{array}{*{20}{c}} {\cos {\theta _z}}&{ - \sin {\theta _z}}&0 \\ {\sin {\theta _z}}&{\cos {\theta _z}}&0 \\ 0&0&1 \end{array}} \right]。 \end{split} $$ (2) 2) 从相机坐标系到图像坐标系的转换为:
$$ Z_{\rm{C}}\left[ {\begin{array}{*{20}{c}} x \\ y \\ 1 \end{array}} \right] = \left[ {\begin{array}{*{20}{c}} f&0&0 \\ 0&f&0 \\ 0&0&1 \end{array}} \right]\left[ {\begin{array}{*{20}{c}} {{X_{\rm{C}}}} \\ {{Y_{\rm{C}}}} \\ {{Z_{\rm{C}}}} \end{array}} \right] ,$$ (3) 式中,
$ f $ 是相机的焦距。3) 从图像坐标到像素坐标的转换为:
$$ \left[ {\begin{array}{*{20}{c}} u \\ v \\ 1 \end{array}} \right]{\text{ = }}\left[ {\begin{array}{*{20}{c}} {\dfrac{1}{{d_x}}}&0&{{u_0}} \\ 0&{\dfrac{1}{{d_y}}}&{{v_0}} \\ 0&0&1 \end{array}} \right]\left[ {\begin{array}{*{20}{c}} x \\ y \\ 1 \end{array}} \right], $$ (4) 式中:
$d_x$ 为单个像素点在像素坐标x方向的长度,单位mm;$d_y$ 为单个像素点在像素坐标y方向的长度,单位mm;u0为像素坐标系和图像坐标在x方向的平移偏量,v0为像素坐标系和图像坐标在y方向的平移偏量,单位mm。2.2 数据融合模型
在相机和激光雷达对地面作物进行识别的过程中,2种传感器会探测到地面作物的不同特征,通过信号处理将2个传感器收集到的目标相应特征输出,其输出数据对目标识别结果的支持程度以及输出数据的一致性函数[22]是系统准确识别作物的重要依据。
假设
$ \alpha$ 是需要识别的作物特征组成的集合,则将$ \alpha$ 中一个模糊集定义为一个隶属函数:$$ {m}_{j}(w)=1-\dfrac{|E-{\mu }_{j}|}{2{\sigma }_{j}}\text{;}{m}_{j}(w): \alpha \to [0,1],w\in \alpha ,$$ (5) 式中,
$ {m_j}(w) $ 表示第$ j $ 个传感器对模糊命题的隶属度,E表示地面目标特征,$ {\mu _j} $ 、$ {\sigma _j} $ 分别表示地面目标特征的均值和标准差,w表示识别的作物目标特征,不同的传感器获取作物的不同特征。因为采用多传感器进行作物识别,所以采用关联性结果来表示2个传感器对同一个支持模糊命题的支持程度。具体操作是先假设2个传感器
$ i $ 和$ j $ 对模糊命题的共同支持程度,即$ {m_i}(w) $ 和$ {m_j}(w) $ 之间的关联性,称之为一致性函数:$$ {S_{ji}} = \dfrac{{{m_j}(w) \wedge {m_i}(w)}}{{{m_j}(w) \vee {m_i}(w)}} = \dfrac{{\min \left\{ {{m_j}(w),{m_i}(w)} \right\}}}{{\max \left\{ {{m_j}(w),{m_i}(w)} \right\}}}, $$ (6) 式中,
$ \wedge $ 表示取两者中数值较小者,$ \vee $ 表示取两者中数值较大者。然后进行判别,当2个传感器对模糊命题的支持度相同时,取
$ {S_{ji}} = 1 $ ;当2个传感器对模糊命题的支持度将近时,表明2个传感器观测值相互支持度高,则$ 0 < {S_{ji}} < 1 $ ;当2个传感器对模糊命题的支持度相差较大,表明2个传感器观测值相互支持度低,甚至相互背离,因此对其信息不采用,则取$ {S_{ji}} = 0 $ 。如果出现2个传感器支持度持续不同的结论,就需要进一步判别这2个传感器是否出现故障。例如采用一组随机变量
$ {E_1},{E_2}, \cdots ,{E_h} $ 来表示通过多传感器获得地面目标的h个特征,通过随机变量的标准差与均值计算隶属度$ {m_j}(w) $ ,从而计算传感器对模糊命题的支持度$ {S_{ji}} $ 。假设随机变量的标准差和均值如表1所示。
表 1 玉米植株特征随机变量及其标准差和均值Table 1. Random variables, standard deviations and means of maize plant characteristics传感器编号
Sensor number目标1 Target 1 目标2 Target 2 目标3 Target 3 随机变量
Random variable标准差
Standard deviation均值
Mean标准差
Standard deviation均值
Mean标准差
Standard deviation均值
Mean1 50.0 246.0 68.0 372.0 23.0 289.0 318.0 2 1.1 1.8 0.6 2.6 0.7 2.9 3.4 将数值带入公式(5)计算的隶属度
$ {m_j}(w) $ 结果如表2所示。表 2 各传感器对不同目标的隶属度Table 2. Membership of each sensor to different targets传感器编号
Sensor number目标1
Target 1目标2
Target 2目标3
Target 31 0.28 0.59 0.37 2 0.27 0.33 0.64 根据公式(6)可以求得
$ {S}_{12}(目标1)\text{=}0.964 $ ,$ {S}_{12} (目标2)\text{=}0.559 $ ,$ {S}_{12}(目标3)\text{=}0.578 $ 。根据相机和激光雷达目标识别输出的一致性函数,构建传感器采集的所有特征对模糊命题的支持度一致性矩阵:
$$ {\boldsymbol{M}} = \left[ {\begin{array}{*{20}{c}} 1&{{S_{12}}}&{...}&{{S_{1i}}}&{...}&{{S_{1l}}} \\ \vdots & \vdots &{}& \vdots &{}& \vdots \\ {{S_{j1}}}&{{S_{j2}}}&{...}&{{S_{ji}}}&{...}&{{S_{jl}}} \\ \vdots & \vdots &{}& \vdots &{}& \vdots \\ {{S_{l1}}}&{{S_{l2}}}&{...}&{{S_{li}}}&{...}&1 \end{array}} \right] ,$$ (7) 式中,
$ l $ 表示采集到的目标的特征数量。利用上式中传感器采集的所有特征对模糊命题的支持度一致性计算出平均一致性,通过归一化处理,计算出第
$ j $ 个传感器作物识别输出的相对一致性函数:$$ M'{'_j} = \dfrac{{M{'_j}}}{{\displaystyle\sum\limits_{i = 1}^l {\dfrac{1}{{l - 1}}\displaystyle\sum\limits_{i = 1,i \ne j}^l {{S_{ji}}} } }} = \dfrac{{\dfrac{1}{{l - 1}}\displaystyle\sum\limits_{i = 1,i \ne j}^l {{S_{ji}}} }}{{\displaystyle\sum\limits_{i = 1}^l {\dfrac{1}{{l - 1}}\displaystyle\sum\limits_{i = 1,i \ne j}^l {{S_{ji}}} } }}, $$ (8) 式中,
$ M{'_j} $ 为第$ j $ 个传感器作物识别输出的一致性函数。通过一致性函数,即可将不同物理量的传感器数据进行匹配,保留图像特征重心和激光点云共同的作物特征点,剔除伪特征点。由于测试环境的目标干扰,行人、测试区域动态变化性等不确定因素,利用2种传感器采集目标的多类型特征数据,并依据获得的目标离散信息点集之间的离散状态确定多传感器的当前权重,离散因子根据不同时间段、多测试区域传感器测量的目标信息进行融合,有效避免了传感器权重选取的经验性和局限性;同时,将传感器当前权重引入到多传感器目标识别的平均加权一致性函数数学模型,通过计算处理给出多传感器目标识别的相对加权一致性计算函数;结合多传感器目标识别的相对一致性、多传感器目标识别的当前权重,构建基于离散因子的多传感器目标识别的数据融合支持度计算模型。
3. 导航线提取
将预处理后的图像与激光雷达特征点进行融合,剔除了伪特征点,对其获得的特征点进行直线拟合即可得到导航基准线。为减少异常点对作物特征点进行直线拟合的影响,引入随机采样一致(Random sample consensus, RANSAC)算法[23],通过反复选择数据集估计出模型,一直迭代到估计出较好的模型。具体步骤为:
1) 首先筛选可以估计出模型的最小数据,使用这个数据计算出数据模;
2) 将所有数据带入这个模型,计算出组成模型参数数据的数量即内点数量;
3) 比较当前模型和之前推出最好模型的内点数量,记录最大内点数的模型参数;
4) 重复上述步骤,直到迭代结束或者当前模型内点数量达到要求。
此时需要对迭代次数进行选择,假设内点在数据中的占比为t。则:
$$ {t} = \dfrac{{{n_{\rm{inliers}}}}}{{{n_{\rm{inliers}}} + {n_{\rm{outliers}}}}}, $$ (9) 式中,
${n_{{\rm{inliers}}}}$ 为内点数量,${n_{{\rm{outliers}}}}$ 为外点数量。在每次计算模型使用N个点的情况下,选取的点至少有1个不是内点的概率是
$ 1 - {t^N} $ ,在迭代k次的情况下,$ {(1 - {t^N})^k} $ 为模型至少1次没有采样到内点时计算出模型的概率,即计算出错误模型的概率。那么能采样到正确的N个点并计算出正确模型的概率为:$$ P = 1 - {(1 - {t^N})^k} ,$$ (10) 通过上式,可以求得:
$$ k = \dfrac{{\lg (1 - P)}}{{\lg (1 - {t^n})}} ,$$ (11) 内点占比t通常为先验值,P是我们希望RANSAC算法得到正确模型的概率,此时可以求出最优的迭代次数k。通过RANSAC算法分别得到左、右导航基准线,2条导航基准线的角平分线即玉米田导航线,如图7所示。设左导航基准线斜率为
$ \;{\beta _{\text{1}}} $ ,右导航基准线斜率为$ \;{\beta _{\text{2}}} $ ,则导航线斜率$\; \beta $ 满足:$$ \dfrac{{\beta - {\beta _1}}}{{1 + \beta {\beta _1}}} = \dfrac{{{\beta _{\text{2}}} - \beta }}{{1 + \beta {\beta _2}}} 。$$ (12) 4. 试验及可靠性分析
试验采用英特尔Pentium(R) CPU G3250 @ 3.20 GHZ、4 GB内存、Windows 7(64位)操作系统的计算机,在Python3.7(Anaconda)的集成开发环境下编程完成。试验中的农业机器人宽45 cm、长60 cm,装有森云智能SG2-AR0233C-5200-GMSL2相机、16线激光雷达、4 G模块等多种传感器,采集前方环境和玉米植株信息,如图8所示。
为了更精确地分析该方法在玉米田中的植保机器人行走区域识别精度,本试验通过布置标定好行距和株距的玉米植株,模拟真实场景开展精度分析试验。结果表明,本文算法可以准确提取导航线。由于实时性和正确性是导航线提取的正确指标,表3统计了本文算法不同传感器对随机900帧图像的平均处理时间及正确率,通过人工标定图像最合理的导航线,定义人工与本文算法提取的导航线之间的夹角为误差角,误差角超过5°时,视为导航线错误。
表 3 不同传感器性能评价指标Table 3. Performance evaluation index of different sensors传感器
Sensor平均处理时间/ms
Average processing time处理帧数
No. of processing frames正确帧数
No. of correct frames正确率/%
Correct rate平均误差角度/(°)
Mean error angle相机 Camera 49.76 300 272 90.67 3.457 三维激光雷达
3D LiDAR58.15 300 278 92.67 2.670 传感器融合
Sensor fusion95.62 300 286 95.33 1.595 由表3可知,分别用相机、三维激光雷达和传感器融合的识别方法,平均处理时间逐渐增加,正确率也随之增加,当将相机和激光雷达融合时,平均处理时间和误差角度均满足要求。将该算法与候补定位点二次判别算法、动态迁移算法进行对比,本算法单帧平均处理时间为95.62 ms,优于候补定位点二次判别算法的单帧平均处理时间200 ms[9]和动态迁移算法的单帧平均处理时间97.56 ms[24],具有更高的实时性;本算法正确率为95.33%,优于候补定位点二次判别算法的正确率90%[9]和动态迁移算法的正确率95%[24],为农业机械的视觉导航提供了可靠保障。
5. 结论
本文基于半径的滤波方法依序迭代剔除点云数据噪音,利用DBSCAN算法进行点云数据聚类,导出最大密度相连的样本集合,采用激光雷达和相机信息融合的方式进行可通行区域提取,在图像数据中加入点云数据的反射率信息,降低了阴影、遮挡对导航线提取的影响。
根据多时间段、多空间区域传感器采集的目标特征量进行系统建模分析,并依据获得的目标特征值之间的离散因子确定多传感器权重选择,根据权重引入多传感器目标识别的平均加权一致性函数,获得具有冗余性的数据融合支持度模型。最终试验平均处理时间仅为95.62 ms,正确率高达95.33%,提高了检测结果的稳定性和准确性,为农业机器人在农田环境中作业提供了可靠的导航路径。
-
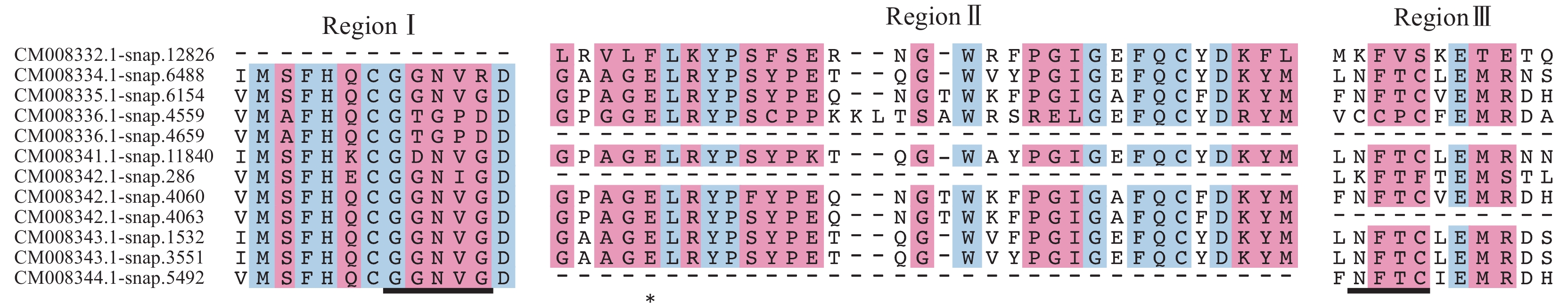
图 1 甘薯β−淀粉酶保守结构域分析
蓝色阴影部分表示该位点氨基酸保守性为100%,红色阴影部分表示该位点氨基酸保守性在70%以上;统计时各区域内由空位所代替的序列不纳入计算;Region I画横线部分表示flexible loop,Region II“*”标注处表示催化活性位点,Region III画横线部分表示Inner loop
Figure 1. Analysis of the conserved domains of β-amylase in Ipomoea batatas
The blue shaded part indicates that the amino acid conservation of the site is 100%, and the red shaded part indicates that the amino acid conservation of the site is over 70%; The series replaced by vacancies in each region are excluded in the calculation; The horizontal line in Region I represents the Flexible loop, the “*” in Region II shows the catalytic active site, and the horizontal line in Region III denotes the Inner loop
表 1 甘薯β−淀粉酶基因序列信息1)
Table 1 The sequence information of β-amylase gene in Ipomoea batatas
基因名称
Gene name外显子
Exon链
Strand长度/bp
LengthCM008332.1-snap.12826 45177690-45177779; 45176575-45176835; 45176174-45176392; − 798 45175888-45176091; 45175660-45175665; 45174982-45174999 CM008334.1-snap.6488 29784343-29784606; 29784692-29784856; 29785241-29785504; + 909 29786090-2978630 CM008335.1-snap.6154 26986968-26987510; 26986519-26986728; 26986320-26986430; − 1 647 26985282-26986064 CM008336.1-snap.4559 14557970-14558494; 14560135-14560344; 14560435-14560551; + 1 701 14560653-14561441; 14561707-14561766 CM008336.1-snap.4659 14866852-14867396; 14867730-14867844; 14868220-14868330 + 771 CM008341.1-snap.11840 40434808-40434870; 40433953-40434363; 40433695-40433859; − 1 521 40432752-40433015; 40432065-40432280; 40431587-40431988 CM008342.1-snap.286 1511146-1511589; 1512051-1512279; 1512390-1512481; + 1 623 1512728-1512931; 1513310-1513504; 1514282-1514491; 1514642-1514861; 1515345-1515373 CM008342.1-snap.4060 14068276-14068818; 14069053-14069265; 14069351-14069461; + 1 650 14069709-14070491 CM008342.1-snap.4063 14080849-14081390; 14081696-14081906; 14082004-14082114; + 1 023 14082365-14082523 CM008343.1-snap.1532 7100890-7100952; 7101137-7101535; 7101690-7101854; + 1 185 7101930-7102196; 7102266-7102487; 7102713-7102781 CM008343.1-snap.3551 12195237-12195299; 12195510-12195908; 12196093-12196257; + 1 134 12196351-12196617; 12196701-12196940 CM008344.1-snap.5492 22226248-22226507; 22227548-22228337 + 1 050 1) 基因名称由以“-”分隔的染色体序列文件名和基因编号2部分组成,例如CM008332.1-snap.12826表示NCBI中‘泰中6号’基因组[9]的染色体序列文件CM008332.1.fasta(第2号染色体)上根据snap预测[10]的编号为snap.12826的基因;基因由外显子组成,表中外显子的位置区间均以正义链坐标为准,链为“+”的基因取正义链的序列,链为“−”的基因取正义链反向互补的序列,外显子排列顺序按照其在基因中出现的顺序,拼接外显子即可得到基因全长序列
1) The gene name consists of the name of chromosome sequence file and the gene number that are separated by the symbol “-”, for example, the CM008332.1-snap.12826 indicates the gene numbered snap.12826, which is predicted with snap[10] in the chromosome sequence file CM008332.1.fasta (chromosome No. 2) of the ‘Taizhong 6’ genome[9] in NCBI; The genes are made up of exons, in the table, the positional interval of the exon is set according to the sense strand, the sequence of gene with strand “+” is taken from the sense strand and the sequence of gene with the strand “−” is taken from the reverse compliment of the sense strand, the exons are arranged corresponding to the sequential position within the gene, the full-length gene can be obtained via merging the exons表 2 甘薯β−淀粉酶保守基序氨基酸组成特征1)
Table 2 Amino acid composition of the conservative motifs of the Ipomoea batatas β-amylase
基序 Motif 氨基酸组成 Amino acid composition 结构域 Structural domain Motif 1 VDVWWGLVEKDSPREYNWAGYSELLQLAKKHGLKVQA VMSFHQCGGNVGD Glyco_hydro_14 Motif 2 GIHWWYGTRSHAAELTAGYYNTRGRDGYLPIARMLARHGAT LNFTCLEMR Glyco_hydro_14 Motif 3 IPLPRWVLEEGDKNPDIFYTDRAGRRNYEYLSLGVDNQPLFKGRTPLQMY Glyco_hydro_14 Motif 4 WVFPGIGEFQCYDKYMVASWKGAAEAAGHPEWGMPGPTDAG Glyco_hydro_14 Motif 5 TEFFRENGTYNTDYGKFFLTWYSQMLIIHGDRILQEANKVF Glyco_hydro_14 Motif 6 FLLGGTIVDIQVGM GPAGELRYPSYPETQ — Motif 7 VKMDHTMNRKKAMEVSLQALKSAGVEGVM — Motif 8 IPKMMSRSRGVPVFVMLPLD — Motif 9 MCAFTYLRMNPELFEARNWIQFVGFVKKMKEGEQRREC SCOP domain d1byb_ Motif 10 DHEQPQHAQCAPEKLVWQVLLATWEARVPLAGENALPRYD Glyco_hydro_14 1) 下划线表示与图1对应的保守区域部分;“—”表示未匹配到相关信息
1) The underlines indicate the conservative regions corresponding to Fig. 1; “—” indicates no relevant information is matched表 3 β−淀粉酶蛋白数据库查询编号
Table 3 The database query numbers of β-amylase proteins
物种
Species蛋白名称
Protein name数据库查询编号
Database query number物种
Species蛋白名称
Protein name数据库查询编号
Database query number水稻
Oryza sativaOsBAM1 LOC_Os02g03690.1 拟南芥
Arabidopsis thalianaAtBAM1 AT3G23920.1 OsBAM2 LOC_Os07g35940.1 AtBAM2 AT4G00490.1 OsBAM3 LOC_Os03g04770.1 AtBAM3 AT4G17090.1 OsBAM4 LOC_Os07g47120.1 AtBAM4 AT5G55700.1 OsBAM5 LOC_Os03g22790.1 AtBAM5 AT4G15210.1 OsBAM6 LOC_Os09g39570.1 AtBAM6 AT2G32290.1 OsBAM7 LOC_Os10g32810.1 AtBAM7 AT2G45880.1 OsBAM8 LOC_Os01g13550.1 AtBAM8 AT5G45300.1 OsBAM9 LOC_Os10g41550.1 AtBAM9 AT5G18670.1 OsBAM10 LOC_Os07g35880.1 马铃薯
Solanum tuberosumStBAM1 PGSC0003DMP400002800 玉米
Zea maysZmBAM1 NP_001148159.2 StBAM3 PGSC0003DMP400035625 ZmBAM2 NP_001151271.2 StBAM4 PGSC0003DMP400021443 ZmBAM3 XP_008658465.1 StBAM5 PGSC0003DMP400045472 ZmBAM4 NP_001168436.1 StBAM7 PGSC0003DMP400000367 ZmBAM5 NP_001130896.1 StBAM8 PGSC0003DMP400041754 ZmBAM6 AQK60892.1 StBAM9 PGSC0003DMP400018848 ZmBAM7 NP_001354441.1 番茄
Solanum lycopersicumSlBAM1 A0A3Q7I9I2 ZmBAM8 NP_001105496.2 SlBAM2 A0A3Q7EKY0 ZmBAM9 NP_001170007.1 SlBAM3 A0A3Q7HEM6 ZmBAM10 NP_001337631.1 SlBAM4 A0A3Q7IDQ8 ZmBAM11 NP_001132696.1 SlBAM5 A0A3Q7IEI0 ZmBAM12 XP_035818275.1 SlBAM6 A0A3Q7HUA2 大豆
Glycine maxGmBAM1 P10538.3 SlBAM7 A0A3Q7HV50 GmBAM2 CAI39245.1 大麦
Hordeum vulgareHvBAM1 CAC16789.1 GmBAM3 CAI39244.1 HvBAM2 AAX37357.1 -
[1] 张勇为, 张义正, 谭文芳, 等. 甘薯贮藏期间淀粉酶种类变化及其部分性质分析[J]. 四川大学学报(自然科学版), 2018, 55(1): 197-200. [2] TODA H, NITTA Y, ASANAMI S, et al. Sweet potato β-amylase: Primary structure and identification of the active-site glutamyl residue[J]. European Journal of Biochemistry, 1993, 216(1): 25-38. doi: 10.1111/j.1432-1033.1993.tb18112.x
[3] 孙俊良, 梁新红, 贾彦杰, 等. 植物β−淀粉酶研究进展[J]. 河南科技学院学报(自然科学版), 2011, 39(6): 1-4. [4] LI H S, ÔBA K. Major soluble proteins of sweet potato roots and changes in proteins after cutting, infection, or storage[J]. Agricultural and Biological Chemistry, 2014, 49(3): 737-744.
[5] NAKAMURA K, OHTO M A, YOSHIDA N, et al. Sucrose-induced accumulation of beta-amylase occurs concomitant with the accumulation of starch and sporamin in leaf-petiole cuttings of sweet potato[J]. Plant Physiology, 1991, 96(3): 902-909. doi: 10.1104/pp.96.3.902
[6] 梁新红, 李英, 孙俊良, 等. β−淀粉酶酶解甘薯淀粉条件分析[J]. 食品工业科技, 2014, 35(7): 178-181. [7] 陈显让, 李红兵, 康乐, 等. 甘薯块根膨大后期β−淀粉酶和淀粉含量相关性分析[J]. 食品工业科技, 2013, 34(19): 93-96. [8] CHEONG C G, EOM S H, CHANG C, et al. Crystallization, molecular replacement solution, and refinement of tetrameric beta-amylase from sweet potato[J]. Proteins, 1995, 21(2): 105-117. doi: 10.1002/prot.340210204
[9] YANG J, MOEINZADEH M H, KUHL H, et al. Haplotype-resolved sweet potato genome traces back its hexaploidization history[J]. Nature Plants, 2017, 3(9): 696-703. doi: 10.1038/s41477-017-0002-z
[10] 黄小芳, 毕楚韵, 石媛媛, 等. 甘薯基因组NBS-LRR类抗病家族基因挖掘与分析[J]. 作物学报, 2020, 46(8): 1195-1207. [11] ALTSCHUL S F, GISH W, MILLER W, et al. Basic local alignment search tool[J]. Journal of Molecular Biology, 1990, 215(3): 403-410. doi: 10.1016/S0022-2836(05)80360-2
[12] LU S, WANG J, CHITSAZ F, et al. CDD/SPARCLE: The conserved domain database in 2020[J]. Nucleic Acids Research, 2020, 48(D1): D265-D268. doi: 10.1093/nar/gkz991
[13] QUEVILLON E, SILVENTOINEN V, PILLAI S, et al. InterProScan: Protein domains identifier[J]. Nucleic Acids Research, 2005, 33: W116-W120. doi: 10.1093/nar/gki442
[14] SIEVERS F, HIGGINS D G. Clustal Omega for making accurate alignments of many protein sequences[J]. Protein Science, 2018, 27(1): 135-145. doi: 10.1002/pro.3290
[15] KRZYWINSKI M, SCHEIN J, BIROL I, et al. Circos: An information aesthetic for comparative genomics[J]. Genome Research, 2009, 19(9): 1639-1645. doi: 10.1101/gr.092759.109
[16] GU Z, CAVALCANTI A, CHEN F C, et al. Extent of gene duplication in the genomes of drosophila, nematode, and yeast[J]. Molecular Biology and Evolution, 2002, 19(3): 256-262. doi: 10.1093/oxfordjournals.molbev.a004079
[17] BAILEY T L, BODEN M, BUSKE F A, et al. MEME SUITE: Tools for motif discovery and searching[J]. Nucleic Acids Research, 2009, 37: W202-W208. doi: 10.1093/nar/gkp335
[18] KUMAR S, STECHER G, LI M, et al. MEGA X: Molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution, 2018, 35(6): 1547-1549. doi: 10.1093/molbev/msy096
[19] JI C Y, KIM H S, LEE C J, et al. Comparative transcriptome profiling of tuberous roots of two sweetpotato lines with contrasting low temperature tolerance during storage[J]. Gene, 2020: 727. doi: 10.1016/j.gene.2019.144244.
[20] KIM D, LANGMEAD B, SALZBERG S L. HISAT: A fast spliced aligner with low memory requirements[J]. Nature Methods, 2015, 12(4): 357-360. doi: 10.1038/nmeth.3317
[21] LOVE M I, HUBER W, ANDERS S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J]. Genome Biology, 2014, 15(12). doi: 10.1186/s13059-014-0550-8.
[22] TOTSUKA A, NONG V H, KADOKAWA H, et al. Residues essential for catalytic activity of soybean beta-amylase[J]. European Journal of Biochemistry, 1994, 221(2): 649-654.
[23] WU S, LAU K H, CAO Q, et al. Genome sequences of two diploid wild relatives of cultivated sweetpotato reveal targets for genetic improvement[J]. Nature Communications, 2018: 9. doi: 10.1038/s41467-018-06983-8.
[24] THALMANN M, COIRO M, MEIER T, et al. The evolution of functional complexity within the β-amylase gene family in land plants[J]. BMC Evolutionary Biology, 2019: 19. doi: 10.1186/s12862-019-1395-2.
[25] KANG Y N, ADACHI M, UTSUMI S, et al. The roles of Glu186 and Glu380 in the catalytic reaction of soybean beta-amylase[J]. Journal of Molecular Biology, 2004, 339(5): 1129-1140. doi: 10.1016/j.jmb.2004.04.029
[26] CHEN Y, CRIPPEN G M. An iterative refinement algorithm for consistency based multiple structural alignment methods[J]. Bioinformatics, 2006, 22(17): 2087-2093. doi: 10.1093/bioinformatics/btl351
[27] VALERIO C, COSTA A, MARRI L, et al. Thioredoxin-regulated beta-amylase (BAM1) triggers diurnal starch degradation in guard cells, and in mesophyll cells under osmotic stress[J]. Journal of Experimental Botany, 2011, 62(2): 545-555. doi: 10.1093/jxb/erq288
[28] 杨泽峰, 徐暑晖, 王一凡, 等. 禾本科植物β−淀粉酶基因家族分子进化及响应非生物胁迫的表达模式分析[J]. 科技导报, 2014, 32(31): 29-36. doi: 10.3981/j.issn.1000-7857.2014.31.002 [29] HOU J, ZHANG H, LIU J, et al. Amylases StAmy23, StBAM1 and StBAM9 regulate cold-induced sweetening of potato tubers in distinct ways[J]. Journal of Experimental Botany, 2017, 68(9): 2317-2331. doi: 10.1093/jxb/erx076
[30] HATTORI T, FUKUMOTO H, NAKAGAWA S, et al. Sucrose-induced expression of genes coding for the tuberous root storage protein, sporamin, of sweet potato in leaves and petioles[J]. Plant and Cell Physiology, 1991, 32(1): 79-86.
[31] 唐君, 周志林, 林冬兰, 等. 甘薯贮藏过程淀粉酶活性变化及对薯块芽萌发的影响[J]. 福建农业学报, 2010, 25(6): 699-702. doi: 10.3969/j.issn.1008-0384.2010.06.008 -
期刊类型引用(7)
1. 李志刚,彭兆伟,宁琳如,李亚美,金海勇,田鹏. 多传感器数据融合下气体密度继电器自动温控技术研究. 粘接. 2025(05): 139-142 .  百度学术
百度学术
2. 王俊鹏,黎良坤,冯杨. 卷烟条包端部贴标技术设计. 自动化仪表. 2025(04): 17-20+25 .  百度学术
百度学术
3. 刘洋,冀杰,赵立军,冯伟,贺庆,王小康. 基于激光雷达与相机融合的树干检测方法. 西南大学学报(自然科学版). 2024(02): 183-196 .  百度学术
百度学术
4. 江雪峰. 传感器信息融合下新能源汽车动力电池信号故障检测方法. 东莞理工学院学报. 2024(03): 94-99 .  百度学术
百度学术
5. 王张夫,汤显峰. 基于协方差交叉融合的多传感器数据融合研究. 电子测量技术. 2024(08): 78-85 .  百度学术
百度学术
6. 朱哲人,张伟. 基于传感器信息融合的运动员上肢运动姿态识别方法. 河北北方学院学报(自然科学版). 2024(07): 4-10 .  百度学术
百度学术
7. 焦萍萍,周显春,高华玲,杨真. 复杂环境下多模态交通警示信息推荐算法仿真. 计算机仿真. 2023(07): 121-125 .  百度学术
百度学术
其他类型引用(4)





 下载:
下载: