Identification of an antagonistic strain Bacillus amyloliquefaciens E3 against Dickeya zeae and its antimicrobial activity
-
摘要:目的
纯化和鉴定从柑橘根际土壤分离得到的可抑制水稻基腐病菌Dickeya zeae EC1生长的解淀粉芽孢杆菌Bacillus amyloliquefaciens,并解析其抑菌机制。
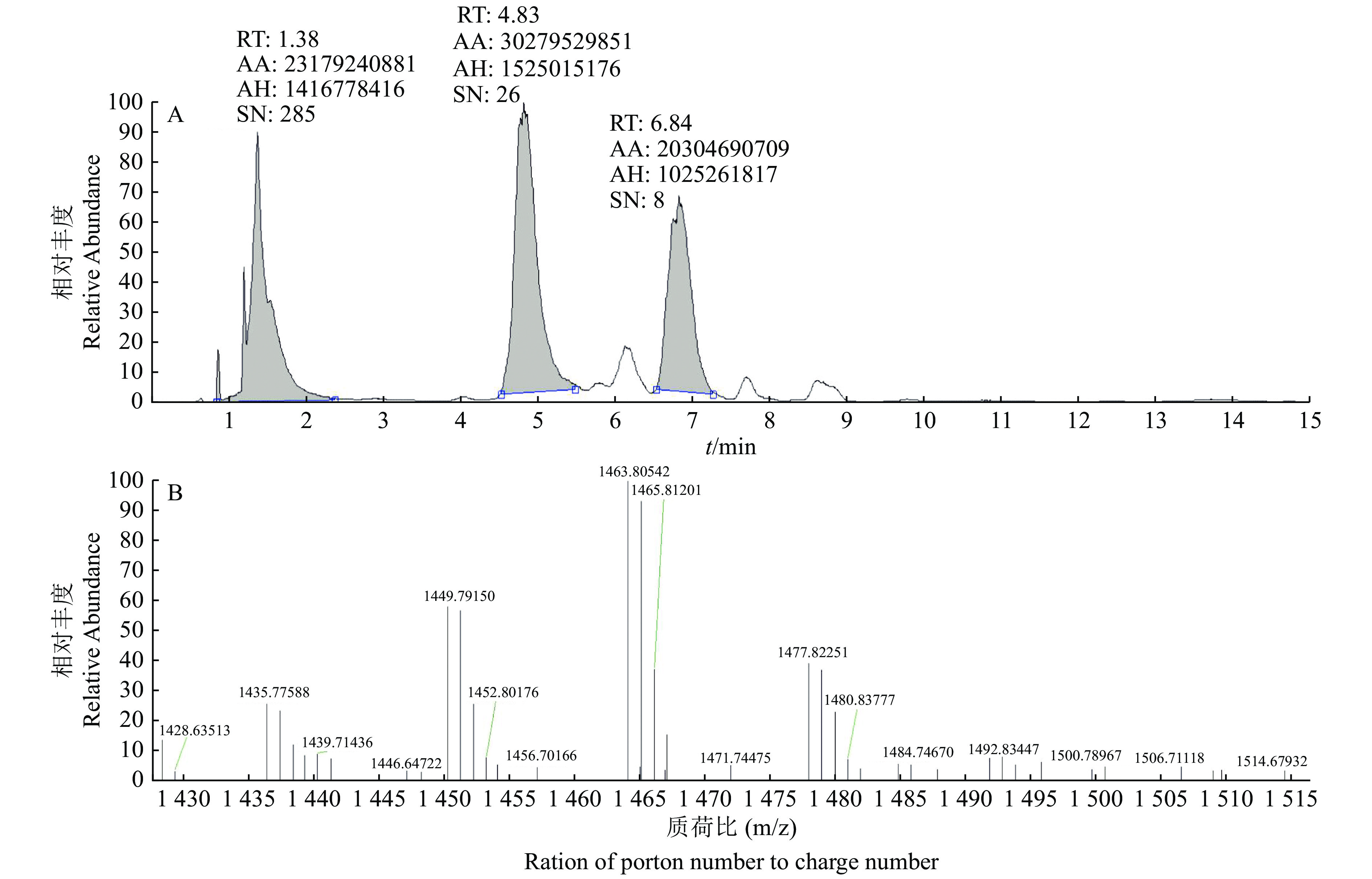
方法采用平板扩散法从根际土壤中筛选出1株拮抗D. zeae EC1的B. amyloliquefaciens E3菌株;根据细菌菌落表型、生理生化特征结合16S rDNA序列分析等方法对E3菌株进行分类鉴定;检测无菌E3培养液对D. zeae EC1侵染水稻种子能力的影响;盐酸沉淀结合丙酮抽提法提取E3菌株脂肽粗提物;采用平板对峙法测定E3菌株脂肽粗提物对病原真菌的抑菌活性,琼脂扩散法测定E3菌株脂肽粗提物对病原细菌的抑菌活性及最小抑菌浓度(Minimum inhibitory concentration, MIC);用Durashell C18柱对脂肽粗提物进行HPLC分离纯化;通过液相色谱−质谱联用(LC-ESI-MS)分析,鉴定抑菌物质的相对分子质量并推测其化学组成。
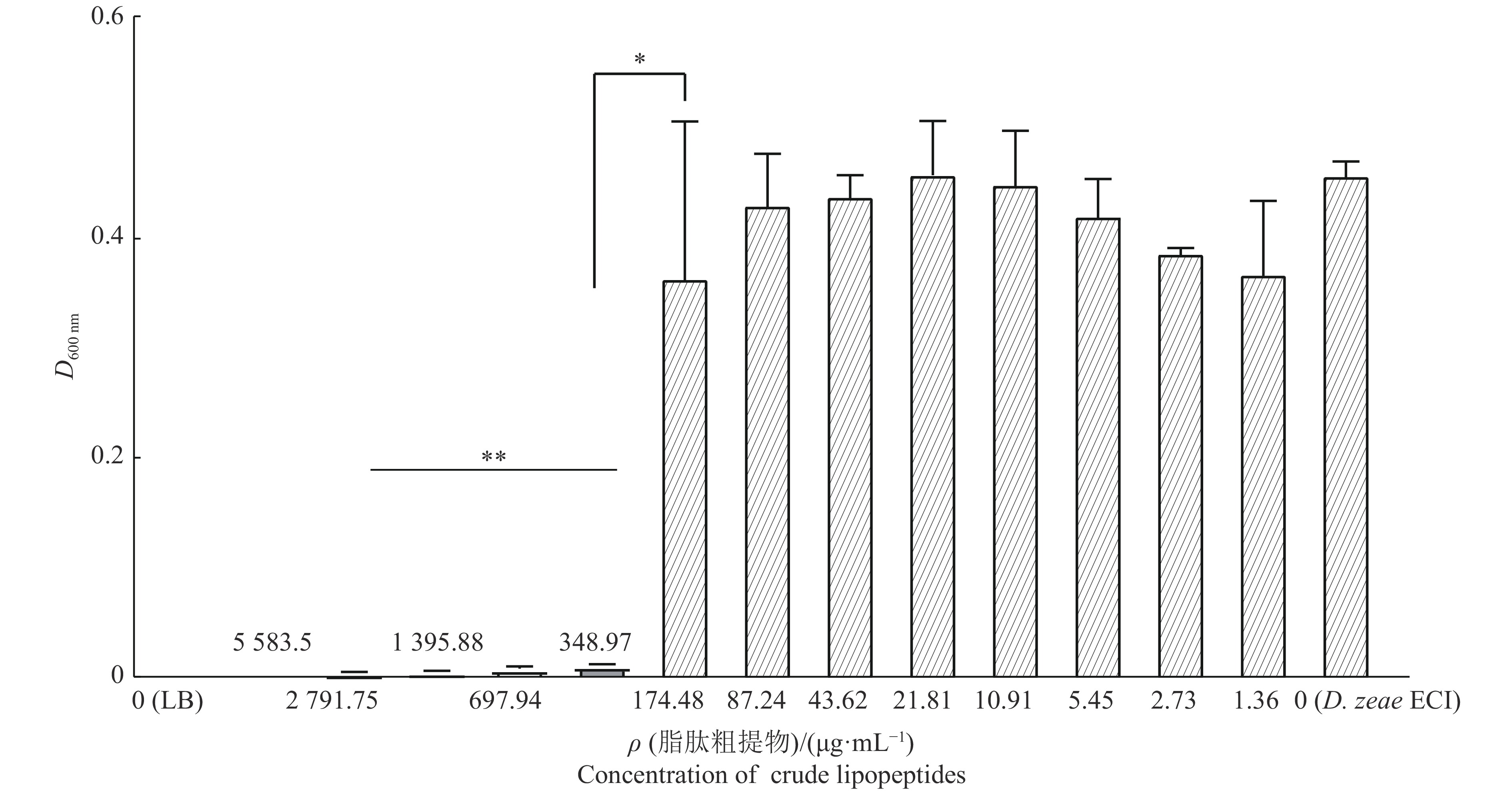
结果B. amyloliquefaciens E3菌株对茄病镰刀菌Fusarium solani、香蕉基腐病菌、茄科劳尔氏菌Ralstonia solanacearum等多种植物病原菌生长具有抑制作用,表现为广谱抗性;B. amyloliquefaciens E3菌株的无菌培养液能够抑制D. zeae EC1侵染萌发的水稻种子,显著提高水稻种子的萌芽率;B. amyloliquefaciens E3菌株的脂肽粗提物可以抑制D. zeae EC1的生长,其MIC为348.97 µg/mL;HPLC分离纯化结合LC-ESI-MS分析发现,B. amyloliquefaciens E3菌株分泌的主要抑菌活性物质包括Surfactin、Fengycin和Iturin 3类脂肽类抗生素。
结论B. amyloliquefaciens E3菌株具有作为生防菌的潜力,它可能通过分泌Surfactin、Fengycin和Iturin 3种脂肽类抑菌活性物质,拮抗水稻基腐病菌、茄科劳尔氏菌、茄病镰刀菌等多种植物病原菌。研究结果可为该菌的生物防治应用提供理论依据。
Abstract:ObjectiveTo isolate and identify Bacillus amyloliquefaciens strain which can inhibit the growth of Dickeya zeae EC1 from the rhizosphere soil of citrus plants, and analyze its antibacterial mechanism.
MethodThe B. amyloliquefaciens E3 strain antagonizing the growth of D. zeae EC1 was screened from the rhizosphere soil by plate diffusion method. The E3 strain was classified and identified based on colony phenotype, physiological and biochemical characteristics of bacteria and 16S rDNA sequence analysis. Effects of E3 sterile culture medium on the seeds germination ability of rice infected by D. zeae EC1 was detected. The crude lipopeptides of E3 strain was extracted by hydrochloric acid precipitation combined with acetone extraction. The antimicrobial activity of crude lipopeptides against pathogenic fungi were determined by plate confrontation method. The antimicrobial activity of crude lipopetides against pathogenic bacteria were determined by agar diffusion method, and the minimum inhibitory concentration (MIC) was obtained. The crude lipopeptides were separated and purified by HPLC with Durashell C18 column. Liquid chromatography-mass spectrometry (LC-ESI-MS) analysis was used to identify the molecular weights of the antimicrobial substances and speculate its chemical composition.
ResultB. amyloliquefaciens E3 strain had an inhibitory effect against Fusarium solani, D. zeae MS1 and Ralstonia solanacearum, etc, indicating that it had broad-spectrum resistance. The sterile culture medium of B. amyloliquefaciens E3 strain inhibited the infection of germinating rice seeds by D. zeae EC1 and significantly increased the seed germination rate. The crude lipopeptides of E3 strain inhibited the growth of D. zeae EC1 with the MIC of 348.97 µg/mL. HPLC combined with LC-ESI-MS analysis showed that the main antibacterial substances secreted by E3 strain included surfactin, fengycin and iturin.
ConclusionThe B. amyloliquefaciens E3 strain is potential to be a biocontrol agent. It can antigonize a variety of plant pathogens such as D. zeae EC1, R. solanacearum and F. solani by secreting three kinds of lipopeptides including surfactin, fengycin and iturin. The results provide a theoretical basis for the application of B. amyloliquefaciens E3 in biological control.
-
苹果作为一种常见的水果,其质量问题直接关系到消费者的健康和生产者的经济利益。我国是苹果种植大国,苹果采摘、运送、储藏与加工处理是不可忽视的关键环节[1]。然而,由于人工质检的主观性和效率低下,传统的苹果质检方式已经无法满足现代农产品质量的需求[2]。基于机器学习的苹果识别技术应运而生,该方法可以对苹果进行自动识别和分类。李大华等[3]针对自然复杂环境下的苹果重叠问题,利用谱聚类算法进行分割,然后使用随机霍夫变换实现果实的识别和定位,尽管改进后的谱聚类算法在计算量上有所优化,但仍然涉及多步骤的处理,包括均值漂移预分割、稀疏矩阵构建、K-means分类和随机霍夫变换,整体流程较为复杂,对硬件性能要求较高,不适合实时性要求较高的应用场景。王迎超等[4]为了准确快速实现多特征融合的苹果分级,提出了一种基于K-means聚类和改进多层感知器(Multilayer perception, MLP)的苹果分级方法,但特征权重的确定存在主观性,且在不同的应用场景或苹果品种中特征的重要性会有所不同,导致分级准确性下降。宋怡焕等[5]提出基于最小二乘支持向量机(LS-SVM)检测苹果果梗/花萼缺陷,试验中仅使用了 180 幅苹果图像,而训练数据的不足会导致模型的泛化能力受限,对不同环境或苹果品种的误判率较高。上述传统的机器学习算法试验设计过于繁琐,对复杂的特征提取和分类能力不佳,易受外界因素的干扰,难以获得较好的检测结果。随着深度学习技术突飞猛进,以深度学习为基础的目标检测技术在苹果果实检测[6]、病虫害监测[7]、果实成熟度[8]和农业自动化[9-10]等领域发挥着重要的作用。目前,基于深度学习的苹果检测算法依据检测的阶段划分主要有2大类:一类是要先用特征提取器生成一系列可能包含待检物体的预选框,然后利用算法对生成的候选区域进行更精细的检测和分类的两阶段算法,主要有Faster-RCNN[11]和Mask-RCNN[12]算法;另一类为直接预测物体类别和位置的一阶段检测算法,主要有RetinaNet[13]、SSD[14]和YOLO系列[15-17]。两阶段算法计算复杂度高、检测速度较慢,一阶段算法在精度上有所欠缺。两阶段算法由于包含多个阶段和较多的候选框处理,推理速度通常比单阶段算法慢,不适合实时应用。为了进一步提升模型的检测速度和准确度,Tian等[18]提出了一种名为VMF-SSD(基于V空间的多尺度特征融合SSD)的新型苹果叶片病害检测方法;Wang等[19]通过迁移学习构建YOLOv5s检测模型的同时,采用通道修剪算法对模型进行修剪及微调,以实现对苹果果实的快速准确检测。
目前,国内外目标检测领域对苹果的研究主要集中在自然环境下的苹果识别、苹果采摘机器人以及叶片病虫害识别方面。虽然已有部分研究针对缺陷苹果的检测[20-21],但仍存在上下文信息和多尺度特征融合不充分的问题。针对缺陷苹果识别,本文提出一种基于BiFPN[22]和Triplet注意力机制的YOLOv5s (BTF-YOLOv5s)缺陷苹果识别算法,通过BiFPN结合特征的上采样和下采样路径,联合跨尺度的特征交互,能够更好地传递和融合来自不同尺度的特征信息,提升目标检测性能;同时,学习动态的特征融合权重,根据不同目标的重要性进行灵活调整,增强模型的鲁棒性;在Neck层应用Triplet注意力机制,模型能更好地捕捉图像中的细节和上下文信息,提升目标检测的精度;采用Focal-CIoU调整正负样本之间的损失权重,让模型对难以分类的样本给予更多关注,提升模型的分类性能。
1. 基于BiFPN和Triplet注意力机制的YOLOv5s算法
本研究以YOLOv5s (版本6.2,包含4种不同大小的模型)为基础,网络结构由输入端、骨干网络、颈部网络和输出端4个部分组成。在输入端引入了Mosaic数据增强方法,随机选取4张图像,对它们进行随机放大、缩小等操作后,拼接成一张新的图像,使模型可以在更小的范围内识别目标;骨干网络主要使用了Focus和CSP1_X结构;颈部网络采用FPN+PAN和CSP2_X结构进一步进行特征融合和上采样操作,以提供更高级的语义信息和适应不同尺度图片的能力;输出端采用CIoU_Loss(Complete intersection over union loss)作为损失函数,并使用了非极大值抑制(Non-maximum suppression,NMS)进行后处理。本文提出了一种改进的YOLOv5s算法(BTF-YOLOv5s)用于缺陷苹果识别,首先,加权双向特征金字塔网络(Bidirectional feature pyramid network,BiFPN)替代YOLOv5中的FPN+PAN结构,通过引入可学习的权重学习不同输入特征的重要性;然后,将Triplet注意力机制应用到模型的最后一层,确保在最终输出前对特征进行充分的处理和优化;最后,采用Focal-CIoU损失函数调整损失权重给予缺陷苹果更多的关注,BTF-YOLOv5s算法结构整体框图如图1所示。
1.1 BiFPN
在目标检测任务中,有效地获取并处理不同尺度的特征信息是一个主要的挑战。传统的特征金字塔网络(FPN)[23]通过自上而下的方式聚合多尺度特征,如图2a所示,但容易受到单向信息流的限制;路径聚合网络(PANet)[24]在此基础上额外添加了一个自下向上的路径聚合网络,如图2b所示。在YOLOv5的Neck结构中,借鉴PANet的思想,高层的特征信息通过FPN+PAN结构进行传递融合。FPN+PAN结构虽然提高了特征传递的效率,但也增加了计算复杂度,尤其是在处理高分辨率输入时,可能导致较高的计算成本和较差的实时性;此外,固定结构导致对不同任务和数据缺乏自适应的能力。
为了解决上述信息流动单一、精度高但参数较多、计算量较大以及简单拼接导致的信息丢失和冗余等问题,本文提出了采用BiFPN替代YOLOv5s中的FPN+PAN结构。BiFPN在PANet和NAS-FPN(图2c)基础上优化了多尺度特征融合方式,结构如图2d所示。
传统方法在融合具有不同分辨率的特征时,平等地对待所有输入特征,并将它们简单相加。但不同分辨率的输入特征对输出特征的贡献通常是不等的。为了解决这一问题,BiFPN为每个输入特征增加了一个额外的权重,并让网络学习每个输入特征的重要性,具体见式(1)和式(2):
$$ P_6^{{\mathrm{td}}} = {\mathrm{Conv}}\left[ {\frac{{{w_1}P_6^{{\mathrm{in}}} + {w_2}{\mathrm{Reseize}}\left( {P_7^{{\mathrm{in}}}} \right)}}{{{w_1} + {w_1} + \varepsilon }}} \right], $$ (1) $$ P_6^{{\mathrm{out}}} = {\mathrm{Conv}}\left[ {\frac{{w_1^{'}P_6^{{\mathrm{in}}} + w_2^{'}P_6^{{\mathrm{td}}} + w_3^{'}{\mathrm{Reseize}}\left( {P_5^{{\mathrm{out}}}} \right)}}{{w_1^{'} + w_2^{'} + w_3^{'} + \varepsilon }}} \right], $$ (2) 式中,
$ P_6^{{\mathrm{td}}}$ 为自顶向下路径第6层的中间特征;$ P_6^{{\mathrm{in}}}$ 为第6层的输入特征; Resize用于分辨率匹配的上采样或下采样运算;$ P_6^{{\mathrm{out}}}$ 为自底向上路径第6层的输出特征。这一改进使得网络能够自适应地调整每个特征图的重要性,从而实现更有效的特征融合。引入BiFPN的YOLOv5s模型通过多尺度特征融合和简化的计算过程,有效地提升了对缺陷苹果检测的准确率;BiFPN通过引入可学习的融合权重优化了不同分辨率特征的重要性,增强了特征复用和多级特征融合路径,使得YOLOv5能够有效地检测出不同大小和形状的苹果缺陷。
1.2 Triplet注意力机制
在缺陷苹果检测任务中,模型需要准确地识别和定位图像中的目标(苹果)。图像中存在大量的背景信息以及不同大小的苹果会分散模型的注意力,使得模型难以准确地区分缺陷苹果和背景,引入注意力机制旨在提升模型对关键目标的感知能力。SENet通过简单的操作(全局平均池化和全连接层)自适应地调整特征图中每个通道的重要性,使网络更加集中地关注重要的特征信息,在低计算成本的条件下通过通道注意力机制显著提升网络的表示能力[25];CBAM将通道注意力和空间注意力相结合,能够同时关注重要的通道特征和空间位置;CBAM中的通道注意力方法虽然提供了一定的性能改进,但通道注意力和空间注意力是相互分离和计算的[26]。
Triplet 注意力机制(简称“Triplet”)以一种有效的方式解释了CBAM未考虑到的跨维度的相互作用[27]。如图3所示,Triplet由3个平行的分支构成,其中2个分支分别用来捕获通道
$C$ 维度和空间维度$ W/H $ 之间的跨通道交互,最后的一个分支用于捕获$ H $ 和$W$ 的空间依赖关系。在第1个分支中,输入张量C×H×W(通道数$C$ 、高度$H$ 和宽度$W$ )沿$H$ 轴逆时针旋转90°,形状变为$W$ ×$H$ ×$C$ ,输入特征先后经过$ Z - {\mathrm{Pool}} $ 、$ K \times K $ 的标准卷积层、批量归一化层、通过Sigmod激活函数生成空间注意力权重后、再沿H轴顺时针旋转90°保持与输入的形状一致。其中$ Z - {\mathrm{Pool}} $ 表示为:$$ Z - {\mathrm{Pool}}(\chi ) = \left[{\mathrm{Max}}{{\mathrm{Pool}}_{0{\mathrm{d}}}}(\chi ),{\mathrm{Avg}}{{\mathrm{Pool}}_{0{\mathrm{d}}}}(\chi )\right]{\text{,}} $$ (3) 式中,
$ 0{\mathrm{d}}$ 表示发生最大池化和平均池化操作的第0维度。第2、第3个分支同理,最后对3个分支输出特征进行平均后聚合在一起,最终输出的张量:
$$\begin{split} y =& \frac{1}{3}\Biggr\{ \overline {\mathop {{\chi _1}}\limits^ \wedge \sigma \left[ {{\psi _1}\left( {\mathop {\chi _1^*}\limits^ \wedge } \right)} \right]} + \overline {{{\mathop \chi \limits^ \wedge }_2}\sigma \left[ {{\psi _2}\left( {\mathop {\chi _2^*}\limits^ \wedge } \right)} \right]} + \Biggr.\\&\Biggr.\chi \sigma \left[ {{\psi _3}\left( {\mathop {{\chi _3}}\limits^ \wedge } \right)} \right] \Biggr\}{\text{,}} \end{split} $$ (4) 式中,
$ \sigma $ 代表Sigmod激活函数;$ \psi _1$ 、$ \psi _2$ 和$ \psi _3$ 表示由核大小$K$ 定义的标准二维卷积层。Triplet通过旋转操作构建各维度间的相关性,再对其进行残差变换,并以极小的计算成本对信息进行编码,在不需要太多可学习参数的前提下建立通道之间的相互依赖关系,有效地捕捉苹果图像中的关键特征。
1.3 Focal-CIoU损失函数
在机器学习任务中,常常面临数据集中不同类别的样本数量差异较大的问题,传统的交叉熵损失函数在处理样本不均衡问题时表现不佳,因为它将所有样本的重要性视为相等,继而导致模型在训练过程中更容易偏向于数量多的类别,公式如下:
$$ {\mathrm{CE}}(p,y) = {\mathrm{CE}}({P_t}) = - \ln ({P_t}) {\text{,}}$$ (5) 式中,CE是交叉熵(Cross entropy),
$p$ 代表模型预测样本属于类别1的概率(取值范围0~1),$y$ 表示样本的标签(取值为−1和1)。定义$$P_t=\left\{ \begin{array}{ll} {p}, &{{\text{当}} \; y=1{\text{时}}}\\ {1-p},& {{\text{其他}}} \end{array}\right. {\text{。}}$$ (6) 数据集中正常苹果和缺陷苹果图片数量均为1 600张左右,但标注数量略有差异,分别为4 624和2 039张。为了进一步加强模型对缺陷苹果的检测能力,引入Focal-CIoU损失函数。Focal Loss(FL)在交叉熵损失的基础上进行了改进,引入了平衡因子和聚焦因子,平衡因子用于调整正负样本之间的权重,聚焦因子用于调整难易样本间的损失权重;这样,既能调整正负样本的权重,又能控制难易分类样本的权重。FL表达式[28]为:
$$ {\mathrm{FL}}({P_t}) = - {\alpha _t}{(1 - {P_t})^\gamma }\ln ({P_t}), $$ (7) 式中,
${\alpha _t}$ 是超参数(取0.25);$ {(1 - {P_t})^\gamma } $ 是调节因子,$\gamma $ 是$ \geqslant $ 0的可调节聚焦参数(取2)。CIoU损失函数(LossCIoU)考虑边界框宽高比的尺度信息,在DIoU的基础上进行优化,计算公式[29]如下:
$$ {\mathrm{DI}}{\text{o}}{\mathrm{U}} = {\mathrm{IoU}} - \frac{{{d^2}}}{{{c^2}}}{\text{,}} $$ (8) $$ {\mathrm{Loss}}_{\mathrm{CIoU}} = {\mathrm{IoU}} - {\frac{{{\rho ^2}\left( {b,{b^{gt}}} \right)}}{{{c^2}}}^{}} - \alpha V{\text{,}} $$ (9) 式中,IoU为交并比(Intersection over Union),即“预测的边框”和“真实的边框”的交集和并集的比值;
$ d$ 为预测框与真实框中心点的长度,$ c$ 为最小外接矩形的对角线距离,$\rho $ 代表的是2个中心点之间的欧式距离,$ b、{b^{gt}} $ 分别代表了预测框和真实框的中心点,$\alpha $ 是权重函数,$V$ 用来衡量长宽比的相似度。Focal-CIoU损失函数(
${\mathrm{L}}{\text{os}}{{\text{s}}_{{\mathrm{Focal}} - {\mathrm{CIoU}}}} $ )为:$$ {\mathrm{L}}{\text{os}}{{\text{s}}_{{\mathrm{Focal}} - {\mathrm{CIoU}}}} = {\mathrm{Io}}{{\mathrm{U}}^\gamma } \times {\mathrm{Los}}{{\mathrm{s}}_{{\mathrm{CIoU}}}} {\text{。}}$$ (10) Focal-CIoU通过调整正负样本的权重和提升边界框回归精度,减少了训练过程中梯度的不稳定性,使模型更关注缺陷苹果,提高了检测性能。
2. 试验与结果分析
2.1 数据集
本研究所用数据集从百度网站和飞桨AI Studio网站上获取,这2个网站提供了丰富的图像资源,包含虫蛀、腐烂、机械损伤和褶皱共4类苹果缺陷图像(图4),数量分别为414、750、148和280张,以确保对于苹果目标的全面覆盖。数据集共
3213 张(正常苹果1621 张、缺陷苹果1592 张),训练集和验证集按照9∶1的比例划分,其中,训练集图片2 891张、验证集图片322张。为了增加数据的多样性,通过Python程序对部分数据集进行水平、垂直方向的翻转以及旋转45º操作。构建和处理数据集,确保模型在训练和验证过程中能够充分学习和适应苹果目标的多样性特征,为后续的目标检测提供数据保障。2.2 模型训练与评价指标
本试验平台基于Ubuntu 18.04.6的64位操作系统,显卡为GPU(NVIDIA GeForce RTX2080Ti),显存为12 G,使用PyTorch框架构建模型,编程语言为Python,Torch版本为1.10.1。在训练过程中,设置初始学习率为0.01、周期学习率为0.20、动量为0.937、权重衰减系数为0.000 5、批量大小为16。模型评估指标包括准确率(Precision,P)、召回率(Recall,R)、平均精确率均值(Mean average precision,mAP)和F1。TP (True positive)表示模型正确地检测出缺陷苹果的数量;TN (True negative)表示模型正确地排除正常苹果的数量;FP (False positive)表示模型错误地将正常苹果判定为缺陷苹果的数量; R指模型正确检测出的缺陷苹果(TP)占所有真正的缺陷苹果(TP + FN)的比例,表示模型对于真实缺陷苹果的检测能力;AP(Average precision)是衡量目标检测模型在不同召回率下准确性的指标,mAP是所有类别AP的平均,用来评估模型整体性能; F1综合了P和R这2个指标,能够更全面地评估模型的性能。
训练过程中损失值参数的动态变化如图5所示,随着训练轮次递增,损失值经历了急剧下降的阶段,最终趋向于0.025左右的平稳水平。训练损失和验证损失逐渐趋于拟合,意味着模型所学习的训练数据中的模式能够有效泛化到验证数据,表明该模型在面对新数据时具有优异的泛化性能。
2.3 Triplet的不同添加位置
Triplet插入YOLOv5s结构中的位置(A表示YOLOv5s-BiFPN-Triplet)主要有以下4种方式:1)在网络结构SPPF前面添加,即第9层[A+CIoU+Focal (9)];2)在网络结构最后一层添加,即第24层[A+ CIoU+Focal(24)];3)在网络结构SPPF前面和最后一层添加,即A+CIoU+Focal(9+25);4)替换Backbone中的C3模块,即A+CIoU+Focal。不同插入位置对比试验结果见表1。由表1可见,A+CIoU+Focal(24)比其他4种模型在各个方面均有不错的提升,准确率比YOLOv5s的提高了5.7个百分点,综合分析表明A+ CIoU+Focal(24)是最优的模型。
表 1 Triplet注意力机制不同插入位置的模型试验结果对比Table 1. Result comparison of Triplet attention mechanism with different insertion position% 模型1)Model P R mAP F1 YOLOv5s 75.4 83.6 86.5 79.29 A+ CIoU+Focal(9) 77.3 81 85.6 79.11 A+CIoU+Focal(9+25) 80.8 79.9 86.2 80.35 A+CIoU+Focal 75.9 85.2 87.1 80.28 A+CIoU+Focal(24) 81.1 85.8 90.0 83.38 1) A:YOLOv5s-BiFPN-Triplet,括号内数字表示Triplet插入位置。
1) The number in parentheses indicated the insertion position of Triplet.2.4 BiFPN添加不同注意力机制
以YOLOv5s-BiFPN为基础,逐步添加SE,CBAM、CA和Triplet注意力机制。SE仅考虑通道间的信息,忽略了位置信息;CBAM通过学习的方式自动获取每个特征通道和特征空间的重要程度;而CA不仅考虑通道间的关系,而且考虑方向相关的位置信息。试验结果见表2,由表2可见,BiFPN+CA在准确率和mAP方面优于BiFPN+SE和BiFPN+CBAM;Triplet注意力机制利用三分支结构能够捕获更多的关键信息,建立通道之间的相互依赖关系,试验结果也验证了BiFPN+Triplet是最优模型。
表 2 BiFPN添加不同注意力机制的模型试验结果对比Table 2. Result comparison of BiFPN with different attention mechanism% 模型Model P R mAP BiFPN+SE 76.1 83.1 87.0 BiFPN+CBAM 77.5 81.5 87.6 BiFPN+CA 78.6 81.3 88.1 BiFPN+Triplet 79.3 83.2 88.4 2.5 消融试验
为了验证各个模块的作用,以YOLOv5s为基础,进行逐步添加和替换,结果见表3。从表3中可以看出,与初始的YOLOv5s模型相比,单独添加BiFPN、Triplet或Focal-CIoU模块的模型准确率、召回率和mAP均有一定的提升。同时添加2种模块时,取得的效果更佳,添加BiFPN+Triplet的模型mPA比单独添加BiFPN的提高了0.4个百分点、准确率比单独添加Triplet的提高2.9个百分点;添加Triplet+Focal-CIoU的模型准确率和mAP比单独添加Triplet的分别提高1.1和0.3个百分点;添加BiFPN+Focal-CIoU的模型准确率和mAP比单独添加Focal-CIoU的分别提高1.7和1.0个百分点。当三者同时添加时,提高了模型对缺陷苹果的感知能力,与YOLOv5s模型相比,准确率、召回率和mAP分别提高5.7、2.2和3.5个百分点。
表 3 消融试验结果Table 3. The result of ablation test% BiFPN Triplet Focal-CIoU P R mAP 75.4 83.6 86.5 √ 80.3 84.3 88.0 √ 76.4 86.1 89.3 √ 78.4 83.0 89.8 √ √ 79.3 83.2 88.4 √ √ 77.5 84.5 89.6 √ √ 80.1 84.0 88.9 √ √ √ 81.1 85.8 90.0 为了验证Focal-CIoU损失函数的优越性,本文设计了YOLOv5s-BiFPN-Triplet+损失函数的消融试验,结果见表4。由表4可知,A+ CIoU+Focal与次优模型A+ SIoU相比,准确率提高了1个百分点、召回率和mAP提高0.2个百分点、F1提高0.62个百分点;虽然A+ WIoU的准确率与A+ CIoU+Focal相同,但召回率、mAP和F1均低于A+ CIoU+Focal。综合准确率、召回率、mAP和F1等指标, A+ CIoU+Focal是最优模型。
表 4 Focal-CIoU与其他损失函数对比Table 4. Comparison of Focal-CIoU with other loss functions% 模型1)Model P R mAP F1 A 79.3 83.2 88.4 81.20 A+ DIoU 79.1 84.8 89.2 81.85 A+ DIoU+Focal 79.4 84.3 89.6 81.78 A+ SIoU 80.1 85.6 89.8 82.76 A+ SIoU+Focal 77.9 85.3 88.5 81.43 A+ EIoU 78.7 84.3 88.7 81.40 A+ EIoU+Focal 80.7 80.0 88.1 80.35 A+ WIoU 81.1 79.4 88.2 80.24 A+CIoU+Focal 81.1 85.8 90.0 83.38 1) A:YOLOv5s-BiFPN-Triplet. 2.6 模型有效性验证
为了证明本文提出模型(BTF-YOLOv5s)的有效性,与当前主流的目标检测模型进行对比(表5)。由表5可见,与SSD相比,BTF-YOLOv5s的准确率提高了4.8个百分点、召回率和mAP分别提高了4.1和5.7个百分点,模型大小大幅度减小;与YOLOv3相比,BTF-YOLOv5s的准确率提高近10个百分点,模型小了近8倍;与YOLOv4相比,BTF-YOLOv5s虽然准确率低了3.1个百分点,但召回率和mAP分别提升近20和13个百分点,模型大小减少了17倍;与YOLOv5s、YOLOv7和YOLOv8s相比,BTF-YOLOv5s在准确率、召回率和mAP方面均有提升;YOLOv8n和YOLOv9的准确率相较于BTF-YOLOv5s各提高0.6和4.9个百分点,但二者召回率过低; F1显示BTF-YOLOv5s是最优的。综合分析,BTF-YOLOv5s显著优于大多数目标检测模型,更具有优势。图6直观地展示了SSD、YOLOv3、YOLOv5s等模型与BTF-YOLOv5s的mAP比较,在训练轮次逐步增加的过程中,BTF-YOLOv5s的mAP最终稳定在90%左右,明显优于其他模型。
表 5 BTF-YOLOv5与其他模型的对比Table 5. Comparison of BTF-YOLOv5 with other models模型
ModelP/% R/% mAP/% 模型大小/MB
Model sizeF1/% 参数
ParameterGFLOPs1) SSD 76.3 81.7 84.3 95.5 78.91 2.49×107 31.4 YOLOv3 71.8 83.8 86.5 123.5 77.34 3.30×107 78.1 YOLOv4 84.2 65.6 76.6 256.3 73.75 6.50×107 142.3 YOLOv5s 75.4 83.6 86.5 14.4 79.29 7.03×106 16.0 YOLOv7 79.6 83.1 87.1 142.1 81.31 3.72×107 105.1 YOLOv8n 81.7 76.6 87.4 6.2 79.07 3.01×106 8.2 YOLOv8s 73.6 83.0 87.2 22.5 78.02 1.11×107 28.6 YOLOv9 86.0 78.8 89.7 102.8 82.24 5.10×107 238.9 BTF-YOLOv5s 81.1 85.8 90.0 14.7 83.38 7.17×106 16.7 1) GFLOPs:每秒10亿次的浮点运算数。
1) GFLOPs: Giga floating-point operations per second.2.7 应用场景试验结果
本模型的应用场景之一即苹果采摘机器人在采摘过程中的分拣,故选择缺陷苹果所处的自然环境下的场景以及摆放不规整的场景。如图7所示,其中,第1行图像为原始图像、第2行图像为YOLOv5s检测图、第3行图像为BTF-YOLOv5s检测图;在图7的第1列中,BTF-YOLOv5s能够有效检测出正常苹果与缺陷苹果,准确率比YOLOv5s有显著提升;在第2列中对于像素更少的瑕疵斑点,YOLOv5s模型不仅出现了误检,而且准确率也低于BTF-YOLOv5s;在第3列中,BTF-YOLOv5s模型的检测效果明显优于YOLOv5s模型。
3. 结论
为了实现对瑕疵苹果的快速准确检测,本文提出基于BiFPN和Triplet注意力机制的YOLOv5s缺陷苹果识别算法。通过加权双向特征金字塔网络(BiFPN)来学习不同的输入特征;采用Triplet注意力机增强模型对目标之间的关联和上下文信息的表示能力;采用Focal-CIoU损失函数,在计算损失时调整损失权重,给予目标物体更多的关注。注意力机制的应用使模型更加关注目标,在4种位置的插入对比中,YOLOv5s网络结构最后一层的插入位置表现最为优越,与在网络结构SPPF前面和最后一层添加相比,准确率、召回率和mAP分别提高了0.3、5.9和3.8个百分点;BiFPN与SE、CBAM、CA、Triplet注意力机制两两组合,验证了BiFPN+Triplet的优越性;相较于BiFPN+CA,准确率、召回率和mAP分别提升了0.7、1.9和0.3个百分点。消融试验表明,以YOLOv5s为基础,同时添加3种模块的准确率、召回率和mAP分别提高了5.7、2.2和3.5个百分点,提升效果显著。以YOLOv5s-BiFPN-Triplet为基础,Focal-CIoU的准确率比次优损失函数SIoU高1个百分点;准确率、召回率和mAP比主流的YOLOv7算法提高了1.5、2.7和2.9个百分点,同时,模型大小从142.1 MB减小到14.7 MB,显著降低了内存占用,在计算资源受限的环境中,为部署目标检测系统提供了有力支持。YOLOv8n和YOLOv9的准确率虽略高于BTF-YOLOv5s,但均面临召回率过低的问题,而YOLOv8s的准确率只有73.6%。在应用场景的对比试验中,BTF-YOLOv5s也表现出较好的优越性。
-
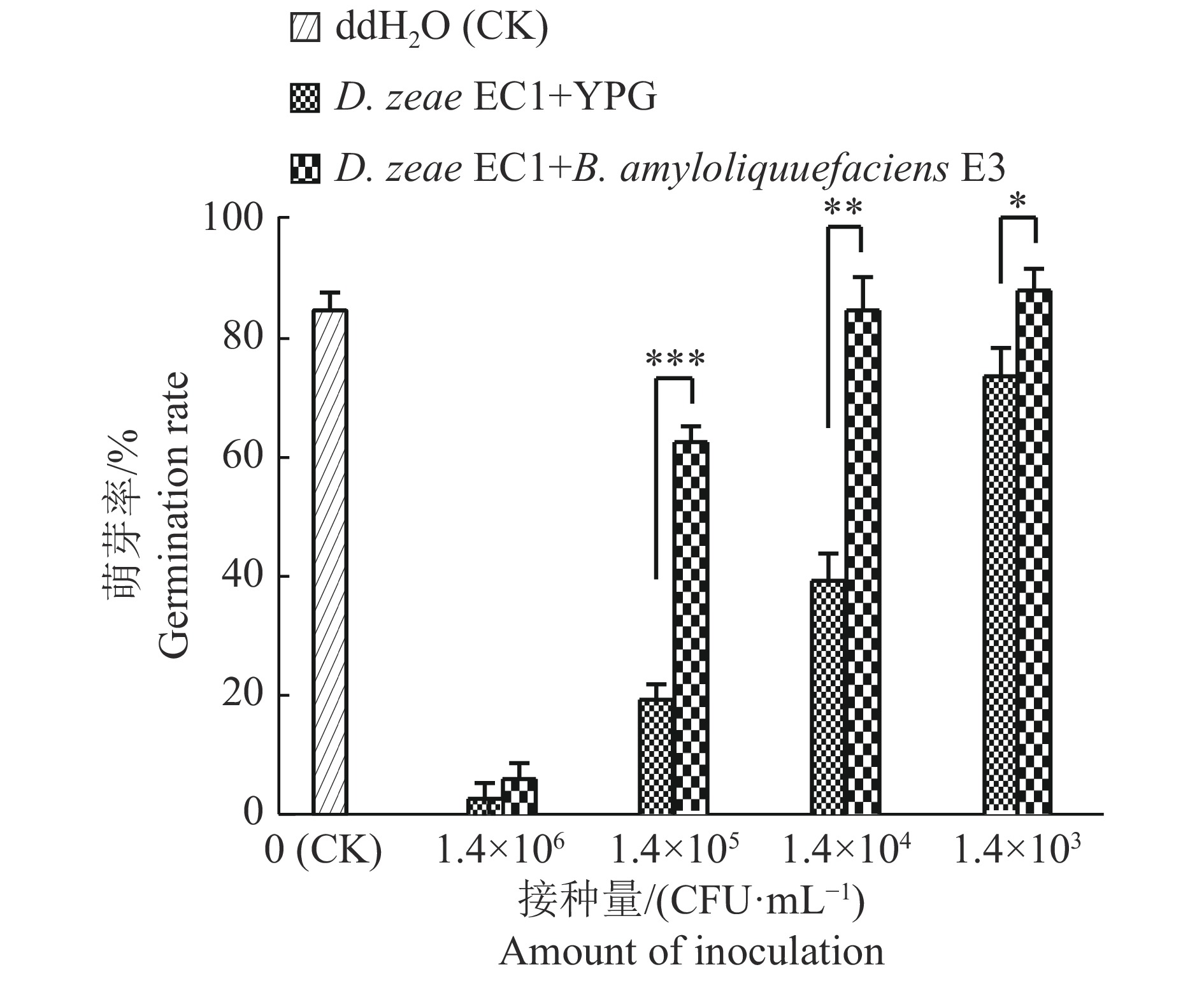
图 5 Bacillus amyloliquefacions E3菌株无菌培养液对Dickeya zeae EC1抑制水稻种子萌芽率的拮抗效果
“*”、“**”、“***”分别表示0.05、0.01和0.000 1水平的差异显著性(t检验)
Figure 5. Antagonism effect of the sterile culture medium of Bacillus amyloliquefaciens E3 strain on the inhibition of rice seeds germination by Dickeya zeae EC1
“*”,“**”,“***” indicate significance differences at the levels of 0.05,0.01 and 0.000 1 respectively (t test)
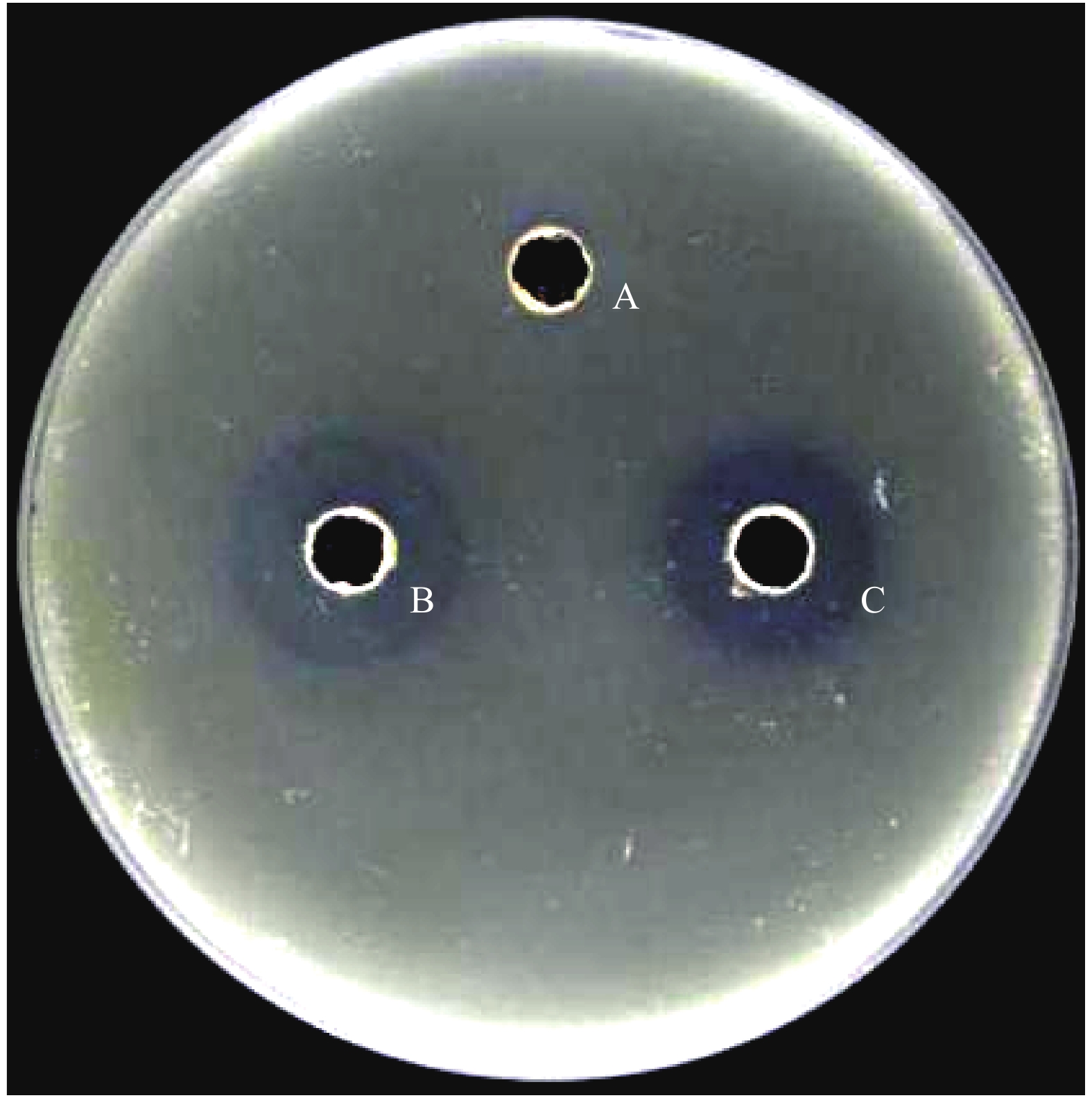
图 7 Bacillus amyloliquefaciens E3菌株脂肽粗提物对Dickeya zeae EC1的抑制作用
“*”、“**”分别表示0.05和0.000 1水平的差异显著性(t检验)
Figure 7. Inhibitory effect of crude lipopeptides extracted from Bacillus amyloliquefaciens E3 strain against Dickeya zeae EC1
“*”,“**” indicate significance differences at the levels of 0.05 and 0.000 1 respectively (t test)
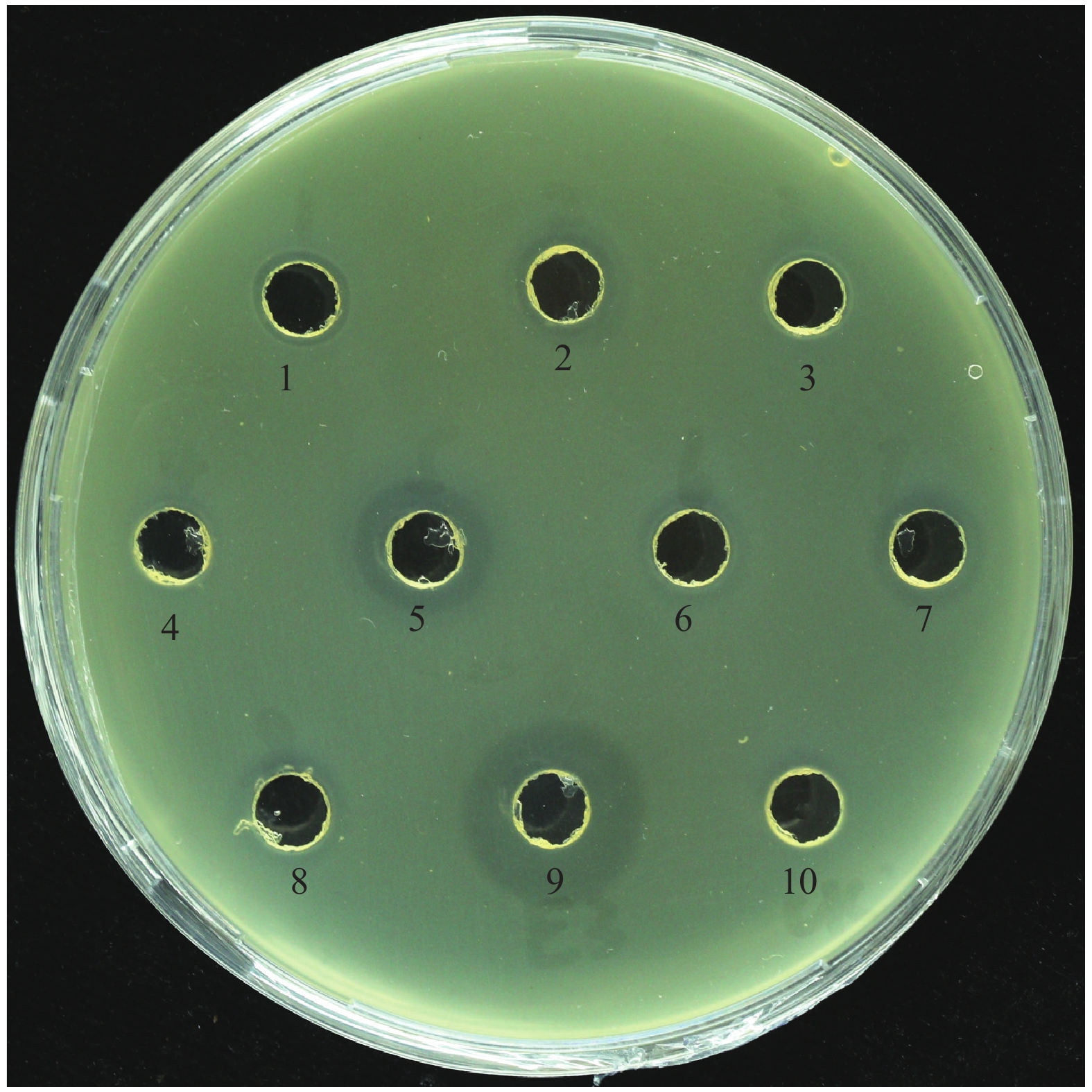
图 8 Bacillus amyloliquefaciens E3菌株的抗真菌谱
A:茄病镰刀菌Fusarium solani;B:黄曲霉Aspergillus flavus;C:黑曲霉Aspergillus niger;D:雪腐镰刀菌Fusarium nival;E:大肠埃希菌Escherichia coli;F:柑橘溃疡菌Xanthomonas citri pv. citri;G:水稻基腐病菌 Dickeya zeae EC1;H:茄科劳尔氏菌Ralstonia solanacearum;I:洋葱伯克氏菌Burkholderia cenocepacia;J:香蕉基腐病菌Dickeya zeae MS1
Figure 8. The antimicrobial spectrum of strain Bacillus amyloliquefaciens E3 strain
表 1 水稻基腐病菌拮抗菌 E3菌株的主要生理生化特性
Table 1 The main physiological and biochemical characteristics of Bacillus amyloliquefaciens E3 strain
测定指标
Detected index生化反应
Biochemical
reaction测定指标
Detected index生化反应
Biochemical
reaction接触酶 Contact enzyme 阳性 Positive 苯丙氨酸脱氢酶 Phenylalanine dehydrogenase 阴性 Negative 氧化酶 Oxidase 阴性 Negative 2%(w) NaCl耐盐试验 Salt tolerance test with 2% NaCl 阳性 Positive 明胶水解 Gelatin hydrolysis 阴性 Negative 5%(w) NaCl耐盐试验 Salt tolerance test with 5% NaCl 阳性 Positive V-P试验 V-P test 阳性 Positive 7%(w) NaCl耐盐试验 Salt tolerance test with 7% NaCl 阳性 Positive 丙酸盐利用 Propionate utilization 阴性 Negative 10%(w) NaCl耐盐试验 Salt tolerance test with 10% NaCl 阳性 Positive 脲酶试验 Urease test 阴性 Negative 淀粉水解 Starch hydrolysis 阳性 Positive 吲哚试验 Indole test 阴性 Negative 葡萄糖产酸 Glucose acidogenesis 阳性 Positive 硝酸盐还原 Nitrate reduction 阳性 Positive 柠檬酸盐利用 Citrate utilization 阳性 Positive 酪氨酸水解 Tyrosine hydrolysis 阴性 Negative 酪素水解 Casein hydrolysis 阳性 Positive pH5.7肉汤 pH5.7 Broth 阳性 Positive pH6.8肉汤 pH6.8 Broth 阳性 Positive -
[1] 冯成玉. 水稻细菌性基腐病发生情况与研究进展[J]. 中国稻米, 2009(4): 21-23. doi: 10.3969/j.issn.1006-8082.2009.04.007 [2] 刘琼光, 张庆, 魏楚丹. 水稻细菌性基腐病研究进展[J]. 中国农业科学, 2013, 46(14): 2923-2931. doi: 10.3864/j.issn.0578-1752.2013.14.008 [3] PU X M, ZHOU J N, LIN B R, et al. First report of bacterial foot rot of rice caused by a Dickeya zeae in China[J]. Plant Disease, 2012, 96(12): 1818.
[4] 张蝶. 浙江省水稻细菌性基腐病发生因素及化学防治研究[D]. 金华: 浙江师范大学, 2016. [5] SANSINENEA E, VACA J, ELENA ROJAS N, et al. A wide spectrum of antibacterial activity of secondary metabolites from Bacillus amyloliquefaciens ELI149[J]. Bioscience Journal, 2020, 36(1): 235-244.
[6] BEN ABDALLAH D, TOUNSI S, GHARSALLAH H, et al. Lipopeptides from Bacillus amyloliquefaciens strain 32a as promising biocontrol compounds against the plant pathogen Agrobacterium tumefaciens[J]. Environmental Science and Pollution Research, 2018, 25(36): 36518-36529.
[7] STEIN T. Bacillus subtilis antibiotics: Structures, syntheses and specific functions[J]. Molecular Microbiology, 2005, 56(4): 845-857. doi: 10.1111/j.1365-2958.2005.04587.x
[8] WU L M, WU H J, CHEN L N, et al. Difficidin and bacilysin from Bacillus amyloliquefaciens FZB42 have antibacterial activity against Xanthomonas oryzae rice pathogens[J]. Scientific Reports, 2015, 5(1). doi: http://doi.org/10.1038/srep12975.
[9] LIU Y F, CHEN Z Y, NG T B, et al. Bacisubin, an antifungal protein with ribonuclease and hemagglutinating activities from Bacillus subtilis strain B-916[J]. Peptides, 2007, 28(3): 553-559. doi: 10.1016/j.peptides.2006.10.009
[10] TAO Y, BIE X M, LV F X, et al. Antifungal activity and mechanism of fengycin in the presence and absence of commercial surfactin against Rhizopus stolonifer[J]. Journal of Microbiology, 2011, 49(1): 146-150. doi: 10.1007/s12275-011-0171-9
[11] CHO S J, LEE S K, CHA B J, et al. Detection and characterization of the Gloeosporium gloeosporioides growth inhibitory compound iturin A from Bacillus subtilis strain KS03[J]. FEMS Microbiology Letters, 2003, 223(1): 47-51. doi: 10.1016/S0378-1097(03)00329-X
[12] CAO H Y, JIAO Y, YIN N, et al. Analysis of the activity and biological control efficacy of the Bacillus subtilis strain Bs-1 against Meloidogyne incognita[J]. Crop Protection, 2019, 122: 125-135. doi: 10.1016/j.cropro.2019.04.021
[13] ZHAO P, QUAN C, WANG Y, et al. Bacillus amyloliquefaciens Q-426 as a potential biocontrol agent against Fusarium oxysporum f. sp spinaciae[J]. Journal of Basic Microbiology, 2014, 54(5): 448-456. doi: 10.1002/jobm.201200414
[14] ARGUELLES-ARIAS A, ONGENA M, HALIMI B, et al. Bacillus amyloliquefaciens GA1 as a source of potent antibiotics and other secondary metabolites for biocontrol of plant pathogens[J]. Microbial Cell Factories, 2009, 8: 63.
[15] ONGENA M, JACQUES P. Bacillus lipopeptides: Versatile weapons for plant disease biocontrol[J]. Trends in Microbiology, 2008, 16(3): 115-125. doi: 10.1016/j.tim.2007.12.009
[16] CAWOY H, MARIUTTO M, HENRY G, et al. Plant defense stimulation by natural isolates of Bacillus depends on efficient surfactin production[J]. Molecular Plant-Microbe Interactions, 2014, 27(2): 87-100. doi: 10.1094/MPMI-09-13-0262-R
[17] ZHOU J, CHENG Y, LV M, et al. The complete genome sequence of Dickeya zeae EC1 reveals substantial divergence from other Dickeya strains and species[J]. BMC Genomics, 2015, 16(1): 571. doi: 10.1186/s12864-015-1545-x
[18] 东秀珠, 蔡妙英. 常见细菌系统鉴定手册[M]. 北京: 科学出版社, 2001: 267-294. [19] 布坎南, 等. 伯杰细菌鉴定手册[M]. 北京: 科学出版社, 1984: 729-758. [20] REDBURN A C, PATEL B K. Phylogenetic analysis of Desulfotomaculum thermobenzoicum using polymerase chain reaction-amplified 16S rRNA-specific DNA[J]. FEMS Microbiology Letters, 1993, 113(1): 81-86. doi: 10.1111/j.1574-6968.1993.tb06492.x
[21] 刘勇, 毛爱军, 李辉, 等. 枯草芽孢杆菌群β-甘露聚糖酶基因克隆及同源性分析[J]. 基因组学与应用生物学, 2009, 28(5): 845-850. doi: 10.3969/gab.028.000845 [22] 韩玉竹, 邓钊, 张宝, 等. 解淀粉芽孢杆菌H15产抗菌肽的发酵条件优化和提取方法比较研究[J]. 食品科学, 2015, 36(15): 135-141. doi: 10.7506/spkx1002-6630-201515025 [23] 张睿佳, 胡杰, 陆建忠, 等. 不同成熟度对水稻种子萌发的影响[J]. 上海农业科技, 2017(5): 62-63. doi: 10.3969/j.issn.1001-0106.2017.05.029 [24] LI S B, XU S R, ZHANG R N, et al. The antibiosis action and rice-induced resistance, mediated by a lipopeptide from Bacillus amyloliquefaciens B014, in controlling rice disease caused by Xanthomonas oryzae pv. oryzae[J]. Journal of Microbiology and Biotechnology, 2016, 26(4): 748-756. doi: 10.4014/jmb.1510.10072
[25] 朱弘元, 康健, 范昕, 等. 解淀粉芽孢杆菌B15产脂肽的分离鉴定及抑菌机理[J]. 江苏农业科学, 2016, 44(5): 186-189. [26] 张玉胜, 谭亚男. 油菜核盘菌拮抗菌的筛选及鉴定[J]. 生命科学仪器, 2009, 7(8): 27-28. [27] 陈华, 王丽, 袁成凌, 等. 高效液相色谱-电喷雾质谱法分离和鉴别枯草芽孢杆菌产生的脂肽类化合物[J]. 色谱, 2008(3): 343-347. doi: 10.3321/j.issn:1000-8713.2008.03.016 [28] CHEN M C, WANG J P, ZHU Y J, et al. Antibacterial activity against Ralstonia solanacearum of the lipopeptides secreted from the Bacillus amyloliquefaciens strain FJAT-2349[J]. Journal of General and Applied Microbiology, 2019, 126(5): 1519-1529. doi: 10.1111/jam.14213
[29] 周海亮. 水稻细菌性基腐病病原学及检测技术的研究[D]. 武汉: 华中农业大学, 2012. [30] VATER J, GAO X, HITZEROTH G, et al. "Whole cell"--matrix-assisted laser desorption ionization-time of flight-mass spectrometry, an emerging technique for efficient screening of biocombinatorial libraries of natural compounds-present state of research[J]. Combinatorial Chemistry& High Throughput Screening, 2003, 6(6): 557-567.
[31] AERON A, KHARE E, JHA C K, et al. Revisiting the plant growth-promoting rhizobacteria: Lessons from the past and objectives for the future[J]. Archives of Microbiology, 2020, 202(4): 665-676. doi: 10.1007/s00203-019-01779-w
[32] GOUDA S, KERRY R G, DAS G, et al. Revitalization of plant growth promoting rhizobacteria for sustainable development in agriculture[J]. Research in Microbiology, 2018, 206: 131-140. doi: 10.1016/j.micres.2017.08.016
[33] KHAYALETHU N, LESIBA K L, MOLEMI E R, et al. The mode of action of bacillus species against Fusarium graminearum, tools for investigation, and future prospects[J]. Toxins, 2019, 11(10). doi: http://doi.org/10.3390/toxins11100606.
[34] 严婉荣, 肖敏, 陈圆, 等. 解淀粉芽孢杆菌抗菌脂肽研究进展[J]. 北方园艺, 2018(7): 162-167. [35] FAN B, WANG C, SONG X, et al. Bacillus velezensis FZB42 in 2018: The gram-positive model strain for plant growth promotion and biocontrol[J]. Frontiers in Microbiology, 2018, 9: 2491. doi: 10.3389/fmicb.2018.02491
[36] CHOWDHURY S P, UHL J, GROSCH R, et al. Cyclic lipopeptides of Bacillus amyloliquefaciens subsp. plantarum colonizing the lettuce rhizosphere enhance plant defense responses toward the bottom rot pathogen Rhizoctonia solani[J]. Molecular Plant-Microbe Interactions, 2015, 28(9): 984-995. doi: 10.1094/MPMI-03-15-0066-R
[37] 向亚萍, 周华飞, 刘永锋, 等. 解淀粉芽孢杆菌B1619脂肽类抗生素的分离鉴定及其对番茄枯萎病菌的抑制作用[J]. 中国农业科学, 2016, 49(15): 2935-2944. doi: 10.3864/j.issn.0578-1752.2016.15.008 [38] FIRA D, DIMKIC I, BERIC T, et al. Biological control of plant pathogens byBacillus species[J]. Biotechnology Journal, 2018, 285: 44-55. doi: 10.1016/j.jbiotec.2018.07.044
[39] ZERIOUH H, ROMERO D, GARCIA-GUTIERREZ L, et al. The iturin-like lipopeptides are essential components in the biological control arsenal of Bacillus subtilis against bacterial diseases of cucurbits[J]. Molecular Plant-Microbe Interactions, 2011, 24(12): 1540-1552. doi: 10.1094/MPMI-06-11-0162
[40] CAO Y, PI H L, CHANDRANGSU P, et al. Antagonism of two plant-growth promoting Bacillus velezensis isolates against Ralstonia solanacearum and Fusarium oxysporum[J]. Scientific Reports, 2018, 8(1). doi: http://doi.org/10.1038/S41598-018-22782-Z.
[41] HADIZADEH I, PEIVASTEGAN B, HANNUKKALA A, et al. Biological control of potato soft rot caused by Dickeya solani and the survival of bacterial antagonists under cold storage conditions[J]. Plant Pathology, 2019, 68(2): 297-311. doi: 10.1111/ppa.12956
[42] CHEN J, LIU T, WEI M, et al. Macrolactin a is the key antibacterial substance of Bacillus amyloliquefaciens D2WM against the pathogen Dickeya chrysanthemi[J]. European Journal of Plant Pathology, 2019, 155(2): 393-404. doi: 10.1007/s10658-019-01774-3
[43] LI J, HU M, XUE Y, et al. Screening, identification and efficacy evaluation of antagonistic bacteria for biocontrol of soft rot disease caused by Dickeya zeae[J]. Microorganisms, 2020, 8(6975). doi: 10.3390/microorganisms8050697.




 下载:
下载: