Identification and genetic evolution analysis of astrovirus RdRpgene of Alopexlagopus
-
摘要:目的
探究广州动物园一例死亡白狐Alopex lagopus肾脏中鉴定出的星状病毒的遗传变异情况。
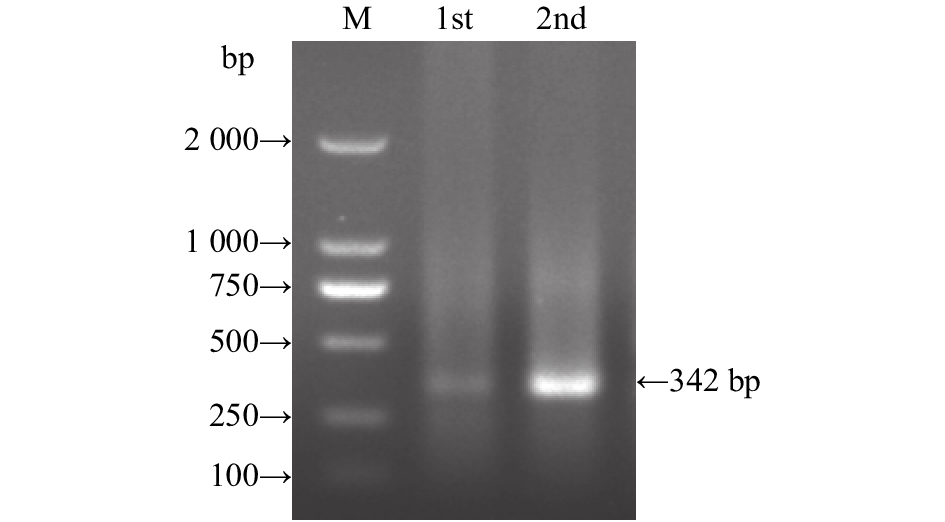
方法对死亡白狐进行剖检,利用半巢式RT-PCR对内脏器官进行病原学检测,对扩增出的病毒株的RdRp基因序列进行相似性分析。
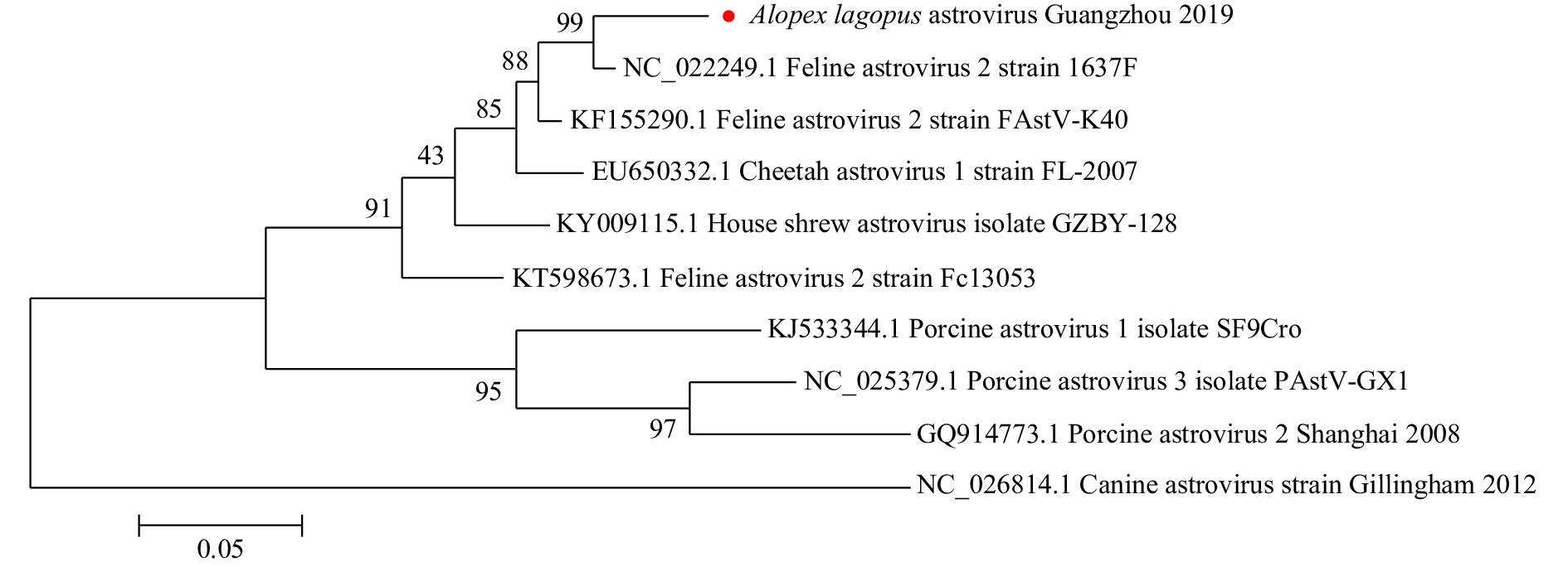
结果死亡白狐的肾脏苍白肿大,被膜难以剥离,肾脏中检测出的星状病毒RdRp基因呈阳性。RdRp基因与9株参考毒株的核苷酸相似性为67.5%~96.2%,其中与香港猫源星状病毒1637F的核苷酸相似性(96.2%)最高。
结论本研究为星状病毒在野生哺乳动物间的跨种传播及肠外器官中的感染提供了依据。
Abstract:ObjectiveTo investigate the genetic variation of astrovirus identified in the kidney of a dead Alopex lagopus in Guangzhou Zoo.
MethodThe autopsy was performed on the dead A. lagopus, and the etiology detection of the internal organs was determined by semi-nested RT-PCR. The amplified RdRp gene sequence of the virus strain was used for similarity analysis.
ResultThe kidney of the dead A. lagopus were pale, enlarged and the capsule was difficult to be stripped off. The astrovirus RdRp gene in kidney showed positive. The astrovirus RdRp gene had nucleotide similarities of 67.5%−96.2% with nine referenced astrovirus strains from other animals, and was most closely related to the feline astrovirus 2 strain 1637F isolated in Hong Kong (96.2% similarity).
ConclusionThe study enlarges the knowledge of the interspecies transmission of astrovirus among wild mammals, also proves the possibility of extraintestinal infection caused by astrovirus.
-
Keywords:
- Alopex lagopus /
- astrovirus /
- kidney /
- wild mammal /
- RdRp gene
-
化学除草是目前最有效且广泛采用的水稻田杂草管控方式[1],常用的方法是对整个作业区域均匀和覆盖式的喷洒除草[2],以无序的“点状”或“片状”形式存在的杂草会导致农药喷洒过量。“精准农业航空”中基于遥感分析的精准施药为解决农药过量施用问题提供了一种新思路[3-4]。精准施药的首要任务是获取水稻田的遥感图像并分析其中的杂草分布状况,得到网格形状的杂草分布图。水稻田图像中水稻及杂草颜色相近,若依据图像识别其差异,则要求遥感图像具有较高的空间分辨率。近年来无人机低空遥感因操作简单、节省人力物力并可获得高分辨率影像的优势为农作物分类调查提供了新思路[5],相比传统卫星影像,具有获取方式更灵活、时效高等特点,在农情监测、精准农业等领域得到越来越多的应用[6-7]。利用无人机采集水稻田的遥感图像并进行农情分析在国内外已有类似的研究。Barrero等[8]使用神经网络(Neural network)对水稻杂草航空图像进行检测,检测准确率高达99%;Barrero等[9]根据无人机拍摄的图像提出一种融合低分辨率多光谱和高分辨率RGB图像的方法检测水稻田杂草,采用神经网络对RGB、RGB+NGRDI和融合RGB-NGRDI 共3种图像进行检测,结果表明基于融合图像的杂草检测精度最高,在70%~85%之间;Uto等[10]使用无人机获取水稻的高光谱数据,通过频繁地监测照明水平和叶绿素指数获得水稻田的生长信息;马明洋[11]使用无人机获取稻田中水稻冠层高光谱影像信息,对光谱特征信息进行分类,其中使用二代小波分解算法分类精度最高,达到90.36%;洪雪[12]利用无人机获得农田中水稻的光谱数据,使用BP神经网络建模对水稻产量进行预测;Ma等[13]使用SegNet、FCN和U-Net 共3种语义分割模型对水稻田中的水稻杂草图像进行像素级的分割,结果表明,SegNet算法识别效果最好,达到92.7%的准确率。相关研究主要集中在对水稻田杂草的识别检测上,获取水稻田中杂草分布信息并生成水稻田间杂草分布图的研究相对较少。本文拟采用大疆精灵4无人机及其携带的可见光相机采集遥感图像,以水稻杂草为识别目标,对比支持向量机(Support vehicle machine, SVM)、K最近邻算法(K-nearest neighbor, KNN)、AdaBoost共3种机器学习方法和卷积神经网络(Convolutional neural network,CNN)方法对水稻和杂草的分类效果,获取水稻田间杂草分布信息,生成分类效果最好的水稻田间杂草分布图,为后续植保无人机进行精准施药提供决策依据。
1. 材料与方法
1.1 供试材料
研究区域位于华南农业大学增城试验基地(中国广东省广州市,坐标23°14′N、113°38′E,基准WGS84)(图1)。本研究于2017年10月2日对试验基地中某一块稻田进行RGB图像采集,数据采集设备为大疆精灵4无人机,结合地面站软件DJI GS PRO进行航线规划。经实地验证,研究区域的杂草主要有千金子和莎草。
无人机飞行高度依次设置为6、8、10 m。横、纵向重叠率分别为60%和50%。本次试验采集图像120幅,每幅图像的分辨率为4 000像素×3 000像素,空间分辨率为0.3 cm。采集到的图像如图2a所示。根据人眼对水稻和杂草的识别效果以及需要对采集的图像使用有监督学习作用的方法进行标注,试验选择飞行高度为6 m。在采集到的无人机遥感图像中,所有物体被分为3个类别:水稻、杂草以及其他(包括水泥地、土壤、地面等)。本文分别使用绿色表示水稻,红色表示杂草,灰色代表其他,对每一张图像中3个类别进行基于像素级别的颜色标注,如图2b所示。
根据田间地面的实际验证,在1个0.3 m×0.3 m的网格中有95%以上概率可确认只有1个类别。考虑有限的计算机资源以及图像的分辨率即像素尺寸为4 000×3 000,空间分辨率为0.3 cm,根据计算,0.3 m×0.3 m的地面区域对应图像的像素尺寸为100×100,因此本文将每幅4 000像素×3 000像素的原图像与标记图像分别切割成1 200块像素大小为100×100的小网格。对原图像切割成的小网格图片与标记图切割成的小网格图片进行效果比对,在标记图小网格图库中,按照单一颜色或者某种颜色超过95%的选取规则与原图像库中的小图片进行对应,按照随机性以及训练集与测试集5∶1的原则选出6 000幅小网格图片作为训练集,选取1 200幅小网格图片作为测试集。
1.2 数据预处理
1.2.1 基于机器学习分类的图像预处理
颜色特征是基于图像像素提取的,具有旋转、尺度和平移不变性等优点[14],是人眼获取的最为有效且明显的信息之一。它的获取及计算较简单,常用作特征向量训练机器学习模型。本试验拟对小样本提取RGB三通道颜色的最大值、最小值与平均值。从原图小网格图库中随机选取3种类别小图片各3 200幅用于统计RGB像素平均值(图3),不同类别样本提取出的颜色特征有一些差异。
本文的图像属于单视图数据,数据维数巨大。尽管高维数据含有更加丰富的信息,但对于分类、聚类等任务,要付出巨大的计算和存储代价,而且在测试集数量一定时,特征维数的增加将使样本统计特性变得困难,降低分类器的泛化能力[15]。本文使用对单视图数据有较好泛化性能的主成分分析(Principal component analysis,PCA)对图像数据进行降维。PCA是Karl Pearson[16]在1901年提出的概念,1933年Hotelling[17]完善其数学基础。本研究原始图像的特征向量有30 000维存在噪声和冗余信息,会对分类器造成干扰,因此采用PCA降维来消除这种影响[18]。经过如图4所示的PCA计算流程,为保留90%有效信息并降到50维,剔除无效的信息,保留有用的特征信息,形成特征向量集。结合提取到的颜色特征分别送入SVM、KNN和AdaBoost 3种分类器中进行识别分类。
1.2.2 基于CNN分类的图像预处理
CNN可以自动学习输入图像中的特征,并对学习到的特征进行自动分类,网络直接输出该图像的类别概率,是一种端到端的图像识别方法[19]。CNN通过卷积运算完成特征提取。图像卷积运算是通过设定各种特征提取滤波器矩阵,然后使用该卷积核在原图像矩阵(图像实际是像素值构成的矩阵)“滑动”,实现卷积运算。因此,基于CNN对图像的分类无需做预处理,可直接进行模型的训练。本文的技术路线如图5所示。对原始图像进行标注、切割后使用3种机器学习算法和CNN对小图片进行分类,并形成水稻田中的杂草分布图。
1.3 分类方法介绍
常用的监督分类方法有最大似然[20]、神经网络[21-22]、SVM[23-24],常用的非监督分类方法有K−均值、模糊C−均值(Fuzzy C-mean,FCM)[25-26]等。获取的图像数据经标注后使得本文的分类方法成为监督分类范畴。
1.3.1 SVM分类模型
SVM是一种可分析数据、识别模型和分类的监督学习模型[27-28]。SVM算法在于寻找一个超平面,该超平面可将训练集中的数据分开,且与类域边界边沿垂直于该超平面方向的距离最大,从而达到最大的泛化能力[29]。对于任意数据集
$\mathbb T $ 和超平面${\boldsymbol{\omega}} {{x + {\boldsymbol{b}} = 0}}$ ,定义超平面关于样本点$({{{x}}_{{i}}},\ {{{y}}_{{i}}})$ 的几何间距(ri)为:$${\gamma _{{i}}}{\rm{ = }}{{{y}}_{{i}}}\Bigg(\frac{{\boldsymbol{\omega}} }{{\parallel {\boldsymbol{\omega}} \parallel }} x_i{\rm{ + }}\frac{{\boldsymbol{b}}}{{\parallel {\boldsymbol{\omega}} \parallel }}\Bigg),$$ (1) 式中,
${\boldsymbol{\omega}}$ 、b分别是超平面的权重向量与截距向量。关于全部样本点的几何间隔的最小值$(\gamma) $ 为:$$\gamma {\rm{ = min}}{\gamma _i}(i = 1,2\cdots ,n),$$ (2) 即向量到超平面的距离。
以上都是针对数据线性可分推导的,本文的图像数据属于线性不可分,必须确定惩罚系数c和径向基函数g,再选择更优的核函数将数据映射到更高维空间,有效解决数据低维和高维空间映射造成计算复杂度增加的问题[30]。常用的核函数有线性核、多项式核、径向基内核和Sigmoid核。选择核函数时要考虑数据的特点和数据量,本次SVM模型使用的是Sigmoid核,通过使用不同的核函数和参数c、g寻优,Sigmoid核的公式如下所示:
$$K(x,y) = \tanh (\mathop {{{a}}x}\nolimits^{\rm T} y + b),$$ (3) 式中,a、b为人工设置的参数,a为正实数,b为非负实数。
1.3.2 KNN分类模型
KNN是机器学习领域最常用的分类算法,也是一种回归的非参数统计方法[31]。优点是可用于非线性分类,适用于本文的遥感数据且对异常数据不敏感,缺点是需要大量的存储空间,计算耗时。分类的思路是假设一个样本在特征空间中的k个最近邻的样本大多数属于某个类别,则将该样本也划分成这个类别。本文使用KNN算法建模时,关于距离的度量方法使用的是欧几里得距离公式,具体如下:
$$D = \sqrt {\sum\limits_{i = 1}^n {{{({x_i} - {y_i})}^2}} } ,$$ (4) 式中,
${x_i}$ 代表待测样本,${y_i}$ 代表已知样本,n代表样本数。1.3.3 AdaBoost分类模型
本文选取AdaBoost算法构建小尺度的水稻田杂草图像分类模型。AdaBoost算法是由Freund等[32]提出的目前使用和研究最多的集成算法,该算法的思想是将若干个分类能力较弱的分类器按照一定的方式混合成为分类能力很强的强分类器[33-34]。AdaBoost算法计算流程如下:
1)构造图像特征数据训练集为G={
$({x_1}, {y_2}), ({x_2},{y_2})\cdots,({x_n},{y_n})$ },xn为图像的特征数据集,yn为标签集。2)初始化每个图像特征数据的权值D1={
${w_1},{w_2}\cdots,{w_n}$ },${w_n}$ =1/n,${w_n}$ 为第n个图像特征数据对应的初始权值。3)迭代k次(k=1, 2···, K),使用决策树算法对有权值分布的训练集训练出弱分类器。
1.3.4 CNN分类模型
作为一种深度学习方法,CNN已成为当前图像理解领域的研究热点[35],其权值共享网络结构在网络输入为多维图像时尤为方便,图像可直接作为网络输入,避免传统识别算法中复杂的特征提取和数据重建过程[36]。本文采用CNN对采集到的水稻田杂草图像进行分类,CNN结构的隐含层一般包含卷积层、采样层、池化层和全连接层等几类常见的层结构,本文使用的卷积层结构如图6所示。
卷积层中的卷积核包含权重系数,用来自动提取图像的特征。本文所使用的CNN结构是在Ubuntu16.04系统下,框架为tensorflow 1.5,编程语言为Python,训练集与测试集的图片分别为6 000与1 200幅。考虑到有限的计算机资源,卷积层只设置5个卷积核,每个卷积核大小为1,利用ReLU函数进行激活。池化层池化窗口大小为5×5,步长为2×2。将提取到的三维特征转化成一维向量,全连接层对提取到的特征进行分类。学习率设置为10−5,最后选用AdamOptimizer优化网络。
2. 结果与分析
2.1 算法的分类结果与分析
对相同的数据集经过颜色特征的提取和PCA降维后,将形成的特征向量集分别送入到SVM、KNN以及AdaBoost分类器中进行分类。对分类器的参数进行多次的调优试验。
试验结果表明,SVM算法对3种作物类别(水稻、杂草、其他)的综合分类精度为89.75%。图7展示的是KNN算法分类精度与k值的关系,当k取14时,KNN综合识别精度最高,为85.58%。AdaBoost分类器对水稻杂草图像的综合分类精度为90.25%,为3种机器学习算法中最高。3种模型的综合识别精度、混淆矩阵精度和运行时间如表1所示。
表 1 3个模型的混淆矩阵精度、运行时间和综合识别精度Table 1. Confusion matrix accuracy, run time and integrated recognition accuracy of three models模型
Model项目
Item精度/% Accuracy 运行时间/s
Run time综合识别精度/%
Integrated recognition accuracy真实为杂草
True weed真实为水稻
True rice真实为其他
True other objectSVM 预测为杂草 Predicted weed 78.04 10.98 10.98 0.500 4 89.75 预测为水稻 Predicted rice 7.07 92.60 0.32 预测为其他
Predicted other object2.40 0.90 96.69 KNN 预测为杂草 Predicted weed 87.80 4.47 0.07 2.209 2 85.58 预测为水稻 Predicted rice 16.88 82.32 0.80 预测为其他
Predicted other object9.94 0 90.06 AdaBoost 预测为杂草 Predicted weed 82.93 11.29 5.28 0.411 1 90.25 预测为水稻 Predicted rice 6.91 92.77 0.32 预测为其他
Predicted other object7.23 1.81 90.96 SVM、KNN和AdaBoost模型对测试集的测试运行时间分别为0.500 4、2.209 2和0.411 1 s。AdaBoost在分类精度和识别效率上都优于其他2种模型。基于CNN试验的模型是深度学习框架TensorFlow的GitHub代码库下的源码,通过对卷积层、池化层以及全连接层做了一些修改(图6)以适应本试验的数据,模型对整个数据集的综合分类精度是92.41%。图8是CNN训练的Loss值与训练步数的关系图。
2.2 生成水稻田间杂草分布图
分别使用这4种训练生成的模型对测试集小样本进行分类,将小样本分类识别结果返回到相应的原始图像中进行标记,4种模型下水稻田杂草分布图与原图、标注图的比较如图9所示。
由以上的试验结果效果图以及得到的分类精度可以得到,基于CNN对水稻杂草的分类效果要优于传统机器学习算法对水稻杂草的分类效果。机器学习算法及CNN均能够有效获取水稻田中杂草分布信息并生成水稻田间杂草分布图,有利于植保无人机对稻田中的杂草进行精准施药。
3. 讨论与结论
本文使用无人机采集水稻杂草RGB可见光图像,将数据切割成像素尺寸为100×100的小网格图像,对数据集进行颜色特征提取和降维后送入SVM、KNN和AdaBoost三种机器学习算法模型中进行分类。对比试验了3种机器学习方法和1种无需提取图像特征和降维的CNN分类方法。从分类的试验结果看,在3种机器学习算法中,不论是分类准确率还是分类效率,最优的分类器是AdaBoost,其综合识别精度为90.25%;在基于CNN的分类中,由于CNN结构可以自动挖掘数据的特征,简化人工提取环节,更具有兼容性和适用性,对不同的水稻杂草图像泛化能力更好,对水稻杂草图像的综合识别精度可以达到92.41%。试验结果还表明,针对无人机拍摄的遥感图像,机器学习算法及CNN能够有效识别出水稻和杂草,获取杂草的分布信息,生成水稻田间的杂草分布图,为后续植保无人机的精准施药提供依据。结合本文的研究和所得结果分析,本研究还存在以下待改进之处:1)本文使用的机器学习算法只提取图像的颜色特征作为模型训练的特征向量集进行训练,可以在后续研究中提取多个图像特征如纹理、形状等特征进行特征融合,结合机器学习算法并在此基础上改善算法模型,形成新算法以改善分类准确率;2)后续考虑采集多时期、多田地的水稻杂草图像,基于这些不同的水稻杂草图像改善模型,提高模型泛化能力;3)本文是进行水稻杂草的图像分类,切割成100像素×100像素小网格后,个别网格中避免不了水稻和杂草共存的情况,下一步可以考虑基于像素级别的分类,提高分类的精度。
-
表 1 引物序列信息
Table 1 Primer sequence information
名称Name 序列(5′→3′)Sequence asrRdRp1R GGYTTNACCCACATNCCAAA asrRdRp2F GAYTGGRCNCGNTWYGATGGNACIAT asrRdRp2R ARNCKRTCATCNCCATA 表 2 白狐肾脏星状病毒RdRp基因核苷酸相似性分析1)
Table 2 Nucleotide similarity analysis of astrovirus RdRp gene in kidney of Alopex lagopus
% 毒株Strain 2 3 4 5 6 7 8 9 10 1 79.8 94.6 89.3 80.8 77.3 90.5 96.2 92.1 67.5 2 82.0 81.4 86.4 90.9 84.5 81.4 83.0 67.8 3 93.1 83.3 79.8 94.3 97.2 96.2 69.1 4 82.0 79.5 92.4 91.8 93.1 69.7 5 85.5 83.3 82.3 82.3 66.2 6 80.8 78.9 80.4 67.2 7 92.7 93.1 69.4 8 94.6 67.8 9 69.1 1:白狐源星状病毒Guangzhou 2019,2:猪源星状病毒PAstV-GX1,3:猫源星状病毒FAstV-K40,4:猫源星状病毒Fc13053,5:猪源星状病毒SF9Cro,6:猪源星状病Shanghai 2008,7:家鼩源星状病毒GZBY-128,8:猫源星状病毒1637F,9:猎豹源星状病毒FL-2007,10:犬源星状病毒Gillingham 2012
1: Alopex lagopus astrovirus Guangzhou 2019, 2: Porcine astrovirus 3 isolate PAstV-GX1, 3: Feline astrovirus 2 strain FAstV-K40, 4: Feline astrovirus 2 strain Fc13053, 5: Porcine astrovirus 1 isolate SF9Cro, 6: Porcine astrovirus 2 Shanghai 2008, 7: House shrew astrovirus isolate GZBY-128, 8: Feline astrovirus 2 strain 1637F, 9: Cheetah astrovirus 1 strain FL-2007, 10: Canine astrovirus strain Gillingham 2012 -
[1] APPLETON H, HIGGINS P G. Letter: Viruses and gastroenteritis in infants[J]. Lancet, 1975, 1(7919): 1297.
[2] BOSCH A, PINTO R M, GUIX S. Human astroviruses[J]. Clinical Microbiology Reviews, 2014, 27(4): 1048-1074. doi: 10.1128/CMR.00013-14
[3] PANTIN-JACKWOOD M J, STROTHER K O, MUNDT E, et al. Molecular characterization of avian astroviruses[J]. Archives of Virology, 2011, 156(2): 235-244. doi: 10.1007/s00705-010-0849-z
[4] REUTER G, PANKOVICS P, BOROS A. Nonsuppurative (aseptic) meningoencephalomyelitis associated with neurovirulent astrovirus infections in humans and animals[J]. Clinical Microbiology Reviews, 2018, 31(4): e00040-18. doi: 10.1128/CMR.00040-18.
[5] WU W, XU R, LV Y, et al. Goose astrovirus infection affects uric acid production and excretion in goslings[J]. Poultry Science, 2020, 99(4): 1967-1974. doi: 10.1016/j.psj.2019.11.064
[6] ARRUDA B, ARRUDA P, HENSCH M, et al. Porcine astrovirus type 3 in central nervous system of swine with polioencephalomyelitis[J]. Emerging Infectious Diseases, 2017, 23(12): 2097-2100. doi: 10.3201/eid2312.170703
[7] XIAO C T, LUO Z, LV S L, et al. Identification and characterization of multiple porcine astrovirus genotypes in Hunan province, China[J]. Archives of Virology, 2017, 162(4): 943-952. doi: 10.1007/s00705-016-3185-0
[8] VU D L, CORDEY S, BRITO F, et al. Novel human astroviruses: Novel human diseases?[J]. Journal of Clinical Virology, 2016, 82: 56-63. doi: 10.1016/j.jcv.2016.07.004
[9] NAGAI M, OMATSU T, AOKI H, et al. Full genome analysis of bovine astrovirus from fecal samples of cattle in Japan: Identification of possible interspecies transmission of bovine astrovirus[J]. Archives of Virology, 2015, 160(10): 2491-2501. doi: 10.1007/s00705-015-2543-7
[10] DE BENEDICTIS P, SCHULTZ-CHERRY S, BURNHAM A, et al. Astrovirus infections in humans and animals: Molecular biology, genetic diversity, and interspecies transmissions[J]. Infection Genetics and Evolution, 2011, 11(7): 1529-1544. doi: 10.1016/j.meegid.2011.07.024
[11] QUAN P L, WAGNER T A, BRIESE T, et al. Astrovirus encephalitis in boy with X-linked agammaglobulinemia[J]. Emerging Infectious Diseases, 2010, 16(6): 918-925. doi: 10.3201/eid1606.091536
[12] WANG L, SHEN H, ZHENG Y, et al. Astrovirus in white-tailed deer, United States, 2018[J]. Emerging Infectious Diseases, 2020, 26(2): 374-376. doi: 10.3201/eid2602.190878
[13] MENDENHALL I H, SMITH G J D, VIJAYKRISHNA D. Ecological drivers of virus evolution: Astrovirus as a case study[J]. Journal of Virology, 2015, 89(14): 6978-6981. doi: 10.1128/JVI.02971-14
[14] MOSER L A, SCHULTZ-CHERRY S. Pathogenesis of astrovirus infection[J]. Viral Immunology, 2005, 18(1): 4-10. doi: 10.1089/vim.2005.18.4
[15] SELIMOVIC-HAMZA S, BOUZALAS I G, VANDEVELDE M, et al. Detection of astrovirus in historical cases of European sporadic bovine encephalitis, Switzerland 1958-1976[J]. Frontiers in Veterinary Science, 2016, 3: 91. doi: 10.3389/fvets.2016.00091.
[16] BLOMSTROM A L, WIDEN F, HAMMER A S, et al. Detection of a novel astrovirus in brain tissue of mink suffering from shaking mink syndrome by use of viral metagenomics[J]. Journal of Clinical Microbiology, 2010, 48(12): 4392-4396. doi: 10.1128/JCM.01040-10
[17] BOROS A, ALBERT M, PANKOVICS P, et al. Outbreaks of neuroinvasive astrovirus associated with encephalomyelitis, weakness, and paralysis among weaned pigs, Hungary[J]. Emerging Infectious Diseases, 2017, 23(12): 1999-2010.
[18] YOKOYAMA C C, LOH J, ZHAO G, et al. Adaptive immunity restricts replication of novel murine astroviruses[J]. Journal of Virology, 2012, 86(22): 12262-12270. doi: 10.1128/JVI.02018-12
[19] ZHANG H H, QIU Q G, LIU S J, et al. Genomic characterization of a novel astrovirus identified in Amur tigers from a zoo in China[J]. Archives of Virology, 2019, 164(12): 3151-3155. doi: 10.1007/s00705-019-04435-2
[20] YI S, NIU J, WANG H, et al. Molecular characterization of feline astrovirus in domestic cats from northeast China[J]. PLoS One, 2018, 13(10): e205441. doi: 10.1371/journal.pone.0205441.
[21] VU D L, BOSCH A, PINTO R M, et al. Epidemiology of classic and novel human astrovirus: Gastroenteritis and beyond[J]. Viruses, 2017, 9(2): 33. doi: 10.3390/v9020033.
-
期刊类型引用(8)
1. 夏晶晶. 基于KNN神经网络的无人机作业操控系统研究. 农机化研究. 2024(07): 183-187 .  百度学术
百度学术
2. 文飞,莫嘉维,胡宇琦,兰玉彬,陈欣,陆健强,邓小玲. 基于卷积神经网络遥感图像的荔枝花期长势评估. 华南农业大学学报. 2023(01): 123-133 .  本站查看
本站查看
3. 曲福恒,李婉婷,杨勇,刘红玉,郝忠林. 基于图像增强和注意力机制的作物杂草识别. 计算机工程与设计. 2023(03): 815-821 .  百度学术
百度学术
4. 陈裕锋,冯佩雯,凌金生,张书琪,余振鹏,熊琳琳,刘洪,谢家兴. 基于无人机多光谱图像的水稻品种鉴定. 南京农业大学学报. 2023(05): 995-1003 .  百度学术
百度学术
5. 张楠楠,张晓,白铁成,袁新涛,马瑞,李莉. 基于无人机可见光影像的新疆棉田田间尺度地物识别. 农业机械学报. 2023(S2): 199-205 .  百度学术
百度学术
6. 曹英丽,赵雨薇,杨璐璐,李静,秦列列. 基于改进DeepLabv3+的水稻田间杂草识别方法. 农业机械学报. 2023(12): 242-252 .  百度学术
百度学术
7. 焦晋康,胡炼,陈高隆,涂团鹏,王志敏,臧英. 水田行间除草装置设计与试验. 农业工程学报. 2023(24): 11-22 .  百度学术
百度学术
8. 卢征. 国土资源数据整合下无人机低空遥感监测系统设计. 电子设计工程. 2022(13): 78-81+86 .  百度学术
百度学术
其他类型引用(5)




 下载:
下载: