Research progress of grain shape genetics in rice
-
摘要:
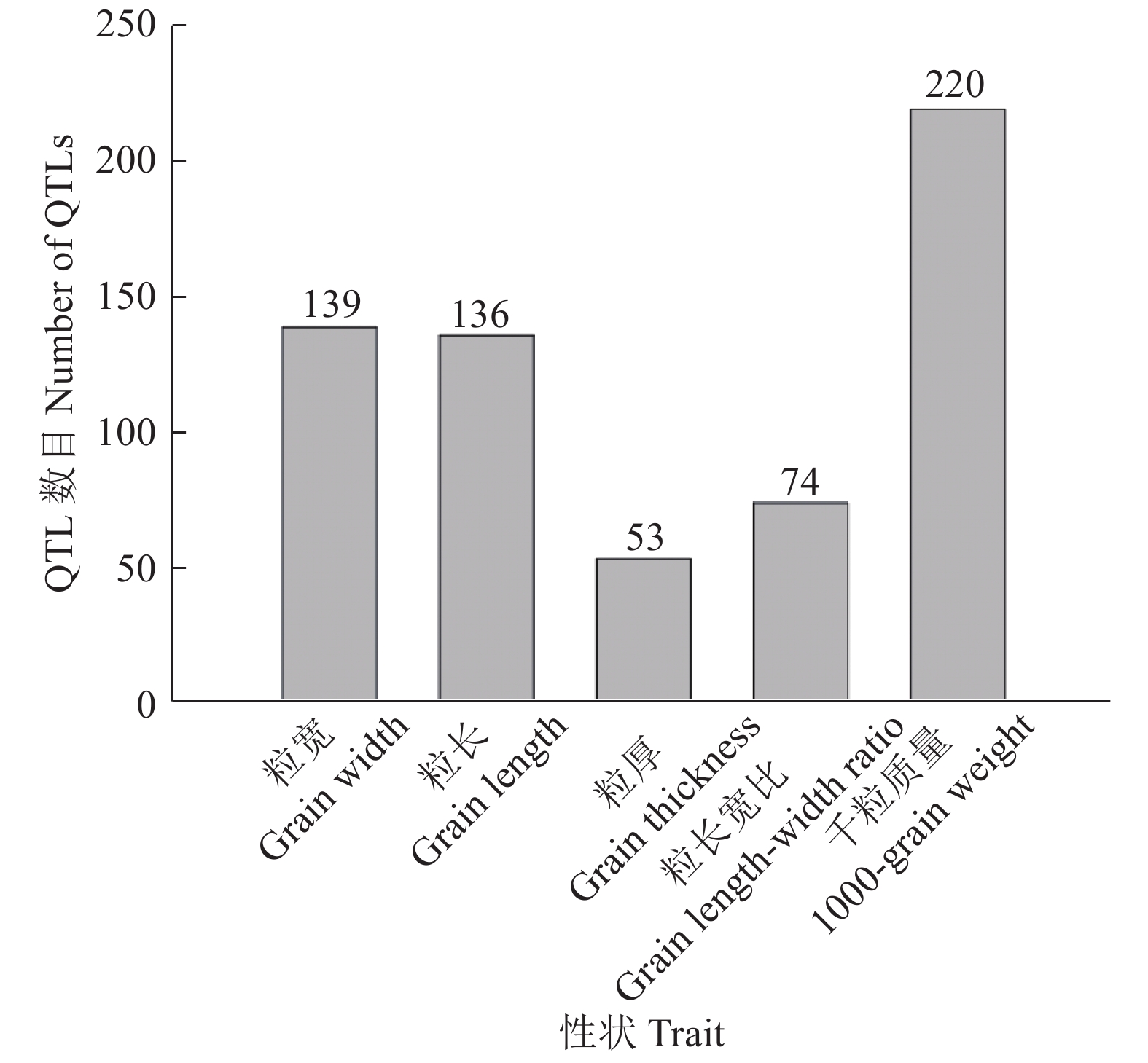
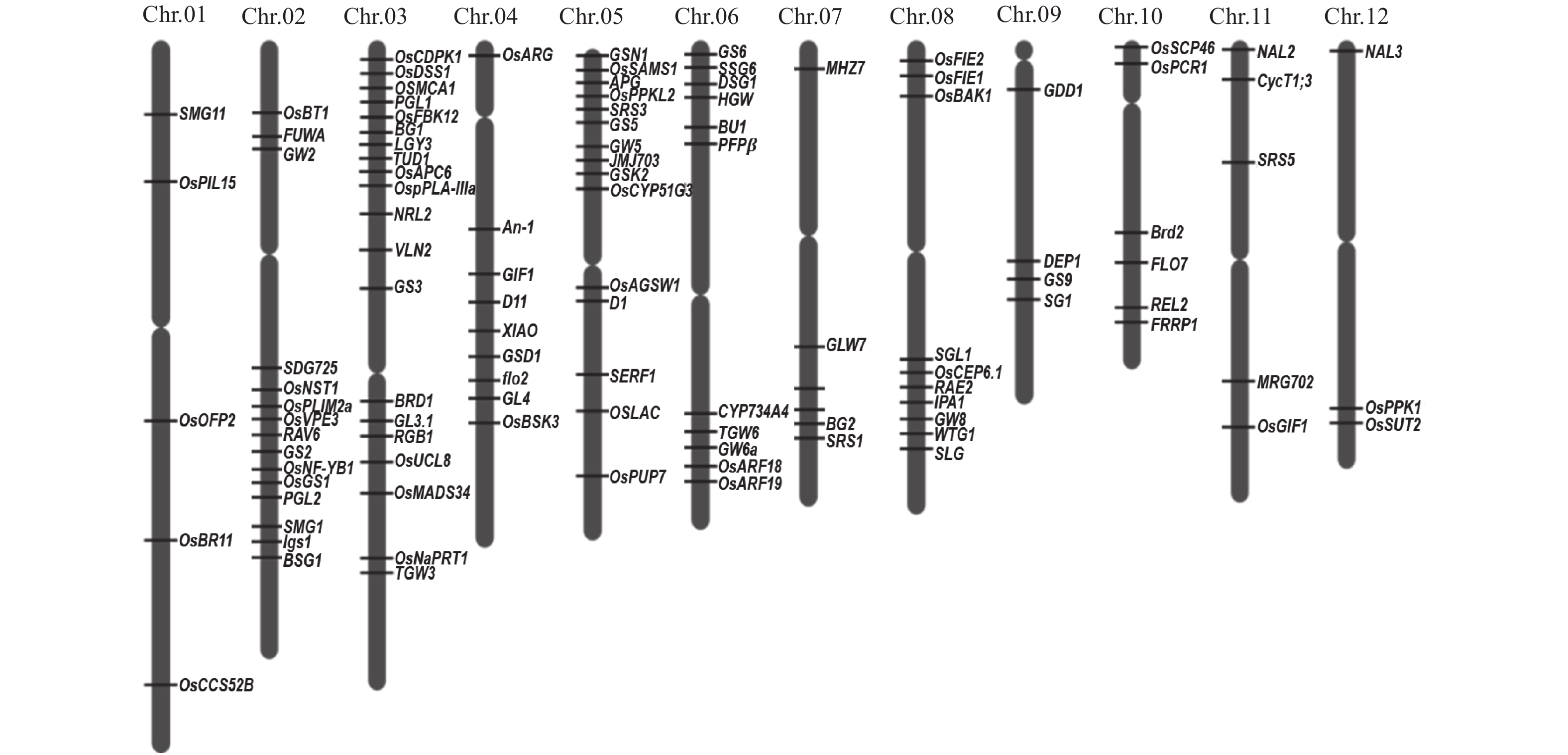
粒形不仅是影响水稻产量的要素,还与稻米的外观品质密切相关,是决定稻米品质的重要因素。随着现代遗传学和基因组学相关理论和技术的发展,人们对粒形的遗传研究日趋深入,目前已有数十个控制水稻粒形的基因被克隆。本文概述了水稻粒形遗传研究主要进展,同时也指出了水稻粒形遗传研究中存在的主要问题,分析了粒形遗传研究的前景。
Abstract:Grain shape controls rice yield. Meanwhile, grain shape is related to rice appearance quality, and an important element determing rice quality. With the developments of modern genetics and genomics related theories and technologies, the genetic researches on grain shape are gradually deepened. Now dozens of QTLs controlling grain shape are cloned. This review summarizes the main progresses of genetic researches on grain shape, points out the major problems and analyzes the prospects.
-
Keywords:
- rice /
- grain shape /
- genetic /
- functional gene /
- molecular breeding
-
苹果作为一种常见的水果,其质量问题直接关系到消费者的健康和生产者的经济利益。我国是苹果种植大国,苹果采摘、运送、储藏与加工处理是不可忽视的关键环节[1]。然而,由于人工质检的主观性和效率低下,传统的苹果质检方式已经无法满足现代农产品质量的需求[2]。基于机器学习的苹果识别技术应运而生,该方法可以对苹果进行自动识别和分类。李大华等[3]针对自然复杂环境下的苹果重叠问题,利用谱聚类算法进行分割,然后使用随机霍夫变换实现果实的识别和定位,尽管改进后的谱聚类算法在计算量上有所优化,但仍然涉及多步骤的处理,包括均值漂移预分割、稀疏矩阵构建、K-means分类和随机霍夫变换,整体流程较为复杂,对硬件性能要求较高,不适合实时性要求较高的应用场景。王迎超等[4]为了准确快速实现多特征融合的苹果分级,提出了一种基于K-means聚类和改进多层感知器(Multilayer perception, MLP)的苹果分级方法,但特征权重的确定存在主观性,且在不同的应用场景或苹果品种中特征的重要性会有所不同,导致分级准确性下降。宋怡焕等[5]提出基于最小二乘支持向量机(LS-SVM)检测苹果果梗/花萼缺陷,试验中仅使用了 180 幅苹果图像,而训练数据的不足会导致模型的泛化能力受限,对不同环境或苹果品种的误判率较高。上述传统的机器学习算法试验设计过于繁琐,对复杂的特征提取和分类能力不佳,易受外界因素的干扰,难以获得较好的检测结果。随着深度学习技术突飞猛进,以深度学习为基础的目标检测技术在苹果果实检测[6]、病虫害监测[7]、果实成熟度[8]和农业自动化[9-10]等领域发挥着重要的作用。目前,基于深度学习的苹果检测算法依据检测的阶段划分主要有2大类:一类是要先用特征提取器生成一系列可能包含待检物体的预选框,然后利用算法对生成的候选区域进行更精细的检测和分类的两阶段算法,主要有Faster-RCNN[11]和Mask-RCNN[12]算法;另一类为直接预测物体类别和位置的一阶段检测算法,主要有RetinaNet[13]、SSD[14]和YOLO系列[15-17]。两阶段算法计算复杂度高、检测速度较慢,一阶段算法在精度上有所欠缺。两阶段算法由于包含多个阶段和较多的候选框处理,推理速度通常比单阶段算法慢,不适合实时应用。为了进一步提升模型的检测速度和准确度,Tian等[18]提出了一种名为VMF-SSD(基于V空间的多尺度特征融合SSD)的新型苹果叶片病害检测方法;Wang等[19]通过迁移学习构建YOLOv5s检测模型的同时,采用通道修剪算法对模型进行修剪及微调,以实现对苹果果实的快速准确检测。
目前,国内外目标检测领域对苹果的研究主要集中在自然环境下的苹果识别、苹果采摘机器人以及叶片病虫害识别方面。虽然已有部分研究针对缺陷苹果的检测[20-21],但仍存在上下文信息和多尺度特征融合不充分的问题。针对缺陷苹果识别,本文提出一种基于BiFPN[22]和Triplet注意力机制的YOLOv5s (BTF-YOLOv5s)缺陷苹果识别算法,通过BiFPN结合特征的上采样和下采样路径,联合跨尺度的特征交互,能够更好地传递和融合来自不同尺度的特征信息,提升目标检测性能;同时,学习动态的特征融合权重,根据不同目标的重要性进行灵活调整,增强模型的鲁棒性;在Neck层应用Triplet注意力机制,模型能更好地捕捉图像中的细节和上下文信息,提升目标检测的精度;采用Focal-CIoU调整正负样本之间的损失权重,让模型对难以分类的样本给予更多关注,提升模型的分类性能。
1. 基于BiFPN和Triplet注意力机制的YOLOv5s算法
本研究以YOLOv5s (版本6.2,包含4种不同大小的模型)为基础,网络结构由输入端、骨干网络、颈部网络和输出端4个部分组成。在输入端引入了Mosaic数据增强方法,随机选取4张图像,对它们进行随机放大、缩小等操作后,拼接成一张新的图像,使模型可以在更小的范围内识别目标;骨干网络主要使用了Focus和CSP1_X结构;颈部网络采用FPN+PAN和CSP2_X结构进一步进行特征融合和上采样操作,以提供更高级的语义信息和适应不同尺度图片的能力;输出端采用CIoU_Loss(Complete intersection over union loss)作为损失函数,并使用了非极大值抑制(Non-maximum suppression,NMS)进行后处理。本文提出了一种改进的YOLOv5s算法(BTF-YOLOv5s)用于缺陷苹果识别,首先,加权双向特征金字塔网络(Bidirectional feature pyramid network,BiFPN)替代YOLOv5中的FPN+PAN结构,通过引入可学习的权重学习不同输入特征的重要性;然后,将Triplet注意力机制应用到模型的最后一层,确保在最终输出前对特征进行充分的处理和优化;最后,采用Focal-CIoU损失函数调整损失权重给予缺陷苹果更多的关注,BTF-YOLOv5s算法结构整体框图如图1所示。
1.1 BiFPN
在目标检测任务中,有效地获取并处理不同尺度的特征信息是一个主要的挑战。传统的特征金字塔网络(FPN)[23]通过自上而下的方式聚合多尺度特征,如图2a所示,但容易受到单向信息流的限制;路径聚合网络(PANet)[24]在此基础上额外添加了一个自下向上的路径聚合网络,如图2b所示。在YOLOv5的Neck结构中,借鉴PANet的思想,高层的特征信息通过FPN+PAN结构进行传递融合。FPN+PAN结构虽然提高了特征传递的效率,但也增加了计算复杂度,尤其是在处理高分辨率输入时,可能导致较高的计算成本和较差的实时性;此外,固定结构导致对不同任务和数据缺乏自适应的能力。
为了解决上述信息流动单一、精度高但参数较多、计算量较大以及简单拼接导致的信息丢失和冗余等问题,本文提出了采用BiFPN替代YOLOv5s中的FPN+PAN结构。BiFPN在PANet和NAS-FPN(图2c)基础上优化了多尺度特征融合方式,结构如图2d所示。
传统方法在融合具有不同分辨率的特征时,平等地对待所有输入特征,并将它们简单相加。但不同分辨率的输入特征对输出特征的贡献通常是不等的。为了解决这一问题,BiFPN为每个输入特征增加了一个额外的权重,并让网络学习每个输入特征的重要性,具体见式(1)和式(2):
$$ P_6^{{\mathrm{td}}} = {\mathrm{Conv}}\left[ {\frac{{{w_1}P_6^{{\mathrm{in}}} + {w_2}{\mathrm{Reseize}}\left( {P_7^{{\mathrm{in}}}} \right)}}{{{w_1} + {w_1} + \varepsilon }}} \right], $$ (1) $$ P_6^{{\mathrm{out}}} = {\mathrm{Conv}}\left[ {\frac{{w_1^{'}P_6^{{\mathrm{in}}} + w_2^{'}P_6^{{\mathrm{td}}} + w_3^{'}{\mathrm{Reseize}}\left( {P_5^{{\mathrm{out}}}} \right)}}{{w_1^{'} + w_2^{'} + w_3^{'} + \varepsilon }}} \right], $$ (2) 式中,
$ P_6^{{\mathrm{td}}}$ 为自顶向下路径第6层的中间特征;$ P_6^{{\mathrm{in}}}$ 为第6层的输入特征; Resize用于分辨率匹配的上采样或下采样运算;$ P_6^{{\mathrm{out}}}$ 为自底向上路径第6层的输出特征。这一改进使得网络能够自适应地调整每个特征图的重要性,从而实现更有效的特征融合。引入BiFPN的YOLOv5s模型通过多尺度特征融合和简化的计算过程,有效地提升了对缺陷苹果检测的准确率;BiFPN通过引入可学习的融合权重优化了不同分辨率特征的重要性,增强了特征复用和多级特征融合路径,使得YOLOv5能够有效地检测出不同大小和形状的苹果缺陷。
1.2 Triplet注意力机制
在缺陷苹果检测任务中,模型需要准确地识别和定位图像中的目标(苹果)。图像中存在大量的背景信息以及不同大小的苹果会分散模型的注意力,使得模型难以准确地区分缺陷苹果和背景,引入注意力机制旨在提升模型对关键目标的感知能力。SENet通过简单的操作(全局平均池化和全连接层)自适应地调整特征图中每个通道的重要性,使网络更加集中地关注重要的特征信息,在低计算成本的条件下通过通道注意力机制显著提升网络的表示能力[25];CBAM将通道注意力和空间注意力相结合,能够同时关注重要的通道特征和空间位置;CBAM中的通道注意力方法虽然提供了一定的性能改进,但通道注意力和空间注意力是相互分离和计算的[26]。
Triplet 注意力机制(简称“Triplet”)以一种有效的方式解释了CBAM未考虑到的跨维度的相互作用[27]。如图3所示,Triplet由3个平行的分支构成,其中2个分支分别用来捕获通道
$C$ 维度和空间维度$ W/H $ 之间的跨通道交互,最后的一个分支用于捕获$ H $ 和$W$ 的空间依赖关系。在第1个分支中,输入张量C×H×W(通道数$C$ 、高度$H$ 和宽度$W$ )沿$H$ 轴逆时针旋转90°,形状变为$W$ ×$H$ ×$C$ ,输入特征先后经过$ Z - {\mathrm{Pool}} $ 、$ K \times K $ 的标准卷积层、批量归一化层、通过Sigmod激活函数生成空间注意力权重后、再沿H轴顺时针旋转90°保持与输入的形状一致。其中$ Z - {\mathrm{Pool}} $ 表示为:$$ Z - {\mathrm{Pool}}(\chi ) = \left[{\mathrm{Max}}{{\mathrm{Pool}}_{0{\mathrm{d}}}}(\chi ),{\mathrm{Avg}}{{\mathrm{Pool}}_{0{\mathrm{d}}}}(\chi )\right]{\text{,}} $$ (3) 式中,
$ 0{\mathrm{d}}$ 表示发生最大池化和平均池化操作的第0维度。第2、第3个分支同理,最后对3个分支输出特征进行平均后聚合在一起,最终输出的张量:
$$\begin{split} y =& \frac{1}{3}\Biggr\{ \overline {\mathop {{\chi _1}}\limits^ \wedge \sigma \left[ {{\psi _1}\left( {\mathop {\chi _1^*}\limits^ \wedge } \right)} \right]} + \overline {{{\mathop \chi \limits^ \wedge }_2}\sigma \left[ {{\psi _2}\left( {\mathop {\chi _2^*}\limits^ \wedge } \right)} \right]} + \Biggr.\\&\Biggr.\chi \sigma \left[ {{\psi _3}\left( {\mathop {{\chi _3}}\limits^ \wedge } \right)} \right] \Biggr\}{\text{,}} \end{split} $$ (4) 式中,
$ \sigma $ 代表Sigmod激活函数;$ \psi _1$ 、$ \psi _2$ 和$ \psi _3$ 表示由核大小$K$ 定义的标准二维卷积层。Triplet通过旋转操作构建各维度间的相关性,再对其进行残差变换,并以极小的计算成本对信息进行编码,在不需要太多可学习参数的前提下建立通道之间的相互依赖关系,有效地捕捉苹果图像中的关键特征。
1.3 Focal-CIoU损失函数
在机器学习任务中,常常面临数据集中不同类别的样本数量差异较大的问题,传统的交叉熵损失函数在处理样本不均衡问题时表现不佳,因为它将所有样本的重要性视为相等,继而导致模型在训练过程中更容易偏向于数量多的类别,公式如下:
$$ {\mathrm{CE}}(p,y) = {\mathrm{CE}}({P_t}) = - \ln ({P_t}) {\text{,}}$$ (5) 式中,CE是交叉熵(Cross entropy),
$p$ 代表模型预测样本属于类别1的概率(取值范围0~1),$y$ 表示样本的标签(取值为−1和1)。定义$$P_t=\left\{ \begin{array}{ll} {p}, &{{\text{当}} \; y=1{\text{时}}}\\ {1-p},& {{\text{其他}}} \end{array}\right. {\text{。}}$$ (6) 数据集中正常苹果和缺陷苹果图片数量均为1 600张左右,但标注数量略有差异,分别为4 624和2 039张。为了进一步加强模型对缺陷苹果的检测能力,引入Focal-CIoU损失函数。Focal Loss(FL)在交叉熵损失的基础上进行了改进,引入了平衡因子和聚焦因子,平衡因子用于调整正负样本之间的权重,聚焦因子用于调整难易样本间的损失权重;这样,既能调整正负样本的权重,又能控制难易分类样本的权重。FL表达式[28]为:
$$ {\mathrm{FL}}({P_t}) = - {\alpha _t}{(1 - {P_t})^\gamma }\ln ({P_t}), $$ (7) 式中,
${\alpha _t}$ 是超参数(取0.25);$ {(1 - {P_t})^\gamma } $ 是调节因子,$\gamma $ 是$ \geqslant $ 0的可调节聚焦参数(取2)。CIoU损失函数(LossCIoU)考虑边界框宽高比的尺度信息,在DIoU的基础上进行优化,计算公式[29]如下:
$$ {\mathrm{DI}}{\text{o}}{\mathrm{U}} = {\mathrm{IoU}} - \frac{{{d^2}}}{{{c^2}}}{\text{,}} $$ (8) $$ {\mathrm{Loss}}_{\mathrm{CIoU}} = {\mathrm{IoU}} - {\frac{{{\rho ^2}\left( {b,{b^{gt}}} \right)}}{{{c^2}}}^{}} - \alpha V{\text{,}} $$ (9) 式中,IoU为交并比(Intersection over Union),即“预测的边框”和“真实的边框”的交集和并集的比值;
$ d$ 为预测框与真实框中心点的长度,$ c$ 为最小外接矩形的对角线距离,$\rho $ 代表的是2个中心点之间的欧式距离,$ b、{b^{gt}} $ 分别代表了预测框和真实框的中心点,$\alpha $ 是权重函数,$V$ 用来衡量长宽比的相似度。Focal-CIoU损失函数(
${\mathrm{L}}{\text{os}}{{\text{s}}_{{\mathrm{Focal}} - {\mathrm{CIoU}}}} $ )为:$$ {\mathrm{L}}{\text{os}}{{\text{s}}_{{\mathrm{Focal}} - {\mathrm{CIoU}}}} = {\mathrm{Io}}{{\mathrm{U}}^\gamma } \times {\mathrm{Los}}{{\mathrm{s}}_{{\mathrm{CIoU}}}} {\text{。}}$$ (10) Focal-CIoU通过调整正负样本的权重和提升边界框回归精度,减少了训练过程中梯度的不稳定性,使模型更关注缺陷苹果,提高了检测性能。
2. 试验与结果分析
2.1 数据集
本研究所用数据集从百度网站和飞桨AI Studio网站上获取,这2个网站提供了丰富的图像资源,包含虫蛀、腐烂、机械损伤和褶皱共4类苹果缺陷图像(图4),数量分别为414、750、148和280张,以确保对于苹果目标的全面覆盖。数据集共
3213 张(正常苹果1621 张、缺陷苹果1592 张),训练集和验证集按照9∶1的比例划分,其中,训练集图片2 891张、验证集图片322张。为了增加数据的多样性,通过Python程序对部分数据集进行水平、垂直方向的翻转以及旋转45º操作。构建和处理数据集,确保模型在训练和验证过程中能够充分学习和适应苹果目标的多样性特征,为后续的目标检测提供数据保障。2.2 模型训练与评价指标
本试验平台基于Ubuntu 18.04.6的64位操作系统,显卡为GPU(NVIDIA GeForce RTX2080Ti),显存为12 G,使用PyTorch框架构建模型,编程语言为Python,Torch版本为1.10.1。在训练过程中,设置初始学习率为0.01、周期学习率为0.20、动量为0.937、权重衰减系数为0.000 5、批量大小为16。模型评估指标包括准确率(Precision,P)、召回率(Recall,R)、平均精确率均值(Mean average precision,mAP)和F1。TP (True positive)表示模型正确地检测出缺陷苹果的数量;TN (True negative)表示模型正确地排除正常苹果的数量;FP (False positive)表示模型错误地将正常苹果判定为缺陷苹果的数量; R指模型正确检测出的缺陷苹果(TP)占所有真正的缺陷苹果(TP + FN)的比例,表示模型对于真实缺陷苹果的检测能力;AP(Average precision)是衡量目标检测模型在不同召回率下准确性的指标,mAP是所有类别AP的平均,用来评估模型整体性能; F1综合了P和R这2个指标,能够更全面地评估模型的性能。
训练过程中损失值参数的动态变化如图5所示,随着训练轮次递增,损失值经历了急剧下降的阶段,最终趋向于0.025左右的平稳水平。训练损失和验证损失逐渐趋于拟合,意味着模型所学习的训练数据中的模式能够有效泛化到验证数据,表明该模型在面对新数据时具有优异的泛化性能。
2.3 Triplet的不同添加位置
Triplet插入YOLOv5s结构中的位置(A表示YOLOv5s-BiFPN-Triplet)主要有以下4种方式:1)在网络结构SPPF前面添加,即第9层[A+CIoU+Focal (9)];2)在网络结构最后一层添加,即第24层[A+ CIoU+Focal(24)];3)在网络结构SPPF前面和最后一层添加,即A+CIoU+Focal(9+25);4)替换Backbone中的C3模块,即A+CIoU+Focal。不同插入位置对比试验结果见表1。由表1可见,A+CIoU+Focal(24)比其他4种模型在各个方面均有不错的提升,准确率比YOLOv5s的提高了5.7个百分点,综合分析表明A+ CIoU+Focal(24)是最优的模型。
表 1 Triplet注意力机制不同插入位置的模型试验结果对比Table 1. Result comparison of Triplet attention mechanism with different insertion position% 模型1)Model P R mAP F1 YOLOv5s 75.4 83.6 86.5 79.29 A+ CIoU+Focal(9) 77.3 81 85.6 79.11 A+CIoU+Focal(9+25) 80.8 79.9 86.2 80.35 A+CIoU+Focal 75.9 85.2 87.1 80.28 A+CIoU+Focal(24) 81.1 85.8 90.0 83.38 1) A:YOLOv5s-BiFPN-Triplet,括号内数字表示Triplet插入位置。
1) The number in parentheses indicated the insertion position of Triplet.2.4 BiFPN添加不同注意力机制
以YOLOv5s-BiFPN为基础,逐步添加SE,CBAM、CA和Triplet注意力机制。SE仅考虑通道间的信息,忽略了位置信息;CBAM通过学习的方式自动获取每个特征通道和特征空间的重要程度;而CA不仅考虑通道间的关系,而且考虑方向相关的位置信息。试验结果见表2,由表2可见,BiFPN+CA在准确率和mAP方面优于BiFPN+SE和BiFPN+CBAM;Triplet注意力机制利用三分支结构能够捕获更多的关键信息,建立通道之间的相互依赖关系,试验结果也验证了BiFPN+Triplet是最优模型。
表 2 BiFPN添加不同注意力机制的模型试验结果对比Table 2. Result comparison of BiFPN with different attention mechanism% 模型Model P R mAP BiFPN+SE 76.1 83.1 87.0 BiFPN+CBAM 77.5 81.5 87.6 BiFPN+CA 78.6 81.3 88.1 BiFPN+Triplet 79.3 83.2 88.4 2.5 消融试验
为了验证各个模块的作用,以YOLOv5s为基础,进行逐步添加和替换,结果见表3。从表3中可以看出,与初始的YOLOv5s模型相比,单独添加BiFPN、Triplet或Focal-CIoU模块的模型准确率、召回率和mAP均有一定的提升。同时添加2种模块时,取得的效果更佳,添加BiFPN+Triplet的模型mPA比单独添加BiFPN的提高了0.4个百分点、准确率比单独添加Triplet的提高2.9个百分点;添加Triplet+Focal-CIoU的模型准确率和mAP比单独添加Triplet的分别提高1.1和0.3个百分点;添加BiFPN+Focal-CIoU的模型准确率和mAP比单独添加Focal-CIoU的分别提高1.7和1.0个百分点。当三者同时添加时,提高了模型对缺陷苹果的感知能力,与YOLOv5s模型相比,准确率、召回率和mAP分别提高5.7、2.2和3.5个百分点。
表 3 消融试验结果Table 3. The result of ablation test% BiFPN Triplet Focal-CIoU P R mAP 75.4 83.6 86.5 √ 80.3 84.3 88.0 √ 76.4 86.1 89.3 √ 78.4 83.0 89.8 √ √ 79.3 83.2 88.4 √ √ 77.5 84.5 89.6 √ √ 80.1 84.0 88.9 √ √ √ 81.1 85.8 90.0 为了验证Focal-CIoU损失函数的优越性,本文设计了YOLOv5s-BiFPN-Triplet+损失函数的消融试验,结果见表4。由表4可知,A+ CIoU+Focal与次优模型A+ SIoU相比,准确率提高了1个百分点、召回率和mAP提高0.2个百分点、F1提高0.62个百分点;虽然A+ WIoU的准确率与A+ CIoU+Focal相同,但召回率、mAP和F1均低于A+ CIoU+Focal。综合准确率、召回率、mAP和F1等指标, A+ CIoU+Focal是最优模型。
表 4 Focal-CIoU与其他损失函数对比Table 4. Comparison of Focal-CIoU with other loss functions% 模型1)Model P R mAP F1 A 79.3 83.2 88.4 81.20 A+ DIoU 79.1 84.8 89.2 81.85 A+ DIoU+Focal 79.4 84.3 89.6 81.78 A+ SIoU 80.1 85.6 89.8 82.76 A+ SIoU+Focal 77.9 85.3 88.5 81.43 A+ EIoU 78.7 84.3 88.7 81.40 A+ EIoU+Focal 80.7 80.0 88.1 80.35 A+ WIoU 81.1 79.4 88.2 80.24 A+CIoU+Focal 81.1 85.8 90.0 83.38 1) A:YOLOv5s-BiFPN-Triplet. 2.6 模型有效性验证
为了证明本文提出模型(BTF-YOLOv5s)的有效性,与当前主流的目标检测模型进行对比(表5)。由表5可见,与SSD相比,BTF-YOLOv5s的准确率提高了4.8个百分点、召回率和mAP分别提高了4.1和5.7个百分点,模型大小大幅度减小;与YOLOv3相比,BTF-YOLOv5s的准确率提高近10个百分点,模型小了近8倍;与YOLOv4相比,BTF-YOLOv5s虽然准确率低了3.1个百分点,但召回率和mAP分别提升近20和13个百分点,模型大小减少了17倍;与YOLOv5s、YOLOv7和YOLOv8s相比,BTF-YOLOv5s在准确率、召回率和mAP方面均有提升;YOLOv8n和YOLOv9的准确率相较于BTF-YOLOv5s各提高0.6和4.9个百分点,但二者召回率过低; F1显示BTF-YOLOv5s是最优的。综合分析,BTF-YOLOv5s显著优于大多数目标检测模型,更具有优势。图6直观地展示了SSD、YOLOv3、YOLOv5s等模型与BTF-YOLOv5s的mAP比较,在训练轮次逐步增加的过程中,BTF-YOLOv5s的mAP最终稳定在90%左右,明显优于其他模型。
表 5 BTF-YOLOv5与其他模型的对比Table 5. Comparison of BTF-YOLOv5 with other models模型
ModelP/% R/% mAP/% 模型大小/MB
Model sizeF1/% 参数
ParameterGFLOPs1) SSD 76.3 81.7 84.3 95.5 78.91 2.49×107 31.4 YOLOv3 71.8 83.8 86.5 123.5 77.34 3.30×107 78.1 YOLOv4 84.2 65.6 76.6 256.3 73.75 6.50×107 142.3 YOLOv5s 75.4 83.6 86.5 14.4 79.29 7.03×106 16.0 YOLOv7 79.6 83.1 87.1 142.1 81.31 3.72×107 105.1 YOLOv8n 81.7 76.6 87.4 6.2 79.07 3.01×106 8.2 YOLOv8s 73.6 83.0 87.2 22.5 78.02 1.11×107 28.6 YOLOv9 86.0 78.8 89.7 102.8 82.24 5.10×107 238.9 BTF-YOLOv5s 81.1 85.8 90.0 14.7 83.38 7.17×106 16.7 1) GFLOPs:每秒10亿次的浮点运算数。
1) GFLOPs: Giga floating-point operations per second.2.7 应用场景试验结果
本模型的应用场景之一即苹果采摘机器人在采摘过程中的分拣,故选择缺陷苹果所处的自然环境下的场景以及摆放不规整的场景。如图7所示,其中,第1行图像为原始图像、第2行图像为YOLOv5s检测图、第3行图像为BTF-YOLOv5s检测图;在图7的第1列中,BTF-YOLOv5s能够有效检测出正常苹果与缺陷苹果,准确率比YOLOv5s有显著提升;在第2列中对于像素更少的瑕疵斑点,YOLOv5s模型不仅出现了误检,而且准确率也低于BTF-YOLOv5s;在第3列中,BTF-YOLOv5s模型的检测效果明显优于YOLOv5s模型。
3. 结论
为了实现对瑕疵苹果的快速准确检测,本文提出基于BiFPN和Triplet注意力机制的YOLOv5s缺陷苹果识别算法。通过加权双向特征金字塔网络(BiFPN)来学习不同的输入特征;采用Triplet注意力机增强模型对目标之间的关联和上下文信息的表示能力;采用Focal-CIoU损失函数,在计算损失时调整损失权重,给予目标物体更多的关注。注意力机制的应用使模型更加关注目标,在4种位置的插入对比中,YOLOv5s网络结构最后一层的插入位置表现最为优越,与在网络结构SPPF前面和最后一层添加相比,准确率、召回率和mAP分别提高了0.3、5.9和3.8个百分点;BiFPN与SE、CBAM、CA、Triplet注意力机制两两组合,验证了BiFPN+Triplet的优越性;相较于BiFPN+CA,准确率、召回率和mAP分别提升了0.7、1.9和0.3个百分点。消融试验表明,以YOLOv5s为基础,同时添加3种模块的准确率、召回率和mAP分别提高了5.7、2.2和3.5个百分点,提升效果显著。以YOLOv5s-BiFPN-Triplet为基础,Focal-CIoU的准确率比次优损失函数SIoU高1个百分点;准确率、召回率和mAP比主流的YOLOv7算法提高了1.5、2.7和2.9个百分点,同时,模型大小从142.1 MB减小到14.7 MB,显著降低了内存占用,在计算资源受限的环境中,为部署目标检测系统提供了有力支持。YOLOv8n和YOLOv9的准确率虽略高于BTF-YOLOv5s,但均面临召回率过低的问题,而YOLOv8s的准确率只有73.6%。在应用场景的对比试验中,BTF-YOLOv5s也表现出较好的优越性。
-
[1] ZUO J, LI J. Molecular dissection of complex agronomic traits of rice: A team effort by Chinese scientists in recent years[J]. Natl Sci Rev, 2014, 1(2): 253-276. doi: 10.1093/nsr/nwt004
[2] 袁隆平. 种业竞争时代的科技创新:超级杂交水稻育种研究新进展[J]. 中国农村科技, 2010(2): 22-25. doi: 10.3969/j.issn.1005-9768.2010.02.006 [3] FITZGERALD M A, MCCOUCH S R, HALL R D. Not just a grain of rice: The quest for quality[J]. Trends Plant Sci, 2009, 14(3): 133-139. doi: 10.1016/j.tplants.2008.12.004
[4] 周开达, 李宏伟. 优质是杂交稻发展的必由之路[J]. 杂交水稻, 1994(z1): 42-45. [5] 肖国樱, 陈芬, 孟秋成, 等. 论湖南水稻育种的主攻方向和技术策略[J]. 杂交水稻, 2015, 30(4): 1-5. [6] HRBERD N P. Shaping taste: The molecular discovery of rice genes improving grain size, shape and quality[J]. J Genet Genomics, 2015, 42(11): 597-599. doi: 10.1016/j.jgg.2015.09.008
[7] XING Y, ZHANG Q. Genetic and molecular bases of rice yield[J]. Annu Rev Plant Biol, 2010, 61(1): 421-442. doi: 10.1146/annurev-arplant-042809-112209
[8] SONG X J, ASHIKARI M. Toward an optimum return from crop plants[J]. Rice, 2008, 1(2): 135-143. doi: 10.1007/s12284-008-9018-3
[9] 杨联松, 白一松, 张培江, 等. 谷粒形状与稻米品质相关性研究[J]. 杂交水稻, 2001(4): 51-53. doi: 10.3969/j.issn.1005-3956.2001.04.024 [10] HUANG R, JIANG L, ZHENG J, et al. Genetic bases of rice grain shape: So many genes, so little known[J]. Trends Plant Sci, 2013, 18(4): 218-226. doi: 10.1016/j.tplants.2012.11.001
[11] 黎毛毛, 徐磊, 刘昌文, 等. 水稻粒形遗传及QTLs定位研究进展[J]. 中国农业科技导报, 2008, 10(1): 34-42. doi: 10.3969/j.issn.1008-0864.2008.01.007 [12] CALINGACION M, LABORTE A, NELSON A, et al. Diversity of global rice markets and the science required for consumer-targeted rice breeding[J]. PLoS One, 2014, 9(1): e85106. doi: 10.1371/journal.pone.0085106.
[13] ZHAO D S, LI Q F, ZHANG C Q, et al. GS9 acts as a transcriptional activator to regulate rice grain shape and appearance quality[J]. Nat Commun, 2018, 9(1). doi: 10.1038/s41467-018-03616-y.
[14] 莫惠栋. 种子性状及其遗传效应的鉴别[J]. 扬州大学学报(农业与生命科学版), 1990, 11(2): 11-15. [15] 林鸿宣, 闵绍楷, 熊振民, 等. 应用RFLP图谱定位分析籼稻粒形数量性状基因座位[J]. 中国农业科学, 1995, 28(4): 1-7. doi: 10.3321/j.issn:0578-1752.1995.04.008 [16] 周清元, 安华, 张毅, 等. 水稻子粒形态性状遗传研究[J]. 西南农业大学学报, 2000(2): 102-104. doi: 10.3969/j.issn.1673-9868.2000.02.002 [17] 谭友斌. 利用高世代回交群体分析水稻粒型QTLs[D]. 武汉: 华中农业大学, 2006. [18] TAN Y F, LI J Y S, XU C G, et al. Genetic bases of appearance quality of rice grains in Shanyou 63, an elite rice hybrid[J]. Theor Appl Genet, 2000, 101(6): 823-829.
[19] 苪重庆, 赵安常. 籼稻粒重及粒形性状F1遗传特性的双列分析[J]. 中国农业科学, 1983, 16(5): 14-20. [20] 石春海, 申宗坦. 籼稻粒形及产量性状的加性相关和显性相关分析[J]. 作物学报, 1996, 22(1): 36-42. doi: 10.3321/j.issn:0496-3490.1996.01.006 [21] 姚国新, 卢磊. 水稻粒重基因定位克隆研究[J]. 安徽农业科学, 2007, 35(27): 8468. doi: 10.3969/j.issn.0517-6611.2007.27.023 [22] 莫惠栋. 我国稻米品质的改良[J]. 扬州大学学报(农业与生命科学版), 1994, 15(1): 8-14. [23] 李欣, 莫惠栋, 王安民, 等. 粳型杂种稻米品质性状的遗传表达[J]. 中国水稻科学, 1999, 13(4): 197-204. [24] 万向元, 陈亮明, 王海莲, 等. 水稻品种胚乳淀粉RVA谱的稳定性分析[J]. 作物学报, 2004(12): 1185-1191. doi: 10.3321/j.issn:0496-3490.2004.12.002 [25] 熊振民, 孔繁林. 大粒型水稻品种的遗传动态及其选育[J]. 浙江农业科学, 1976(2): 26-29. [26] MCKENZIE K S, RUTGER J N. Genetic analysis of amylose content, alkali spreading score, and grain dimensions in rice[J]. Crop Sci, 1983, 23(2): 306-313. doi: 10.2135/cropsci1983.0011183X002300020031x
[27] JUN C L, NUTIK S L. Surgical approaches to intraventricular meningiomas of the trigone[J]. Neurosurgery, 1985, 16(3): 416-420. doi: 10.1227/00006123-198503000-00025
[28] 林荔辉, 吴为人. 水稻粒型和粒重的QTL定位分析[J]. 分子植物育种, 2003(3): 337-342. doi: 10.3969/j.issn.1672-416X.2003.03.007 [29] 石春海, 申宗坦. 早籼粒形的遗传和改良[J]. 中国水稻科学, 1995(1): 27-32. doi: 10.3321/j.issn:1001-7216.1995.01.006 [30] 杨联松, 白一松, 陈多璞, 等. 粳稻粒形遗传初步研究[J]. 杂交水稻, 2002, 17(6): 46-48. doi: 10.3969/j.issn.1005-3956.2002.06.021 [31] 邱先进, 袁志华, 何文静, 等. 水稻垩白性状遗传育种研究进展[J]. 植物遗传资源学报, 2014(5): 992-998. [32] 彭波, 孙艳芳, 李琪瑞, 等. 水稻垩白性状的遗传研究进展[J]. 信阳师范学院学报(自然科学版), 2016, 29(2): 304-312. doi: 10.3969/j.issn.1003-0972.2016.02.035 [33] 国家市场监督管理总局中国国家标准化管理委员会. 大米: GB/T—2018[S]. 北京: 中国标准出版社, 2018:1-10. [34] 徐正进, 陈温福, 马殿荣, 等. 稻谷粒形与稻米主要品质性状的关系[J]. 作物学报, 2004(9): 894-900. doi: 10.3321/j.issn:0496-3490.2004.09.009 [35] 李泽福, 夏加发, 刘礼明, 等. 籼粳交重组自交系群体主要稻米品质性状分析[J]. 安徽农业科学, 2004, 32(6): 1112-1115. doi: 10.3969/j.issn.0517-6611.2004.06.003 [36] 石春海, 朱军. 水稻植株农艺性状与稻米碾磨品质的遗传相关性分析[J]. 浙江农业大学学报, 1997(3): 105-111. [37] 杨联松, 白一松, 许传万, 等. 水稻粒形与稻米品质间相关性研究进展[J]. 安徽农业科学, 2001(3): 312-316. doi: 10.3969/j.issn.0517-6611.2001.03.018 [38] LI Y, FAN C, XING Y, et al. Chalk5 encodes a vacuolar H+-translocating pyrophosphatase influencing grain chalkiness in rice[J]. Nat Genet, 2014, 46(4): 398-404. doi: 10.1038/ng.2923
[39] 李仕贵, 黎汉云, 周开达, 等. 杂交水稻稻米外观品质性状的遗传相关分析[J]. 西南农业大学学报, 1995, 17(3): 197-201. [40] 石春海. 水稻粒形与优质米育种[J]. 中国农学通报, 1994(1): 41-45. [41] ZUO J, LI J. Molecular genetic dissection of quantitative trait loci regulating rice grain size[J]. Annu Rev Genet, 2014, 48: 99-118. doi: 10.1146/annurev-genet-120213-092138
[42] ZHAO H, SUN L, XIONG T, et al. Genetic characterization of the chromosome single-segment substitution lines of O. glumaepatula and O. barthii and identification of QTLs for yield-related traits[J]. Mol Breeding, 2019, 39(4). doi: 10.1007/s11032-019-0960-0.
[43] BHATIA D, WING R A, YU Y, et al. Genotyping by sequencing of rice interspecific backcross inbred lines identifies QTLs for grain weight and grain length[J]. Euphytica, 2018, 214(2). doi: 10.1007/s10681-018-2119-1.
[44] HE N, WU R, PAN X, et al. Development and trait evaluation of chromosome single-segment substitution lines of O. meridionalis in the background of O. sativa[J]. Euphytica, 2017, 213(12). doi: 10.1007/s10681-017-2072-4.
[45] QI L, SUN Y, LI J, et al. Identify QTLs for grain size and weight in common wild rice using chromosome segment substitution lines across six environments[J]. Breeding Sci, 2017, 67(5): 472-482. doi: 10.1270/jsbbs.16082
[46] GAO F, ZENG L, QIU L, et al. QTL mapping of grain appearance quality traits and grain weight using a recombinant inbred population in rice (Oryza sativa L.)[J]. J Integr Agr, 2016, 15(8): 1693-1702. doi: 10.1016/S2095-3119(15)61259-X
[47] LIU D, KANG M, WANG F, et al. Mapping of the genetic determinant for grain size in rice using a recombinant inbred line (RIL) population generated from two elite indica parents[J]. Euphytica, 2015, 206(1): 159-173. doi: 10.1007/s10681-015-1493-1
[48] 刘丹, 王嘉宇, 柴永山, 等. 利用籼粳交RILs群体的水稻粒形QTLs定位[J]. 沈阳农业大学学报, 2015, 46(4): 385-390. doi: 10.3969/j.issn.1008-9713.2015.04.001 [49] 孙滨, 占小登, 林泽川, 等. 水稻粒形和粒重性状的相关性分析及QTL定位[J]. 分子植物育种, 2015, 13(12): 2663-2672. [50] YAN B, LIU R, LI Y, et al. QTL analysis on rice grain appearance quality, as exemplifying the typical events of transgenic or backcrossing breeding[J]. Breeding Sci, 2014, 64(3): 231-239. doi: 10.1270/jsbbs.64.231
[51] ZHANG H, WANG H, QIAN Y, et al. Simultaneous improvement and genetic dissection of grain yield and its related traits in a backbone parent of hybrid rice (Oryza sativa L.) using selective introgression[J]. Mol Breeding, 2013, 31(1): 181-194. doi: 10.1007/s11032-012-9782-z
[52] SINGH R, SINGH A K, SHARMA T R, et al. Fine mapping of grain length QTLs on chromosomes 1 and 7 in Basmati rice (Oryza sativa L.)[J]. J Plant Biochem Biot, 2012, 21(2): 157-166. doi: 10.1007/s13562-011-0080-3
[53] 梅德勇, 朱玉君, 樊叶杨. 籼稻稻米碾磨与外观品质性状的QTL定位[J]. 遗传, 2012(12): 1591-1598. [54] LIANG Y, ZHAN X, GAO Z, et al. Mapping of QTLs associated with important agronomic traits using three populations derived from a super hybrid rice Xieyou9308[J]. Euphytica, 2012, 184(1): 1-13. doi: 10.1007/s10681-011-0456-4
[55] MARATHI B. QTL analysis of novel genomic regions associated with yield and yield related traits in new plant type based recombinant inbred lines of rice (Oryza sativa L.)[J]. BMC Plant Biol, 2012, 12(1): 137. doi: 10.1186/1471-2229-12-137
[56] 张强, 姚国新, 胡广隆, 等. 利用极端材料定位水稻粒形性状数量基因位点[J]. 作物学报, 2011, 37(5): 784-792. [57] 姚国新, 李金杰, 张强, 等. 利用4个姊妹近等基因系群体定位水稻粒重和粒形QTL[J]. 作物学报, 2010, 36(8): 1310-1317. [58] AMARAWATHI Y, SINGH R, SINGH A K, et al. Mapping of quantitative trait loci for basmati quality traits in rice (Oryza sativa L.)[J]. Mol Breeding, 2008, 21(1): 49-65.
[59] 陈冰嬬, 石英尧, 崔金腾, 等. 利用BC2F2高代回交群体定位水稻籽粒大小和形状QTL[J]. 作物学报, 2008(8): 1299-1307. [60] 赵明富, 黄招德, 吴春珠, 等. 水稻谷粒粒长显性主效QTL的遗传分析与定位[J]. 分子植物育种, 2008(6): 1057-1060. doi: 10.3969/j.issn.1672-416X.2008.06.005 [61] YOON D B, KANG K H, KIM H J, et al. Mapping quantitative trait loci for yield components and morphological traits in an advanced backcross population between Oryza grandiglumis and the O. sativa japonica cultivar Hwaseongbyeo[J]. Theor Appl Genet, 2006, 112(6): 1052-1062. doi: 10.1007/s00122-006-0207-4
[62] ZHOU L Q, WANG Y P, LI S G. Genetic analysis and physical mapping of Lk-4(t), a major gene controlling grain length in rice, with a BC2F2 population[J]. Acta Genetica Sinica, 2006, 33(1): 72-79. doi: 10.1016/S0379-4172(06)60011-5
[63] WAN X Y, WAN J M, WENG J F, et al. Stability of QTLs for rice grain dimension and endosperm chalkiness characteristics across eight environments[J]. Theor Appl Genet, 2005, 110(7): 1334-1346. doi: 10.1007/s00122-005-1976-x
[64] MARRI P R, SARLA N, REDDY L V, et al. Identification and mapping of yield and yield related QTLs from an Indian accession of Oryza rufipogon[J]. BMC Genet, 2005, 6. doi: 10.1186/1471-2156-6-33.
[65] ALUKO G, MARTINEZ C, TOHME J, et al. QTL mapping of grain quality traits from the interspecific cross Oryza sativa × O. glaberrima[J]. Theor Appl Genet, 2004, 109(3): 630-639. doi: 10.1007/s00122-004-1668-y
[66] RABIEI B, VALIZADEH M, GHAREYAZIE B, et al. Identification of QTLs for rice grain size and shape of Iranian cultivars using SSR markers[J]. Euphytica, 2004, 137(3): 325-332. doi: 10.1023/B:EUPH.0000040452.76276.76
[67] LI Z F, WAN J M, XIA J F, et al. Mapping quantitative trait loci underlying appearance quality of rice grains (Oryza sativa L.)[J]. Acta Genetica Sinica, 2003, 30(3): 251-259.
[68] YAN C J, LIANG G H, FENG C, et al. Mapping quantitative trait loci associated with rice grain shape based on an indica/japonica backcross population[J]. Acta Genetica Sinica, 2003, 30(8): 711-716.
[69] 汪斌, 兰涛, 吴为人. 应用微卫星图谱定位稻米性状的QTL[J]. 福建农业学报, 2003(1): 11-15. doi: 10.3969/j.issn.1008-0384.2003.01.003 [70] YOSHIDA S, IKEGAMI M, KUZE J, et al. QTL analysis for plant and grain characters of sake-brewing rice using a doubled haploid population[J]. Breeding Sci, 2002, 52(4): 309-317. doi: 10.1270/jsbbs.52.309
[71] 吴长明, 孙传清, 陈亮, 等. 应用RFLP图谱定位分析稻米粒形的QTL[J]. 吉林农业科学, 2002, 27(5): 3-7. doi: 10.3969/j.issn.1003-8701.2002.05.001 [72] 徐建龙, 薛庆中, 罗利军, 等. 水稻粒重及其相关性状的遗传解析[J]. 中国水稻科学, 2002(1): 7-11. [73] BRONDANI C, RANGEL P, BRONDANI R, et al. QTL mapping and introgression of yield-related traits from Oryza glumaepatula to cultivated rice (Oryza sativa L.) using microsatellite markers[J]. Theor Appl Genet, 2002, 104(6): 1192-1203. doi: 10.1007/s00122-002-0869-5
[74] 邢永忠, 谈移芳, 徐才国, 等. 利用水稻重组自交系群体定位谷粒外观性状的数量性状基因[J]. 植物学报, 2001, 43(8): 840-845. doi: 10.3321/j.issn:1672-9072.2001.08.012 [75] MONCADA P, MARTÍNEZ C P, BORRERO J, et al. Quantitative trait loci for yield and yield components in an Oryza sativa×Oryza rufipogon BC2F2 population evaluated in an upland environment[J]. Theor Appl Genet, 2001, 102(1): 41-52. doi: 10.1007/s001220051616
[76] LI J X, YU S B, XU C G, et al. Analyzing quantitative trait loci for yield using a vegetatively replicated F2 population from a cross between the parents of an elite rice hybrid[J]. Theor Appl Genet, 2000, 101(1/2): 248-254.
[77] REDOÑA E D, MACKILL D J. Quantitative trait locus analysis for rice panicle and grain characteristics[J]. Theor Appl Genet, 1998, 96(6/7): 957-963.
[78] HUANG N, PARCO A, MEW T, et al. RFLP mapping of isozymes, RAPD and QTLs for grain shape, brown planthopper resistance in a doubled haploid rice population[J]. Mol Breeding, 1997, 3(2): 105-113. doi: 10.1023/A:1009683603862
[79] LI Z K, PINSON S R M, PARK W D, et al. Epistasis for three grain yield components in rice (Oryza Sativa L.)[J]. Genetics, 1997, 145(2): 453-465.
[80] 尉鑫, 曾智锋, 杨维丰, 等. 水稻粒形遗传调控研究进展[J]. 安徽农业科学, 2019, 47(5): 21-28. doi: 10.3969/j.issn.0517-6611.2019.05.006 [81] SONG X, HUANG W, SHI M, et al. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J]. Nat Genet, 2007, 39(5): 623-630. doi: 10.1038/ng2014
[82] CHE R, TONG H, SHI B, et al. Control of grain size and rice yield by GL2-mediated brassinosteroid responses[J]. Nat Plants, 2015, 2(1). doi: 10.1038/NPLANTS.2015.195.
[83] DUAN P, NI S, WANG J, et al. Regulation of OsGRF4 by OsmiR396 controls grain size and yield in rice[J]. Nat Plants, 2016, 2(1). doi: 10.1038/NPLANTS.2015.203.
[84] FAN C, XING Y, MAO H, et al. GS3, a major QTL for grain length and weight and minor QTL for grain width and thickness in rice, encodes a putative transmembrane protein[J]. Theor Appl Genet, 2006, 112(6): 1164-1171. doi: 10.1007/s00122-006-0218-1
[85] QI P, LIN Y S, SONG X J, et al. The novel quantitative trait locus GL3.1 controls rice grain size and yield by regulating Cyclin-T1;3[J]. Cell Res, 2012, 22(12): 1666-1680. doi: 10.1038/cr.2012.151
[86] ZHANG X, WANG J, HUANG J, et al. Rare allele of OsPPKL1 associated with grain length causes extra-large grain and a significant yield increase in rice[J]. Proc Natl Acad Sci USA, 2012, 109(52): 21534-21539. doi: 10.1073/pnas.1219776110
[87] LIU Q, HAN R, WU K, et al. G-protein βγ subunits determine grain size through interaction with MADS-domain transcription factors in rice[J]. Nat Commun, 2018, 9(1). doi: 10.1038/s41467-018-03047-9.
[88] SHOMURA A, IZAWA T K, EBITANI T, et al. Deletion in a gene associated with grain size increased yields during rice domestication[J]. Nat Genet, 2008, 40(8): 1023-1028. doi: 10.1038/ng.169
[89] LIU J, CHEN J, ZHENG X, et al. GW5 acts in the brassinosteroid signalling pathway to regulate grain width and weight in rice[J]. Nat Plants, 2017, 3(5). doi: 10.1038/nplants.2017.43.
[90] LI Y, FAN C, XING Y, et al. Natural variation in GS5 plays an important role in regulating grain size and yield in rice[J]. Nat Genet, 2011, 43(12): 1266-1269. doi: 10.1038/ng.977
[91] SUN L, LI X, FU Y, et al. GS6, a member of the GRAS gene family, negatively regulates grain size in rice[J]. J Integr Plant Biol, 2013, 55(10): 938-949.
[92] ISHIMARU K, HIROTSU N, MADOKA Y, et al. Loss of function of the IAA-glucose hydrolase gene TGW6 enhances rice grain weight and increases yield[J]. Nat Genet, 2013, 45(6): 707-713. doi: 10.1038/ng.2612
[93] SONG X J, KUROHA T, AYANO M, et al. Rare allele of a previously unidentified histone H4 acetyltransferase enhances grain weight, yield, and plant biomass in rice[J]. Proc Natl Acad Sci USA, 2015, 112(1): 76-81. doi: 10.1073/pnas.1421127112
[94] WANG Y, XIONG G, HU J, et al. Copy number variation at the GL7 locus contributes to grain size diversity in rice[J]. Nat Genet, 2015, 47(8): 944-948. doi: 10.1038/ng.3346
[95] WANG S, LI S, LIU Q, et al. The OsSPL16-GW7 regulatory module determines grain shape and simultaneously improves rice yield and grain quality[J]. Nat Genet, 2015, 47(8): 949-954. doi: 10.1038/ng.3352
[96] SI L, CHEN J, HUANG X, et al. OsSPL13 controls grain size in cultivated rice[J]. Nat Genet, 2016, 48(4): 447-456. doi: 10.1038/ng.3518
[97] WANG S, WU K, YUAM Q, et al. Control of grain size, shape and quality by OsSPL16 in rice[J]. Nat Genet, 2012, 44(8): 950-954. doi: 10.1038/ng.2327
[98] LI N, XU R, LI Y. Molecular networks of seed size control in plants[J]. Annu Rev Plant Biol, 2019, 70: 435-463. doi: 10.1146/annurev-arplant-050718-095851
[99] JIAO Y, WANG Y D, WANG J, et al. Regulation of OsSPL14 by OsmiR156 defines ideal plant architecture in rice[J]. Nat Genet, 2010, 42(6): 541-544. doi: 10.1038/ng.591
[100] MATSUOKA M, ASHIKARI M. A quantitative trait locus regulating rice grain width[J]. Nat Genet, 2007, 39(5): 583-584. doi: 10.1038/ng0507-583
[101] SHI C, REN Y, LIU L, et al. Ubiquitin specific protease 15 has an important role in regulating grain width and size in rice[J]. Plant Physiol, 2019, 180(1): 381-391. doi: 10.1104/pp.19.00065
[102] WENG J, GU S, WAN X, et al. Isolation and initial characterization of GW5, a major QTL associated with rice grain width and weight[J]. Cell Res, 2008, 18(12): 1199-1209. doi: 10.1038/cr.2008.307
[103] SUN S, WANG L, MAO H, et al. A G-protein pathway determines grain size in rice[J]. Nat Commun, 2018, 9. doi: 10.1038/s41467-018-03141-y.
[104] ASHIKARI M, SAKAKIBARA H, Lin S Y, et al. Cytokinin oxidase regulates rice grain production[J]. Science, 2005, 309(5735): 741-745. doi: 10.1126/science.1113373
[105] XU C, LIU Y, LI Y, et al. Differential expression of GS5 regulates grain size in rice[J]. J Exp Bot, 2015, 66(9): 2611-2623. doi: 10.1093/jxb/erv058
[106] SAKAMOTO T, MATSUOKA M. Identifying and exploiting grain yield genes in rice[J]. Curr Opin Plant Biol, 2008, 11(2): 209-214. doi: 10.1016/j.pbi.2008.01.009
[107] MIURA K, MATSUOKA M. Rice genetics: Control of grain length and quality[J]. Nat Plants, 2015, 1(8). doi: 10.1038/NPLANTS.2015.112.
[108] 万建民. 作物分子设计育种[J]. 作物学报, 2006, 32(3): 455-462. doi: 10.3321/j.issn:0496-3490.2006.03.023 [109] QIAN Q, GUO L, SMITH S M, et al. Breeding high-yield superior quality hybrid super rice by rational design[J]. Natl Sci Rev, 2016, 3(3): 283-294. doi: 10.1093/nsr/nww006
[110] HU J, WANG Y, FANG Y, et al. A rare allele of GS2 enhances grain size and grain yield in rice[J]. Mol Plant, 2015, 8(10): 1455-1465. doi: 10.1016/j.molp.2015.07.002
[111] GONG J, MIAO J, ZHAO Y, et al. Dissecting the genetic basis of grain shape and chalkiness traits in hybrid rice using multiple collaborative populations[J]. Mol Plant, 2017, 10(10): 1353-1356. doi: 10.1016/j.molp.2017.07.014
[112] GAO X, ZhANG X, LAN H, et al. The additive effects of GS3 and qGL3 on rice grain length regulation revealed by genetic and transcriptome comparisons[J]. BMC Plant Biol, 2015, 15. doi: 10.1186/s12870-015-0515-4.
[113] ZENG D, TIAN Z, RAO Y, et al. Rational design of high-yield and superior-quality rice[J]. Nat Plants, 2017, 3(4). doi: 10.1038/nplants.2017.31.
[114] 黄海祥, 钱前. 水稻粒形遗传与长粒型优质粳稻育种进展[J]. 中国水稻科学, 2017, 31(6): 665-672. [115] 杨梯丰, 曾瑞珍, 朱海涛, 等. 水稻粒长基因GS3在聚合育种中的效应[J]. 分子植物育种, 2010, 8(1): 59-66. [116] DAI Z, LU Q, LUAN X, et al. Development of a platform for breeding by design of CMS lines based on an SSSL library in rice (Oryza sativa L.)[J]. Euphytica, 2015, 205(1): 63-72. doi: 10.1007/s10681-015-1384-5




 下载:
下载: