Construction of a gE/gI-deleted mutant strain of epidemic porcine pseudorabies virus and its biological characteristics
-
摘要:目的
为研制针对猪伪狂犬病病毒流行毒株的疫苗提供候选毒株。
方法构建针对猪伪狂犬病病毒流行毒株的gE/gI缺失重组转移质粒pMD-LA-RA及携带EGFP标记基因的重组转移质粒pMD-LA-EGFP-RA,将pMD-LA-EGFP-RA与伪狂犬病病毒流行毒株PRV AH进行同源重组,利用EGFP为筛选标记,获得携带EGFP基因的gE/gI基因缺失突变株PRV AH gE –/gI –/EGFP+,以此毒株与pMD-LA-RA进行第2次同源重组,筛选去除EGFP基因的gE/gI基因缺失突变株PRV AH gE –/gI –,并通过生长曲线、易感细胞连续传代和动物免疫评价其增殖能力、遗传稳定性及免疫原性。
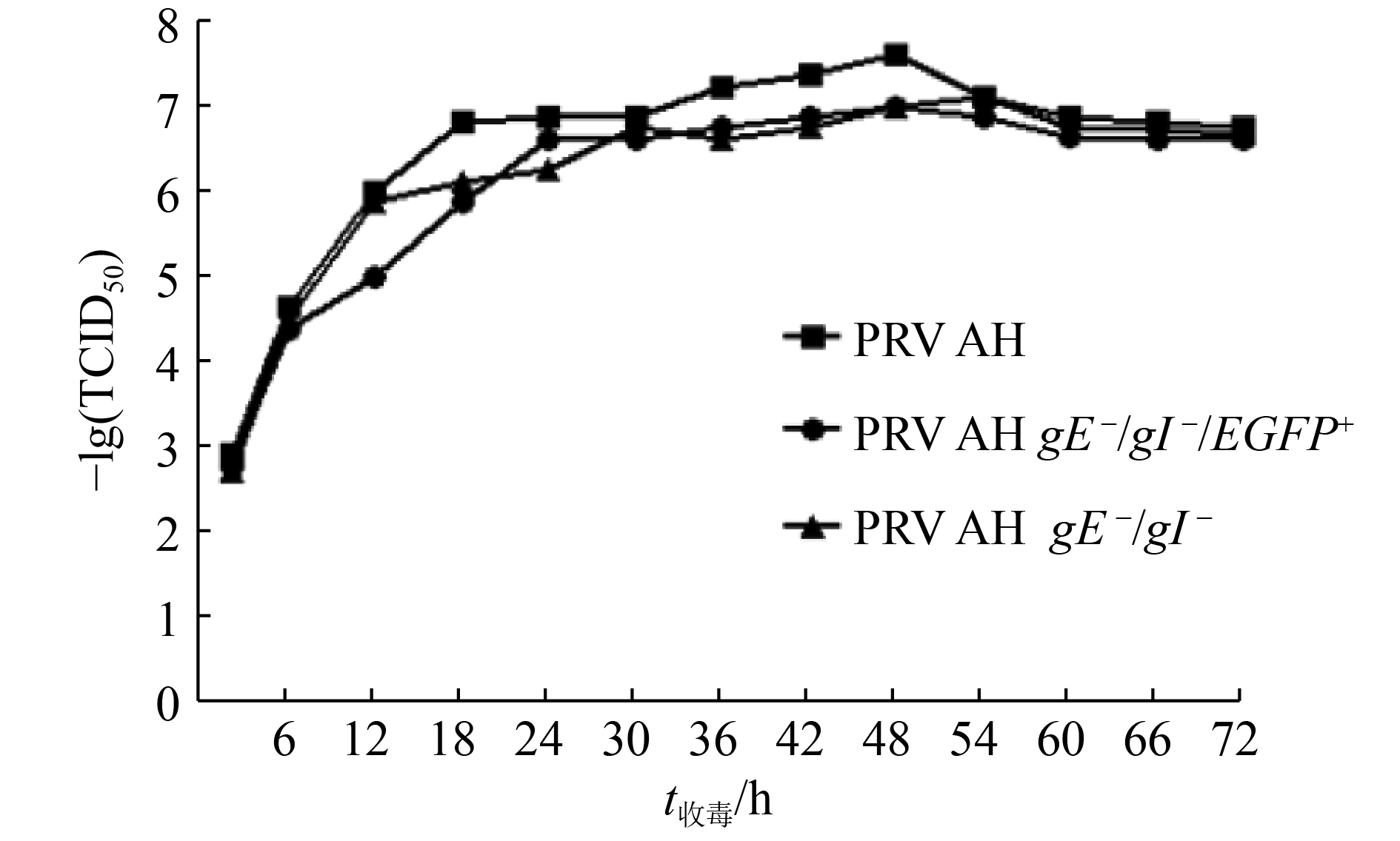
结果通过2次同源重组,结合荧光观察、空斑纯化和PCR检测,成功获得了PRV AH gE –/gI –,经PCR鉴定、荧光观察及测序鉴定,证实该毒株gE和gI基因被成功缺失,且不携带EGFP标记基因。生物学特性研究结果表明,该毒株增殖能力与亲本毒株相当,遗传稳定性及免疫原性良好。
结论采用同源重组技术成功构建了免疫原性良好的猪伪狂犬病病毒流行毒株gE/gI基因缺失突变株,为研制针对流行毒株的基因缺失疫苗奠定了一定的基础。
Abstract:ObjectiveTo obtain a candidate vaccine strain against epidemic porcine pseudorabies virus.
MethodA gE/gI-deleted transferring plasmid pMD-LA-RA and a recombinant plasmid carrying EGFP gene were constructed according to the sequence of epidemic porcine pseudorabies virus. The homologous recombination was operated between pMD-LA-EGFP-RA and PRV AH, and then the recombinant mutant virus PRV AH gE –/gI –/EGFP+ was selected using EGFP as screening marker. In order to obtain the gE/gI-deleted mutant strain PRV AH gE –/gI – without the EGFP gene, the second homologous recombination was carried out between pMD-LA-RA and PRV AH gE –/gI –/EGFP+. The proliferation ability, genetic stability and immunogenicity of PRV AH gE –/gI – were evaluated by its growth curve, continuous passage in susceptible cells and animal immunization.
ResultPRV AH gE –/gI – was obtained through two homologous recombinations with fluorescence observation, plaque purification and PCR. The deletion of gE, gI genes and EGFP gene marker in PRV AH gE –/gI – was identified by PCR, fluorescence observation and sequencing. PRV AH gE –/gI – had similar proliferation ability to the parental strain and had good genetic stability and immunogenicity.
ConclusionA gE/gI-deleted mutant strain of epidemic porcine pseudorabies virus with good immunogenicity was constructed successfully, which provides a basis for the development of gene deleted vaccine targeting epidemic strains.
-
植物固着生长在不断变化的环境中,不能通过移动来规避周围不良环境,需要根据环境灵活地调整自身生长状况进行应对。植物通过多种复杂的机制感知各种环境因素,并做出相应的反应,已经确定多种转录因子、转运蛋白等分子成分参与植物的胁迫响应[1-2]。在响应环境变化时,植物可以通过染色质结构的变化(包括DNA甲基化、组蛋白修饰、组蛋白变体、染色质重塑、非编码RNA等)来调控不同的生物学进程,从而适应外界环境的变化[3-4]。
H3K27me3是一种重要的组蛋白修饰,是一种抑制性的表观遗传标记,由Polycomb repressive complex 2(PRC2)复合物催化建立[5]。PRC2属于多梳家族蛋白(Polycomb group, PcG),PcG在表观遗传的调控方面起重要的作用,其最先在果蝇中被发现,参与许多重要的细胞活动和发育过程,具体包括干细胞多能性和可塑性、基因组印迹、X染色体失活等多种表观遗传过程[6-8]。在果蝇中PRC2的核心组分是一个含有SET结构域的甲基转移酶E[z](Enhancer of zeste),具有催化H3K27me3建立的活性[5]。
PRC2在果蝇、哺乳动物和植物中高度保守[9],拟南芥中存在和果蝇PRC2复合物核心亚基高度相似的蛋白,其中CURLY LEAF(CLF)、MEDEA (MEA)、SWINGER(SWN)是E[z]的同源蛋白[10-12]。在拟南芥生长发育过程中PRC2对调控基因的表达至关重要,涉及从种子萌发到胚形成的整个生命周期[13]。CLF和SWN主要在营养期起作用,而MEA主要在生殖期起作用[9, 13]。
拟南芥中CLF蛋白是PRC2蛋白复合物的重要组分,是H3K27me3甲基转移酶,CLF的突变会使H3K27me3的水平降低[14]。CLF影响植物叶片、花序、种子和根的发育,具体表现为:clf突变体叶片窄小而卷曲,叶片的细胞伸长长度和细胞数量显著减少[10, 15];萼片弯曲不足,不能包裹花蕾,导致早花表型,在短日照条件下开花时间比野生型早约3周,在长日照条件下开花时间比野生型早约2 d[10];种子更大,含油量更高[16-17];主根长度伸长,侧根的数目增加[18]。CLF影响拟南芥生长发育过程的多个方面,调控植物的多种性状。
温度是控制植物生长发育、分布和季节行为的关键因素,植物的整个生命周期都受到环境温度的影响,植物通过优化生长发育,不断适应自然季节温度的变化[19]。植物对温度非常敏感,甚至能感知到1 ℃的温差[20]。极端高温和低温都会对拟南芥的生长造成胁迫,导致生长缓慢、形态异常、种子产量降低[21]。在温和高温条件下,拟南芥表现出早花、下胚轴伸长、叶柄伸长、叶片变薄等性状[22];而在温和低温条件下,拟南芥表现出晚花、下胚轴变短等表型[20, 23]。本研究通过在不同温度条件(16、22 ℃)下,对野生型与clf-29突变体进行表型分析和差异表达基因分析,探索拟南芥表观遗传调控因子CLF在响应环境温度和温度形态建成中的作用。
1. 材料与方法
1.1 供试材料与引物设计
本研究使用的拟南芥植株为哥伦比亚(Col-0)生态型,其中clf-29(SALK_021003)为T-DNA插入突变体,鉴定突变体和RT-qPCR验证所需的引物见表1。
表 1 本研究用到的引物Table 1. Primers used in this study用途
Usage名称
Name序列
Sequence基因分型
Genotypingclf-29-BP 5′-ATTTTGCCGATTTCGGAAC-3′ clf-29-LP 5′-AAGAAACTTGCTAGTTCCGCC-3′ clf-29-RP 5′-GAGGCATTGACTTTGATTTGC-3′ RT-qPCR SUS1-F 5′-GGCTAGGCTTGATCGTGTCA-3′ SUS1-R 5′-GATCCACCTGAACTGACCGT-3′ LHCA1-F 5′-CAGTCCCGTGGGGTACTTTG-3′ LHCA1-R 5′-GCCGCCCGTTCTTGATCTC-3′ B1L-F 5′-AATCTCCGATGGACCGTTTGA-3′ B1L-R 5′-AGAGCTTTCTTAGCTCGCCG-3′ DIN10-F 5′-CGCTTTCTGATCTTGGAAATCGC-3′ DIN10-R 5′-ACACCGGTTAGAATCGTCCG-3′ ACTIN 2-F 5′-AGTGTTAGCTGCTGCCGCTGT-3′ ACTIN 2-R 5′-ACCAGCAAAACCAGCCTTCACCA-3′ 1.2 拟南芥生长条件和表型观察
拟南芥种子先用75%(φ)乙醇溶液消毒12 min,再用95%(φ)乙醇溶液继续消毒10 min后,在4 ℃冰箱避光放置2 d,然后在22或16 ℃长日照(16 h光∶8 h暗)条件下生长。22 ℃条件下,取用生长18 d的植株拍摄表型;16 ℃条件下,取用生长36 d的植株拍摄表型。
1.3 RNA-seq文库构建
取0.1~0.2 g植物材料迅速用液氮预冷,于研钵中研磨成粉末后使用植物总RNA提取试剂盒(Tiangen Biotech,Beijing,China)进行RNA提取,每个样品3次重复。链特异性RNA-seq文库的构建参照KAPA stranded mRNA-seq kit(Illumina® Platforms,KR0960-v5.17)进行。构建好的文库进行琼脂糖凝胶电泳,选取250~500 bp的片段进行回收纯化。文库送到上海逆耳生物科技有限公司用Illumina Hiseq 6000测序仪测序,测序模式为双端150 bp。
1.4 RNA-seq数据分析
使用cutadapt(v3.5)去除接头和测序质量低于Q20的碱基[24],再用HISAT2(v2.1.0)将筛选后的reads比对到拟南芥基因组(tair10)[25],通过SAMtools(v1.9)筛选出高比对质量的reads进行后续分析[26]。每个基因的reads数目通过featureCounts(v1.6.2)进行计算[27],并使用R包DESeq2(v1.34.0)以|log2(Fold change,FC)|≥log21.5和P<0.05的条件鉴定差异基因[28]。此外使用deeptools(v3.5.1)的bamCoverage工具将reads进行标准化处理生成bigwig文件[29],并使用IGV(v2.11.2)进行可视化[30]。分析基因的GO功能注释时使用了R包clusterProfiler(v4.2.2)[31]。
1.5 RT-qPCR
取0.1~0.2 g植物材料迅速用液氮预冷,于研钵中研磨成粉末后使用TRIzol(Invitrogen,15596026)进行RNA提取,每个样品3次重复。利用反转录试剂盒(TaKaRa,RR047A)将提取的RNA反转录成cDNA,之后利用设计好的引物(表1)进行RT-qPCR,以ACTIN 2作为内参基因,根据3次生物学重复计算平均值和标准差。
2. 结果与分析
2.1 clf-29突变体在不同温度条件下的表型分析
我们分别在22和16 ℃条件下种植拟南芥Col-0和clf-29突变体,使用三引物法对T-DNA插入材料进行鉴定,PCR结果显示,clf-29仅在T-DNA边界引物+右侧基因组引物(BP+RP)组合中才出现特异性条带,并且未出现Col-0中左侧基因组引物+右侧基因组引物(LP+RP)组合的特异性条带(图1),说明所用植物材料为纯合突变体。
16 ℃条件下,clf-29突变体植株比Col-0稍小,但整体差异不大,叶片正常伸展,略小;而在22 ℃条件下,clf-29突变体与Col-0差异明显,植株更小,叶片卷曲而且显著减小(图2、3a)。无论在22 ℃还是16 ℃,相比Col-0,clf-29突变体都表现为抽苔时间提前和莲座叶数目减少,在16和22 ℃条件下,抽苔时间分别提前6和3 d,叶片数目分别减少4和1片(图3b、3c)。clf-29突变体在不同温度条件下的形态差异,说明CLF参与叶片形态、植株大小等发育进程,暗示CLF参与调控了拟南芥的温度形态建成。
![]() 图 3 Col-0和clf-29在不同温度条件的表型统计图a统计的叶片数量为3片,图b、c统计的植株数量为25株;“*”和“***”分别表示Col-0和clf-29在P < 0.05和P < 0.001水平差异显著(t检验)Figure 3. Phenotypic statistics of Col-0 and clf-29 at different temperaturesThe number of leaves counted in figure a is 3, the number of plants counted in figure b, c is 25; “*” and “***” indicate significant differences at P < 0.05 and P < 0.001 levels between Col-0 and clf-29 respectively (t test)
图 3 Col-0和clf-29在不同温度条件的表型统计图a统计的叶片数量为3片,图b、c统计的植株数量为25株;“*”和“***”分别表示Col-0和clf-29在P < 0.05和P < 0.001水平差异显著(t检验)Figure 3. Phenotypic statistics of Col-0 and clf-29 at different temperaturesThe number of leaves counted in figure a is 3, the number of plants counted in figure b, c is 25; “*” and “***” indicate significant differences at P < 0.05 and P < 0.001 levels between Col-0 and clf-29 respectively (t test)2.2 RNA-seq数据的获取和处理
为了更深刻及全面地了解温度对clf-29突变体形态建成的调控机制,我们进行了高通量RNA-seq测序分析。使用在22 ℃培养箱生长18 d和在16 ℃培养箱生长36 d的整株植物材料进行取样、建库、测序,每个样品各有3次重复。筛选出碱基质量高的reads比对到拟南芥基因组,保留仅比对到染色质且比对质量较高的reads进行后续分析。结果显示,所有样品的原始Reads都在2 000万条以上,比对率都在97%以上,表明所建的RNA-seq文库的数据质量较高(表2)。为确定样品材料无误,查看了CLF基因的转录水平,在clf-29突变体中,CLF的表达量显著下降,而其邻近基因AT2G23370的表达量未受影响,说明试验材料是正确无误的(图4a)。此外转录组数据之间的相关性显示,每个样品的3次重复各自聚在一起,数据有良好的重复性(图4b)。
表 2 RNA-seq数据统计1)Table 2. Statistical analyses of RNA-seq dataθ/ ℃ 样品
SampleNraw Ntrim ηtrim/% Nmap ηmap/% Nfilter ηfilter/% 22 Col-0-1 37 244 778 37 224 204 99.94 36 546 723 98.18 32 101 104 87.84 Col-0-2 22 235 624 22 206 546 99.87 21 582 542 97.19 19 117 943 88.58 Col-0-3 33 187 892 33 167 450 99.94 32 477 567 97.92 22 820 849 70.27 clf-29-1 34956528 34 928 744 99.92 34 052 033 97.49 30 609 312 89.89 clf-29-2 32 170 570 32 138 512 99.90 31 122 935 96.84 27 113 262 87.12 clf-29-3 33 342 874 33 311 222 99.91 32 488 435 97.53 29 058 947 89.44 16 Col-0-1 22 773 106 22 750 524 99.90 22 163 560 97.42 20 064 343 90.53 Col-0-2 28 556 576 28 521 012 99.88 27 642 565 96.92 24 617 366 89.06 Col-0-3 29 554 426 29 506 424 99.84 28 694 997 97.25 26 892 274 93.72 clf-29-1 31 924 456 31 886 896 99.88 30 662 439 96.16 28 160 281 91.84 clf-29-2 34 539 696 34 510 886 99.92 33 692 978 97.63 29 642 353 87.98 clf-29-3 30 517 278 30 473 504 99.86 29 717 761 97.52 25 571 998 86.05 1) Nraw:原始测序的reads数目;Ntrim、ηtrim:除去低质量碱基后的reads数目和对应的比例;Nmap、ηmap:比对到拟南芥基因组的reads数目和对应的比例;Nfilter、ηfilter:比对到拟南芥染色质上且高比对质量的reads数目和对应的比例
1) Nraw: The number of raw reads; Ntrim, ηtrim: The number and corresponding proportion of reads after removing the low quality bases; Nmap, ηmap: The number and corresponding proportion of reads that aligned on Arabidopsi genome; Nfilter, ηfilter: The number and corresponding proportion of reads that aligned on Arabidopsi with high quality2.3 差异表达基因的筛选
为了探究温度对CLF的影响,以|log2FC|≥log21.5和P<0.05为筛选条件,对转录组数据进行差异基因分析。我们鉴定到了Col-0、clf-29各自在16 ℃相较于22 ℃的差异基因,其中Col-0表达上调和下调的基因分别有2 284和1 628个,clf-29突变体分别是2 345和2 077个(图5a、5b),Col-0和clf-29在响应温度变化时改变的基因数目相近。我们还鉴定到了相比于Col-0,clf-29突变体分别在22和16 ℃的差异基因,22 ℃条件下clf-29突变体相比野生型上调801个基因、下调688个基因,而在16 ℃条件下clf-29突变体相比野生型上调574个基因、下调1 012个基因(图5c、5d),说明CLF的缺失会导致大量基因异常表达。
将Col-0在16、22 ℃的差异基因和clf-29在16、22 ℃的差异基因做交集。结果发现,对于上调基因,两者的交集是1 326个基因,仅在Col-0的是958个基因,仅在clf-29的是1 019个基因;对于下调基因,两者的交集是919个基因,仅在Col-0的是709个基因,仅在clf-29的是1 158个基因(图6a、6b)。在不同温度条件下,Col-0和clf-29有很多基因是共同改变的(图7),而那些仅在Col-0或者仅在clf-29变化的基因更为重要。我们对这些基因进行GO富集分析,发现仅在Col-0上调的基因更多地富集在环境响应、物质转运等途径(图8a);仅在clf-29上调的基因主要富集在响应刺激、激素运输、细胞壁生长等途径(图8b);仅在Col-0下调的基因富集在物质合成、物质代谢等途径(图8c);仅在clf-29下调的基因富集在刺激响应、合成代谢调控等途径(图8d)。
2.4 温度响应基因的改变
温度的变化造成了clf-29突变体的不同表型,因此我们找到了上述4类差异表达基因(图6),相比于22 ℃,在16 ℃条件下在Col-0显著上调而在clf-29中未显著上调的,称之为Col-0上调,同样得到了clf-29上调、Col-0下调、clf-29下调,通过GO注释在这4类基因中寻找与温度响应相关的基因进行分析。有19个基因仅在Col-0上调表达,有14个基因仅在clf-29上调表达(图9a);仅在Col-0下调的是26个基因,仅在clf-29下调的是37个基因(图9b)。在这4类基因中各选取特定基因SUCROSE SYNTHASE 1(SUS1)、PHOTOSYSTEM Ⅰ LIGHT HARVESTING COMPLEX GENE 1(LHCA1)、BYPASS1-LIKE(B1L)、DARK INDUCIBLE 10 (DIN10)进行RT-qPCR验证(材料情况与构建RNA-seq文库一致),相比于clf-29,SUS1在Col-0上调更加显著,与之类似,LHCA1在clf-29上调更加显著,B1L在Col-0、DIN10在clf-29的下调程度更高(图10),这与热图结果基本一致。Col-0材料在12 ℃处理1、3、5、10 h后LHCA1表达量上升,处理24 h后表达量下降[32],我们的结果与短时间处理一致;Col-0材料在4 ℃处理1、3、6、12 h后B1L表达量上升[33],处理24 h后表达量下降,我们的结果与处理24 h一致。我们的结果与其他已发表文章结果的差异,可能是处理温度与处理时间都存在差别而共同导致的。综上,CLF的缺失造成了大量温度响应基因表达改变,造成一系列的表型差异,证明了CLF在不同温度条件下调控生长发育的重要作用。
3. 讨论与结论
拟南芥中的CLF是PRC2复合物的重要亚基,是H3K27me3甲基转移酶,CLF蛋白结构保守,在多种植物如水稻、番茄中都报道过CLF的同源蛋白[34-35]。CLF调控植物生长发育的多个阶段,在拟南芥中鉴定到6 854个H3K27me3富集的基因,这些基因都是潜在的CLF可以调控的靶基因,涉及多种生长发育、响应刺激相关的通路,暗示CLF对植物的正常生长起至关重要的作用[14]。本研究发现不同温度条件下clf-29突变体的形态建成存在差异,并且找到了多类可能导致表型差异的重要基因,其中仅在Col-0响应而在clf-29突变体未响应的基因说明CLF的缺失使突变体无法正常发挥功能,仅在clf-29响应的差异基因说明缺失CLF使其产生了更多的变化,并找到了多个和温度相关的基因,其发生明显的表达变化,这些结果说明CLF是参与拟南芥温度形态建成的重要表观调控因子。
不同温度条件下clf-29突变体的形态差异,极有可能是因为在不同温度下CLF介导的H3K27甲基化在基因组上的不同丰度造成的。温度是一种重要的环境影响因子,低温会影响拟南芥H3K27me3的富集。在低温条件(4 ℃)下,低温响应基因COR15A和ATGOLS3的H3K27me3水平会降低从而导致基因表达的变化[36-37]。温度变化也可以通过改变开花抑制基因FLOWERING LOCUS C(FLC)的H3K27me3水平来调控FLC的活性,从而调控植物开花时间[38-39]。然而温度如何影响甲基转移酶CLF介导的H3K27甲基化的分布,是需要进一步探索的问题。
除了H3K27甲基化以外,其他的表观遗传标记也参与植物对环境温度的适应性调控,当植物生长环境的温度较低时,组蛋白变体H2A.Z会富集在DNA上,抑制基因的表达,当温度升高时,H2A.Z富集程度降低,使基因正常表达[20]。因此,在不同温度条件下,H3K27me3与其他的表观遗传标记会产生怎样的偶联反应也是值得深思和深入探究的问题。
-
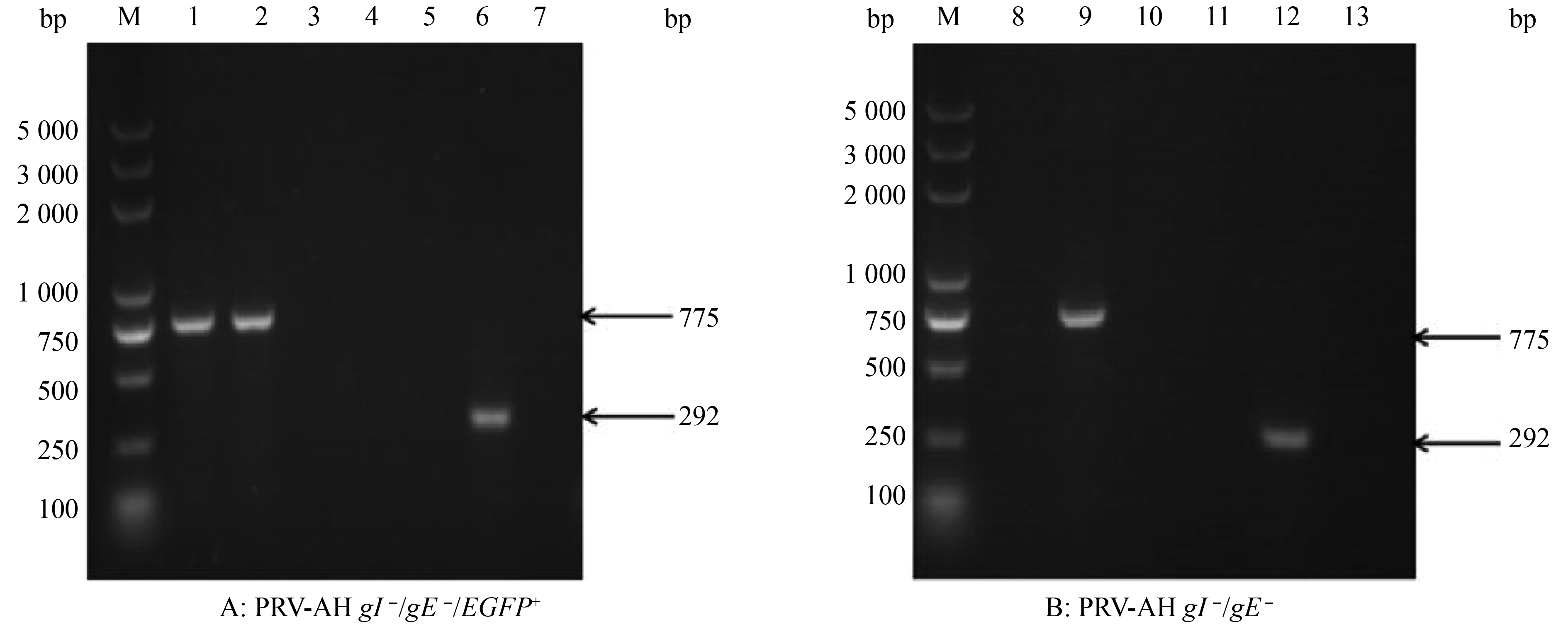
图 3 重组病毒PRV-AH gI –/gE –/EGFP+与PRV-AH gI –/gE –的PCR鉴定结果
M:DL5000 DNA Marker;1~4:EGFP扩增结果(775 bp);5~7:gE片段扩增结果(292 bp);1、5:PRV-AH gI –/gE –/EGFP+; 2:pEGFP-N1;3、6:PRV AH;4、7:阴性对照;8~10:EGFP片段扩增结果(775 bp);11~13:gE片段扩增结果(292 bp);8、11:PRV AH gI –/gE –;9:pEGFP-N1;10、13:阴性对照;12:PRV AH
Figure 3. Identification of recombinant virus PRV-AH gI –/gE –/EGFP+ and PRV-AH gI –/gE – by PCR
图 4 重组病毒感染BHK-21细胞的病变情况
A1、B1、C1:普通光观察PRV AH、PRV AH gE –/gI –/EGFP+及PRV AH gE –/gI –感染BHK-21细胞的病变;A2、B2、C2:荧光观察PRV AH、PRV AH gE –/gI –/EGFP+及PRV AH gE –/gI –感染BHK-21细胞的病变;A3、B3、C3:普通光与荧光观察结果的叠加图(merge);D1、D2、D3:未感染病毒的BHK-21细胞
Figure 4. Cytopathic effect of BHK-21 cells infected by recombinant viruses
表 1 本试验所涉及的引物
Table 1 Primers used in this study
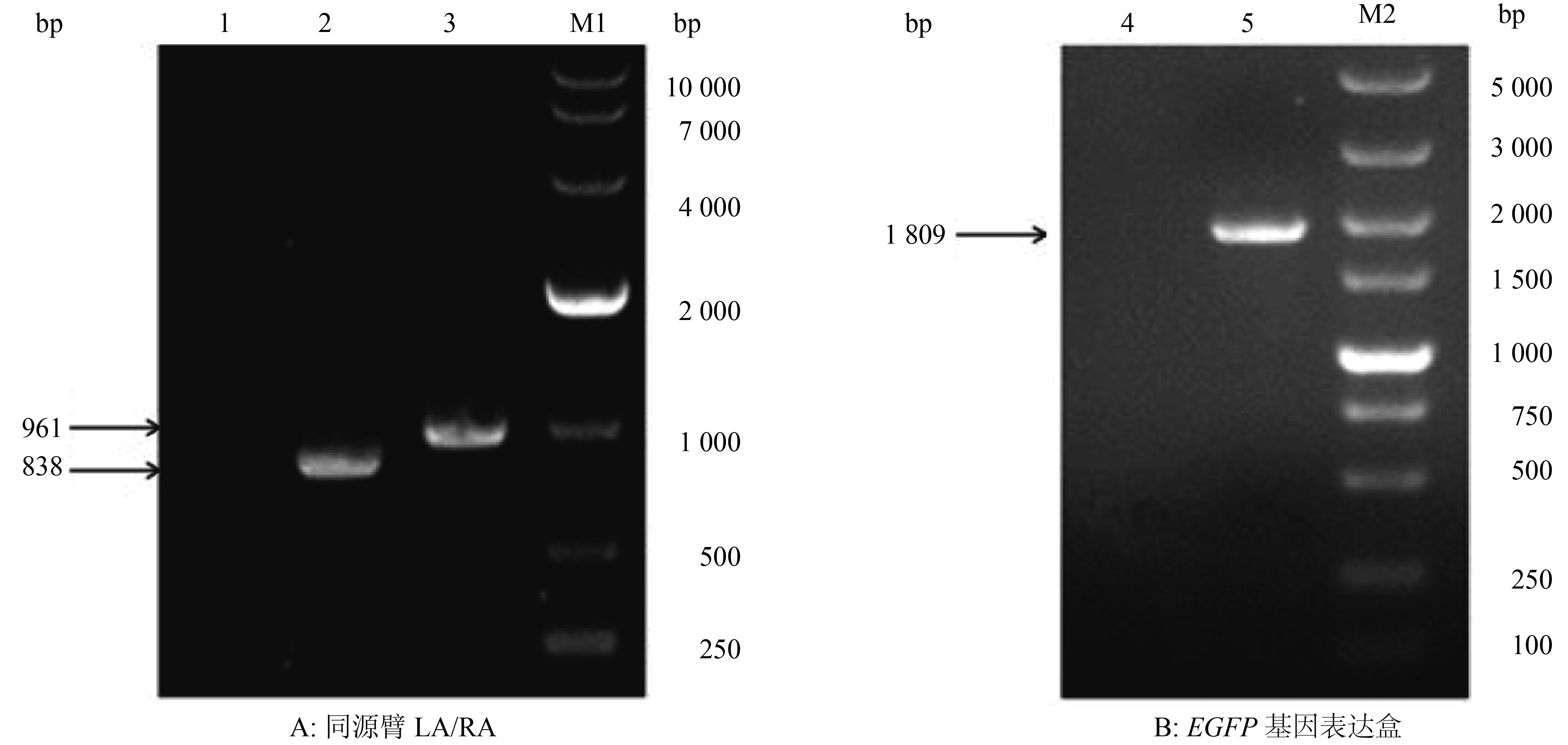
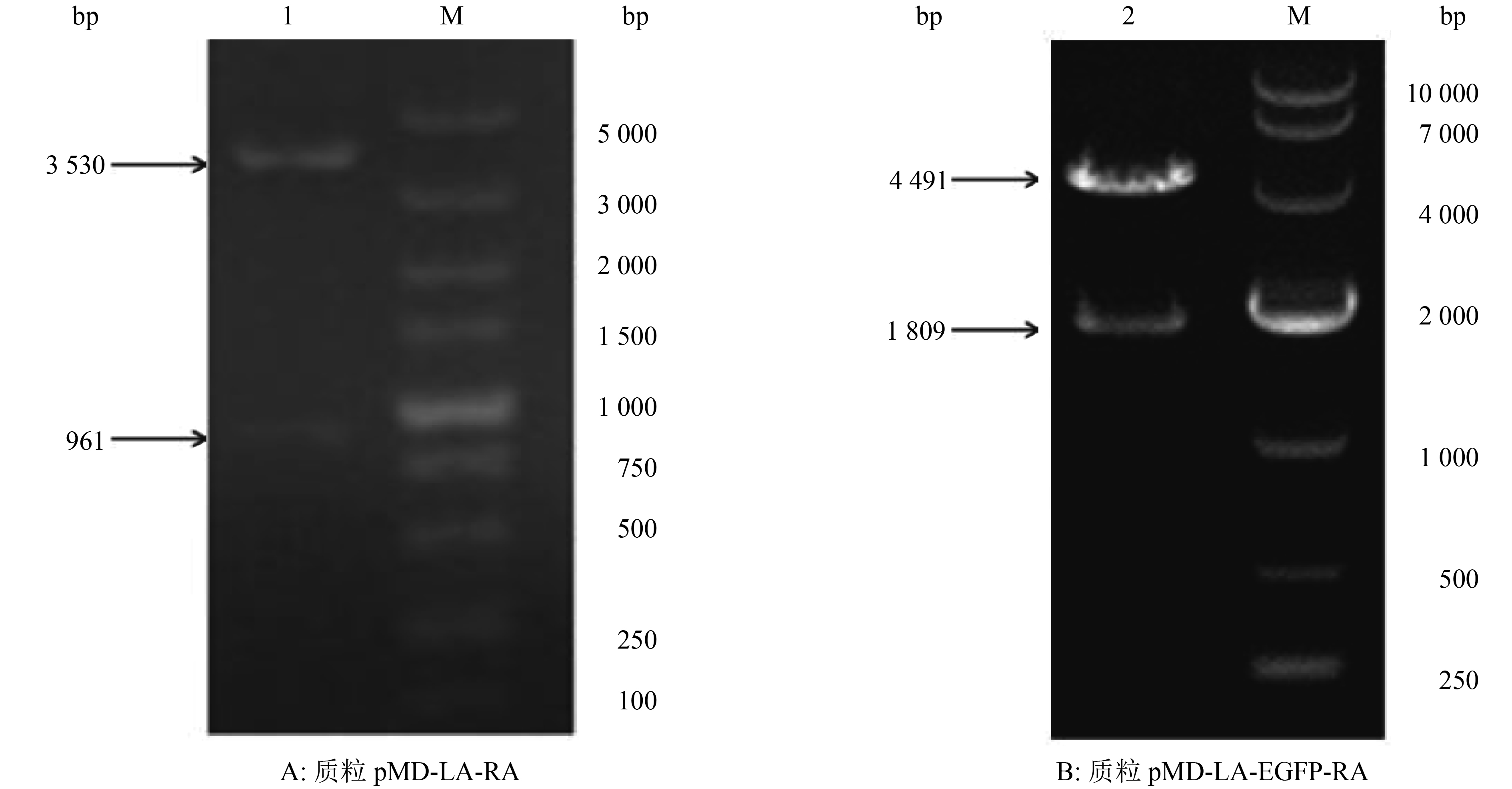
引物名称 引物序列(5′→3′) 扩增位置(碱基位点) 扩增长度/bp LA-F CCGGAATTCACCAGCACCGCACGTACAAGTT 120 511~121 349 838 LA-R CAGCAGCGTCCCGTCTATCGT RA-F AAACTGCAGGATATCCGGAAGTGACGAATGG 124 033~124 994 961 RA-R CTCGGTGGTGATGTAGAAAAGCTTGGG EGFP-F1 AACGATATCGTTTAAACGTTCTTTCCTGCGTTATCC 4 679~4 733;0~1 755 1 809 EGFP-R1 AACGATATCAACCCTATCTCGGTCTATTCT EGFP-F2 GTGGATAGCGGTTTGACTC 396~1 170 775 EGFP-R2 CACCTTGATGCCGTTCTT gE-F GTGATGACCCACAACGG 123 426~123 717 292 gE-R GCACGCAGAGCCAGAT LA-F2 AGTACCGGCGTCGATGATGA 121 144~121 348;
124 033~124 137310 RA-R2 GGTCAAACGTGTCCATGTCG 表 2 不同剂量PRV AH gE –/gI –灭活疫苗免疫小鼠后的中和抗体水平
Table 2 Neutralizing antibody titers of immunized mice with different dosages of PRV AH gE –/gI – inactivated vaccine
组别(免疫剂量)1) 首次免疫后不同时间的抗性水平 0 2周 4周 5周 6周 7周 8周 A(105.0TCID50) <2 <2 <2 <2 2.45±4.60 7.35±2.70 4.36±0.14 B(105.0TCID50) <2 <2 <2 <2 2.13±0.30 6.88±0.27 4.18±0.30 C(106.0TCID50) <2 <2 4.39±2.43 7.83±3.17 8.96±4.35 19.13±3.08 9.46±2.72 D(106.0TCID50) <2 <2 3.98±1.54 7.37±5.86 7.70±4.73 15.73±6.06 9.28±6.89 E、F、G(未免疫对照) <2 <2 <2 <2 <2 <2 <2 1) A、C、E为PRV AH株;B、D、F为PRV经典株;G为空白对照 -
[1] 童光志, 陈焕春. 伪狂犬病流行现状及我国应采取的防制措施[J]. 中国兽医学报, 1999, 19(1): 1-2. [2] 彭金美, 安同庆, 赵鸿远, 等. 猪伪狂犬病病毒新流行株的分离鉴定及抗原差异性分析[J]. 中国预防兽医学报, 2013, 35(1): 1-4. [3] 童武, 张青占, 郑浩, 等. 免疫后发病仔猪中伪狂犬病毒的分离和鉴定[J]. 中国动物传染病学报, 2013, 21(3): 1-7. [4] 叶培根. 猪伪狂犬病: 剖析猪伪狂犬病的新动向[J]. 北方牧业, 2013, 12: 23. [5] AN T Q, PENG J M, TIAN Z J, et al. Pseudorabies virus variant in Bartha-K61-vaccinated pigs, China, 2012[J]. Emerg Infect Dis, 2013, 19(11): 1749-1755.
[6] WU R, BAI C, SUN J, et al. Emergence of virulent pseudorabies virus infection in Northern China[J]. J Vet Sci, 2013, 14(3): 363-365.
[7] YU X L, ZHOU Z, HU D M, et al. Pathogenic pseudorabies virus, China, 2012[J]. Emerg Infect Dis, 2014, 20(1): 102-104.
[8] LUO Y Z, LI N, CONG X, et al. Pathogenicity and genomic characterization of a pseudorabies virus variant isolated from Bartha-K61-vaccinated swine population in China[J]. Vet Microbiol, 2014, 174(1/2): 107-115.
[9] 向柯宇, 潘慧, 吉艺宽, 等. 猪伪狂犬病病毒新流行株主要糖蛋白的分子特征分析[J]. 华南农业大学学报, 2016, 37(3): 23-28. [10] WANG C H, YUAN J, QIN H Y, et al. A novel gE-deleted pseudorabies virus (PRV) provides rapid and complete protection from lethal challenge with the PRV variant emerging in Bartha-K61-vaccinated swine population in China[J]. Vaccine, 2014, 32(27): 3379-3385.
[11] GU Z Q, DONG J, WANG J C, et al. A novel inactivated gE/gI deleted pseudorabies virus (PRV) vaccine completely protects pigs from an emerged variant PRV challenge[J]. Virus Res, 2015, 195: 57-63.
[12] TONG W, LI G X, LIANG C, et al. A live, attenuated pseudorabies virus strain JS-2012 deleted for gE/gI protects against both classical and emerging strains[J]. Antiviral Res, 2016, 130: 110-117.
[13] ZHANG C L, GUO L H, JIA X R, et al. Construction of a triple gene-deleted Chinese pseudorabies virus variant and its efficacy study as a vaccine candidate on suckling piglets[J]. Vaccine, 2015, 33(21): 2432-2437.
[14] HU R M, ZHOU Q, SONG W B, et al. Novel pseudorabies virus variant with defects in TK, gE and gI protects growing pigs against lethal challenge[J]. Vaccine, 2015, 33(43): 5733-5740.
[15] CONG X, LEI J L, XIA S L, et al. Pathogenicity and immunogenicity of a gE/gI/TK gene-deleted pseudorabies virus variant in susceptible animals[J]. Vet Microbiol, 2016, 182: 170-177.
[16] LIANG X, SUN L Q, YU T, et al. A CRISPR/Cas9 and Cre/Lox system-based express vaccine development strategy against re-emerging Pseudorabies virus[J]. Sci Rep, 2016, 6: 19176.



 下载:
下载: