Over-expression of rabies virus G protein and its inhibitory effect on the virus in neuroblastoma cells
-
摘要:目的
探究G蛋白在狂犬病病毒(Rabies virus,RABV)复制中的作用,以揭示携带双G基因的重组RABV的Hep-dG与亲代毒株rHep-Flury在神经母细胞瘤(NA)细胞中滴度差异的原因,为RABV致病机制的研究奠定基础。
方法通过病毒吸附、入侵、荧光定量PCR、Western-blot以及中和抗体阻断等试验,检测G蛋白过表达对IFN-β以及相关因子转录的影响。
结果Hep-dG感染能显著上调NA细胞中IFN-β mRNA的表达,激活了下游因子STAT1的表达与磷酸化,在较低的感染复数(MOI=0.01)下,Hep-dG感染后24 h即可显著促进IFN-β 基因的表达,36 h达到最高水平(P<0.001)。该病毒进入细胞后,产生了更多的病毒Leader RNA和RIG-I mRNA,且与IFN-β mRNA的表达高度一致。抗体阻断IFN-β后,Hep-dG在NA细胞中的病毒滴度显著上升(P<0.01),约为阻断前的7.9倍,且与亲代毒株rHep-Flury无显著差异。与阴性对照比较,5 μg的pH-G质粒转染能刺激IFN-β的转录(P<0.05),表明真核表达RABV G蛋白能在一定程度上刺激IFN-β的转录。
结论本研究初步揭示了G蛋白激活先天性免疫应答的原因和作用。RABV G蛋白的过表达,通过促进病毒Leader RNA的转录,激活了RIG-I介导的IFN-β通路,进而抑制了Hep-dG在NA细胞的繁殖。G蛋白的过表达也对干扰素通路起到一定的作用。
Abstract:ObjectiveTo explore the role of G protein in rabies virus (RABV) replication, reveal the reason for the difference of virus titer in neuroblastoma (NA) cells between the recombinant RABV Hep-dG with dual copy of G gene and the parental strain rHep-Flury, and lay a foundation for the study of RABV pathogenesis.
MethodThe effects of G protein over-expression on transcriptions ofIFN-β and related factors were examined by the virus binding assay, virus entry assay, fluorescence quantitative PCR, Western-blot and neutralizing antibody blocking assay.
ResultHep-dG infection significantly increased the expression of IFN-β mRNA and activated the expression of the downstream factor STAT1 in NA cells. Under the low multiplicity of infection (MOI=0.01), the expression of IFN-β gene significantly increased at 24 h after Hep-dG infection and reached the highest level at 36 h. After the virus entered the cells, there were more viral Leader RNA and RIG-I mRNA, which were highly consistent with the expression of IFN-β mRNA. The block of IFN-β expression by neutralizing antibody in NA cells significantly increased the virus titer of Hep-dG in cell culture supernatant(P<0.01), which was 7.9 times before blocking. Meanwhile the virus titer of Hep-dG had no significant difference with the parental strain rHep-Flury. Compared with the negative control, transfection of 5 μg pH-G plasmid could stimulate the transcription ofIFN-β(P<0.05), which showed that eukaryotic expression of RABV G protein could stimulateIFN-β transcription to a certain extent.
ConclusionThis study preliminarily reveals the cause and role of G protein in activating innate immune response. Over-expression of RABV G protein activates the RIG-I-mediated IFN-β pathway by promoting transcription of the viral Leader RNA, which in turn inhibits Hep-dG replication in NA cells and finally results in the lower virus titer in NA cells.
-
Keywords:
- rabies virus /
- glycoprotein /
- IFN-β pathway /
- interferon-β /
- virus titer
-
狂犬病是一种重大的人畜共患病,它是由弹状病毒科Rhabdoviridae狂犬病病毒属Lyssavirus的狂犬病病毒(Rabies virus,RABV)引起。RABV基因组编码5种结构蛋白质,分别是核蛋白(N蛋白)、磷蛋白(P蛋白)、基质蛋白(M蛋白)、跨膜糖蛋白(G蛋白)和病毒聚合酶蛋白(L蛋白)。病毒基因组RNA与核蛋白、磷蛋白和聚合酶蛋白缠绕组成核糖核蛋白(RNP),成为RABV的复制单位[1-2]。M蛋白在病毒装配和出芽过程中发挥重要作用,其在G蛋白的辅助作用下介导病毒粒子和细胞的分离,从而顺利出芽[3-4]。G蛋白是唯一刺激机体产生中和抗体的表面抗原,同时也能诱导T细胞,刺激机体产生细胞免疫,不仅能在中枢神经系统(CNS)中清除病毒,也能诱导神经细胞发生凋亡[5-7]。研究表明,RABV感染后发生的细胞凋亡主要由G蛋白引起,且病毒的毒力越弱诱导的凋亡越严重[8-10]。
前期的研究发现,携带双G基因的重组RABV Hep-dG感染神经母细胞瘤(Neuroblastoma, NA)细胞后病毒滴度明显低于亲代毒株rHep-Flury[11],而在BHK-21细胞中没有相同现象[8]。蛋白质组研究发现,Hep-dG感染NA细胞后诱发干扰素通路上游和下游蛋白的大量上调,生物信息学预测结合Western-blot和荧光定量PCR证明干扰素通路仅在Hep-dG感染组强烈激活,表明RABV弱毒病毒G蛋白的过表达与干扰素的诱导呈高度的正相关[11],由于BHK-21是RIG-I介导IFN通路的缺陷型细胞[12],推测2株重组RABV在BHK-21细胞和NA细胞中的病毒滴度可能与干扰素通路的激活有关。在此基础之上,本研究进一步研究重组RABV感染NA细胞诱导产生干扰素的原因,以揭示G蛋白与干扰素诱导之间的内在关系;并通过抗体阻断试验,证明干扰素诱导在RABV复制中的作用。
1. 材料与方法
1.1 材料
1.1.1 细胞和病毒
鼠NA细胞,购自武汉生物制品研究所,在含体积分数为10%胎牛血清的RPMI 1640中培养;亲代RABV rHep-Flury和携带双G基因的重组RABV Hep-dG[8],保存于华南农业大学兽医微生物实验室。
1.1.2 质粒
pH-G(含有狂犬病病毒Hep-Flury株的G基因),由日本国立传染病研究所Dr. Morimoto教授惠赠。pH-M和pH-GFP,均由华南农业大学兽医微生物实验室罗永文博士在pcDNA3.2插入了Hep-Flury的M基因和GFP基因而构建。
1.1.3 主要试剂
STAT1、p-STAT1和β-actin单克隆抗体,HRP标记的山羊抗鼠IgG、HRP标记的山羊抗兔IgG和绿色荧光Dylight 488标记的山羊抗鼠IgG,均为美国Bioworld公司产品。小鼠抗IFN-β中和抗体和正常鼠源IgG阴性对照,购自美国R&D公司。Clontech Xfect质粒转染试剂购自日本TaKaRa公司。荧光定量PCR试剂盒(THUNDERBIRD SYBR qPCR Mix)购自日本东洋纺公司。抗RABV G蛋白和M蛋白单克隆抗体由华南农业大学兽医微生物实验室制备。
1.2 方法
1.2.1 荧光定量PCR检测IFN-β mRNA的表达
将rHep-Flury和Hep-dG以相同的感染复数(MOI=0.01)感染6孔板中的NA细胞,同时设未接毒的细胞为空白对照。在接毒后12、24、36和48 h,用1 mL 4 ℃预冷的PBS(pH7.4,0.01 mol·L–1)洗涤细胞1次,再加入1 mL Trizol,抽提RNA,定量检测不同时间IFN-β mRNA的表达。
1.2.2 Western-blot检测STAT1和p-STAT1的表达
将rHep-Flury和Hep-dG以相同的感染复数(MOI=0.01)感染6孔板中的NA细胞,同时设未接毒的细胞作为空白对照。在接毒后24和48 h,分别提取细胞蛋白,进行STAT1和p-STAT1的蛋白定量和Western-blot检测。
1.2.3 重组病毒吸附细胞能力的检测
将rHep-Flury和Hep-dG分别以MOI为0.01、0.10和1.00接种6孔板中的NA细胞,同时设未接毒的细胞作为空白对照。细胞在4 ℃冰箱内放置2 h后,PBS洗去未结合的病毒粒子,然后提取细胞RNA,以特异引物vRNA-F和vRNA-R(表1)进行荧光定量PCR(同时以GAPDH-F和GAPDH-R为参照)检测细胞表面病毒的基因组vRNA,由此可检测不同重组RABV的吸附能力[13]。
表 1 荧光定量PCR检测引物Table 1. Primers used for fluorescence quantitative PCR detection被检基因 引物序列 引物名称 产物长度/bp Leader RNA Leader-F 5'-CCAGATGCTTGGCGTCCT-3' 67 Leader-R 5'-ACGCTTAACAACAAAACC-3' vRNA vRNA-F 5'-GGAAAAGGGACATTTGAAAGAA-3' 130 vRNA-R 5'-AGTCCTCGTCATCGGAGTTGAC-3' RIG-I RIGI-F 5'-GCAAGTGCTTCCTCCTGACC-3' 142 RIGI-R 5'-ATGCGGTGAACCGTCTTTCC-3' GAPDH GAPDH-F 5'-AGAGTGTTTCCTCGTCCCGT-3' 199 GAPDH-R 5'-CTGTGCCGTTGAATTTGCCG-3' 1.2.4 重组病毒入侵细胞能力的检测
将rHep-Flury和Hep-dG分别以MOI为0.01、0.10和1.00接种6孔板中的NA细胞,同时设未接毒的细胞为空白对照。细胞在4 ℃冰箱内放置2 h后,PBS洗去未结合的病毒粒子,继续在37 ℃细胞培养箱放置2 h。用质量分数为0.25%不含EDTA的胰酶处理病毒吸附的细胞膜,去掉细胞膜表面RABV,然后提取细胞RNA,用荧光定量PCR检测内部残余的病毒基因组vRNA,即为入侵细胞的病毒量。
1.2.5 重组病毒Leader RNA、vRNA和RIG-I转录水平的检测
将rHep-Flury和Hep-dG以MOI=0.01接种12孔板中的NA细胞,同时设未接毒的NA细胞作为空白对照。分别在感染后12、24、36和48 h取出细胞,弃培养液。用PBS洗涤细胞1次,每孔加入500 μL Trizol裂解液,抽提样品RNA。以5'-CCAGATGCTTGGCGTCCTCTTTGCAACTGACGATGT-3'和5'-GGAAAAGGGACATTTGAAAGAA-3'为反转录引物,分别合成RABV Leader RNA和vRNA的cDNA。RIG-I的反转录不需要特异性的反转引物,使用ReverTra Ace® qPCR RT Master Mix with gDNA Remover逆转录反应试剂盒进行反转录。以表1的特异引物将获得的cDNA进行荧光定量PCR检测,同时GAPDH作为内参引物。
1.2.6 重组病毒Leader RNA的二级结构分析
通过RNA fold Web SeVer在线软件分析2株重组RABV Leader RNA的二级结构。
1.2.7 pH-G与pH-M质粒在NA细胞上的表达
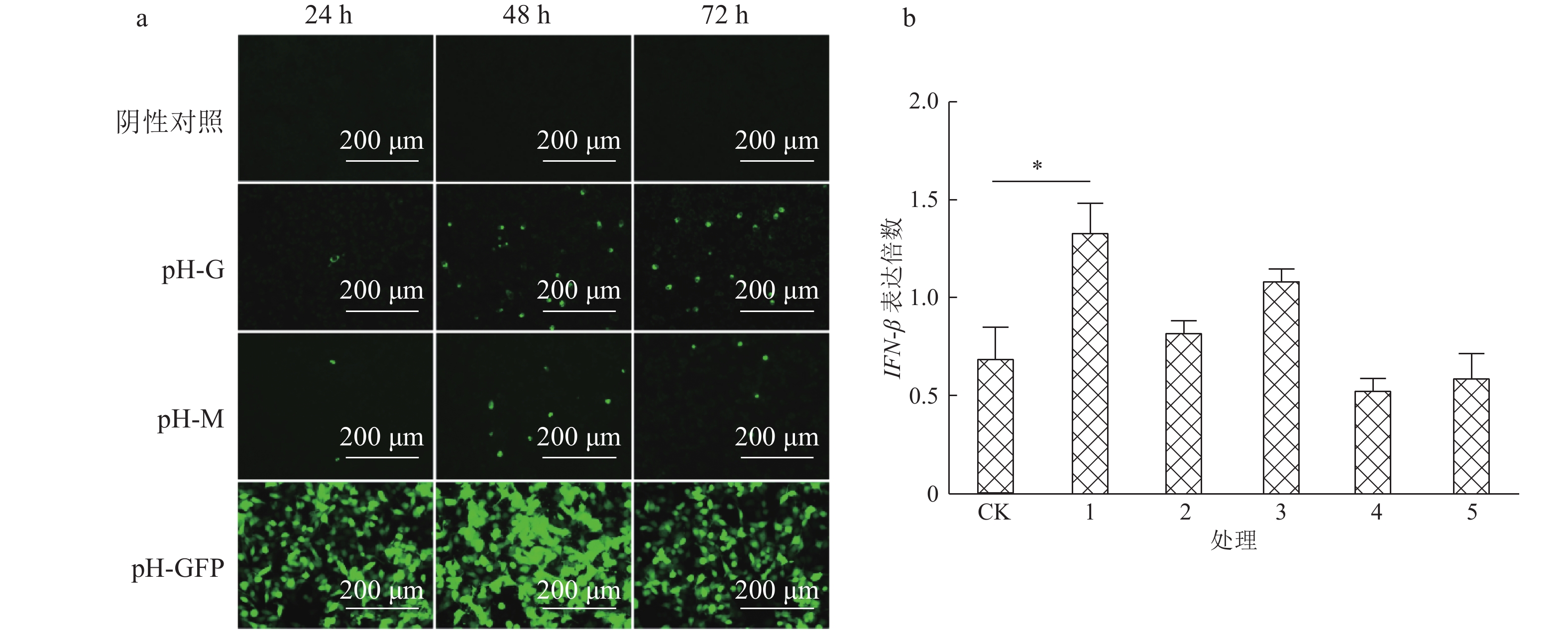
将5 μg质粒DNA用转染缓冲液稀释至最终体积为100 μL,添加1.5 μL Xfect Polymer,在涡旋混匀器中混匀10 s。室温孵育10 min后,加入到细胞汇合度为50%~70%、37 ℃培养了12~16 h的NA细胞中。37 ℃继续培养4 h后,更换培养液后,放入37 ℃细胞培养箱,分别在24、48和72 h后取出,加入抗G和M蛋白抗体,进行间接免疫荧光检测。
1.2.8 IFN-β的转录水平和抗体阻断试验
将pH-G与pH-M质粒DNA按上述方法转染NA细胞,在合适的时间点取出,提取RNA,荧光定量PCR检测真核表达G、M蛋白后IFN-β的转录水平。
将NA细胞铺于6孔板中,37 ℃培养12~16 h。待细胞汇合度为80%,弃培养液,在各孔中加入2 mL无血清的DMEM和Anti-IFN-β(10 mg·L–1),预处理细胞1 h。将rHep-Flury和Hep-dG以MOI=0.01接种NA细胞,在37 ℃吸附1 h,同时设未接毒的NA细胞作为空白对照。更换含有Anti-IFN-β的新鲜培养基。在接毒后48 h,收集上清液,检测RABV的病毒滴度。同时,提取细胞RNA,用荧光定量PCR检测IFN-β的阻断情况[14]。
2. 结果与分析
2.1 重组病毒感染NA细胞对IFN-β通路的影响
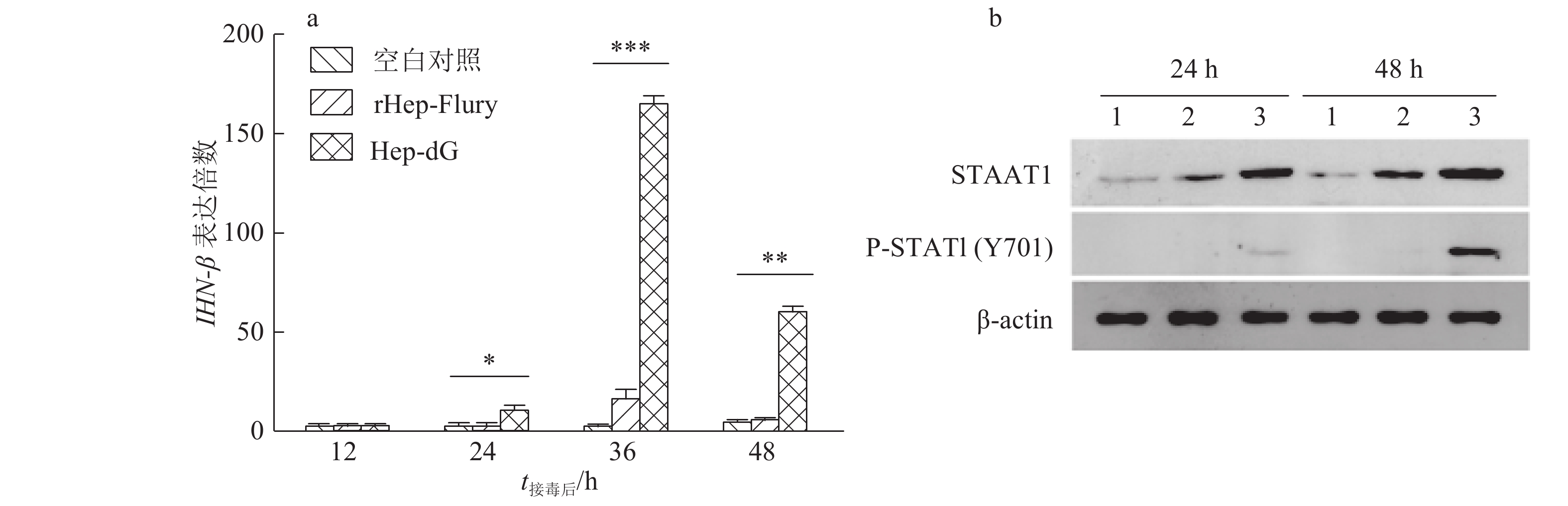
为了确定重组RABV感染对NA细胞产生IFN-β的影响,在接毒后12、24、36和48 h,通过荧光定量PCR分别对rHep-Flury和Hep-dG感染细胞和空白对照细胞IFN-β的转录水平进行了分析,结果如图1a所示。在较低的感染复数(MOI=0.01)下,Hep-dG感染后24 h即可显著促进IFN-β基因的表达,36 h达到最高水平(P<0.001)。同时,Hep-dG感染24和48 h均可激活STAT1的磷酸化,Hep-dG感染组的STAT1的表达水平较亲代毒株rHep-Flury明显提高(图1b)。由此说明,在NA细胞中,Hep-dG感染能显著上调IFN-β mRNA的表达和激活干扰素下游因子STAT1。
2.2 重组病毒对NA细胞的吸附能力和入侵能力分析
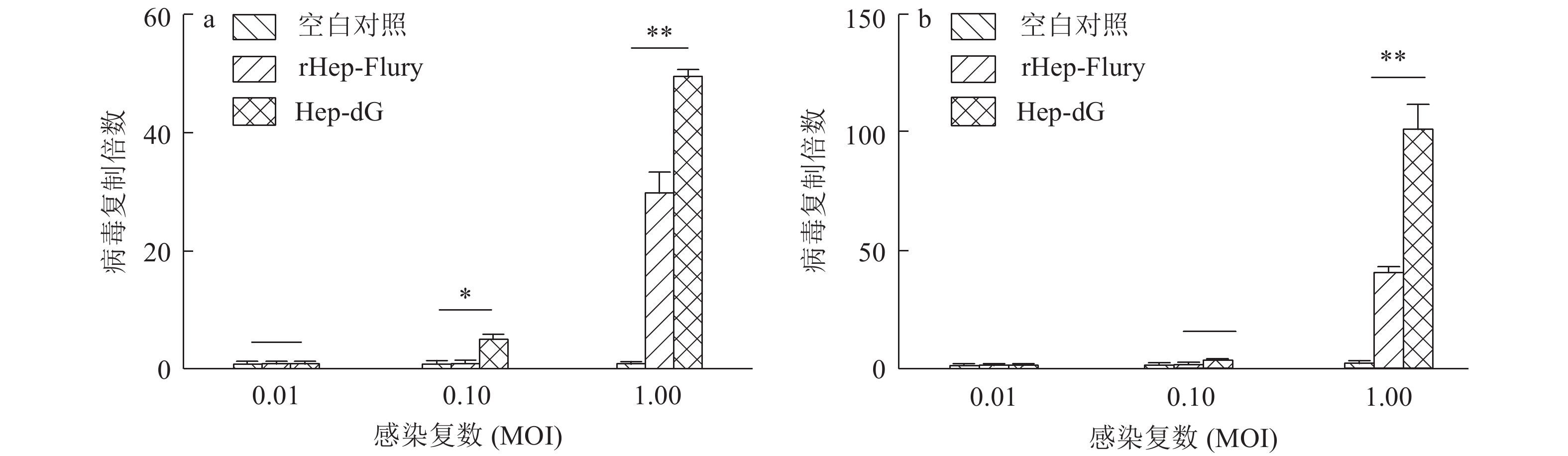
为了比较G蛋白过表达的Hep-dG株与亲代毒株rHep-Flury对NA细胞的吸附能力,我们模拟了病毒吸附NA细胞的过程,并用荧光定量PCR检测了细胞表面病毒。结果(图2a)表明,Hep-dG在不同感染复数下对NA细胞的吸附能力均高于rHep-Flury。在很低的感染复数(MOI=0.01)下,Hep-dG对NA细胞的吸附能力也明显高于rHep-Flury(P<0.01)。为了比较G蛋白过表达的Hep-dG与rHep-Flury对NA细胞的入侵能力,我们模拟了病毒进入NA细胞的过程,并用荧光定量PCR检测了入侵细胞的病毒量。结果(图2b)表明,在较高感染复数(MOI=1.00)下,Hep-dG进入NA细胞的能力显著高于rHep-Flury(P<0.01);而在很低的感染复数(MOI=0.01)下,Hep-dG进入NA细胞的能力与rHep-Flury无显著性差异。表明在较高的感染复数下,G蛋白的过表达显著提高了RABV对神经细胞的吸附与入侵能力。
2.3 重组病毒Leader RNA和vRNA与RIG-I基因的关系
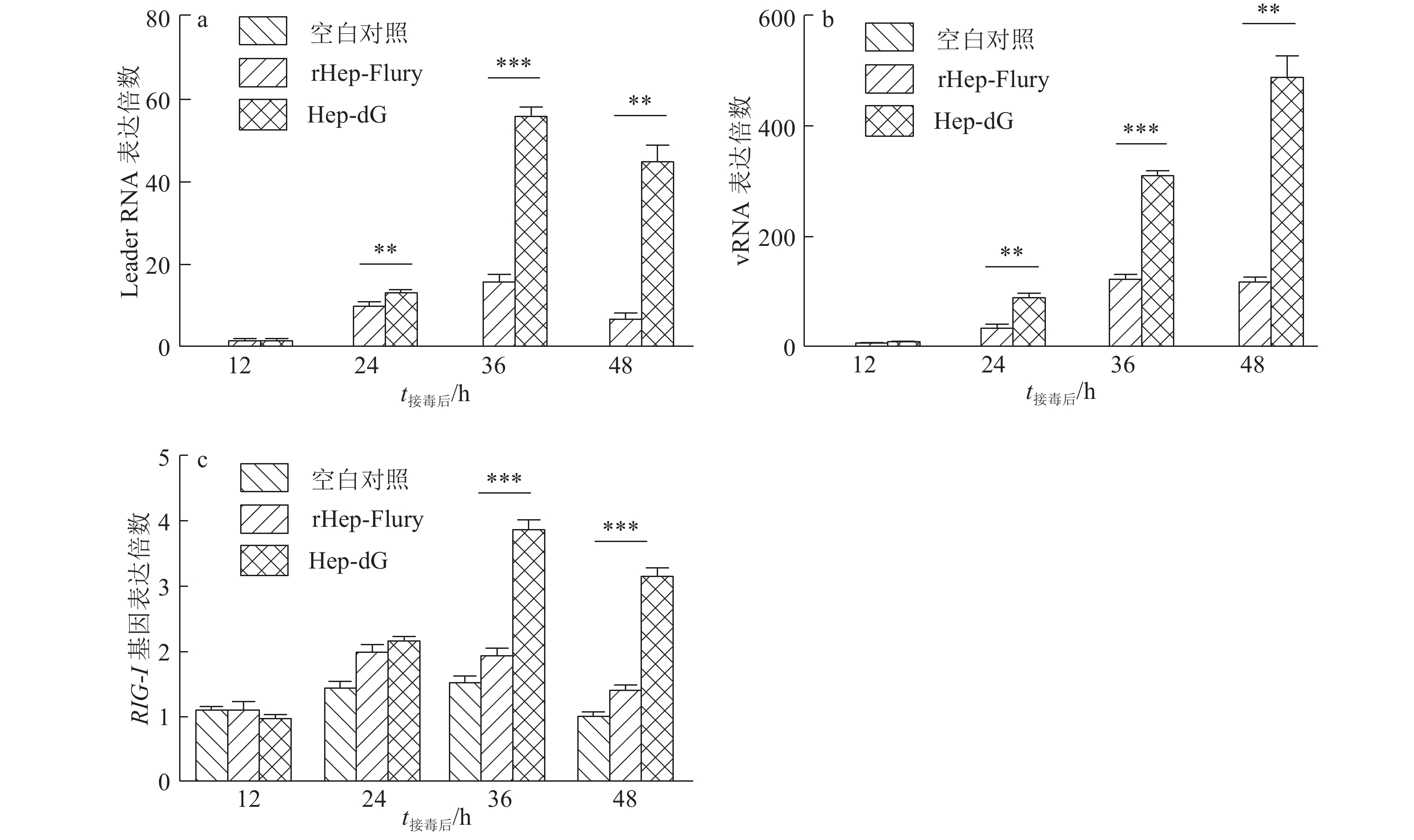
为了研究重组RABV在细胞内Leader RNA的表达差异,将Hep-dG与rHep-Flury以相同的感染复数(MOI=0.01)感染NA细胞,利用荧光定量PCR检测了不同时间点细胞内Leader RNA的含量,同时检测了病毒基因组vRNA和RIG-I基因的转录水平,结果见图3。图3a显示,在感染后的不同时间点,Hep-dG Leader RNA在NA细胞中的含量明显高于亲代毒株rHep-Flury,其趋势与IFN-β基因的表达(图1a)一致。Hep-dG Leader RNA的表达量在接毒后36 h达到最高水平,48 h有所下降,但仍然显著高于亲代毒株rHep-Flury(P<0.01)。在感染36 h前,细胞内Leader RNA与病毒基因组vRNA的含量趋势一致;在感染36 h后,Leader RNA的表达水平开始下降,而NA细胞内vRNA的含量继续升高(图3b)。同时,RABV感染后,细胞内RIG-I基因的变化趋势与Leader RNA一致;在接毒后36和48 h,Hep-dG感染的NA细胞中RIG-I基因的表达量显著高于亲代毒株rHep-Flury(图3c, P<0.001)。表明Hep-dG感染NA细胞后,病毒的Leader RNA和vRNA升高,诱导RIG-I mRNA增多,导致了IFN-β mRNA增多。
2.4 重组病毒Leader RNA的二级结构预测
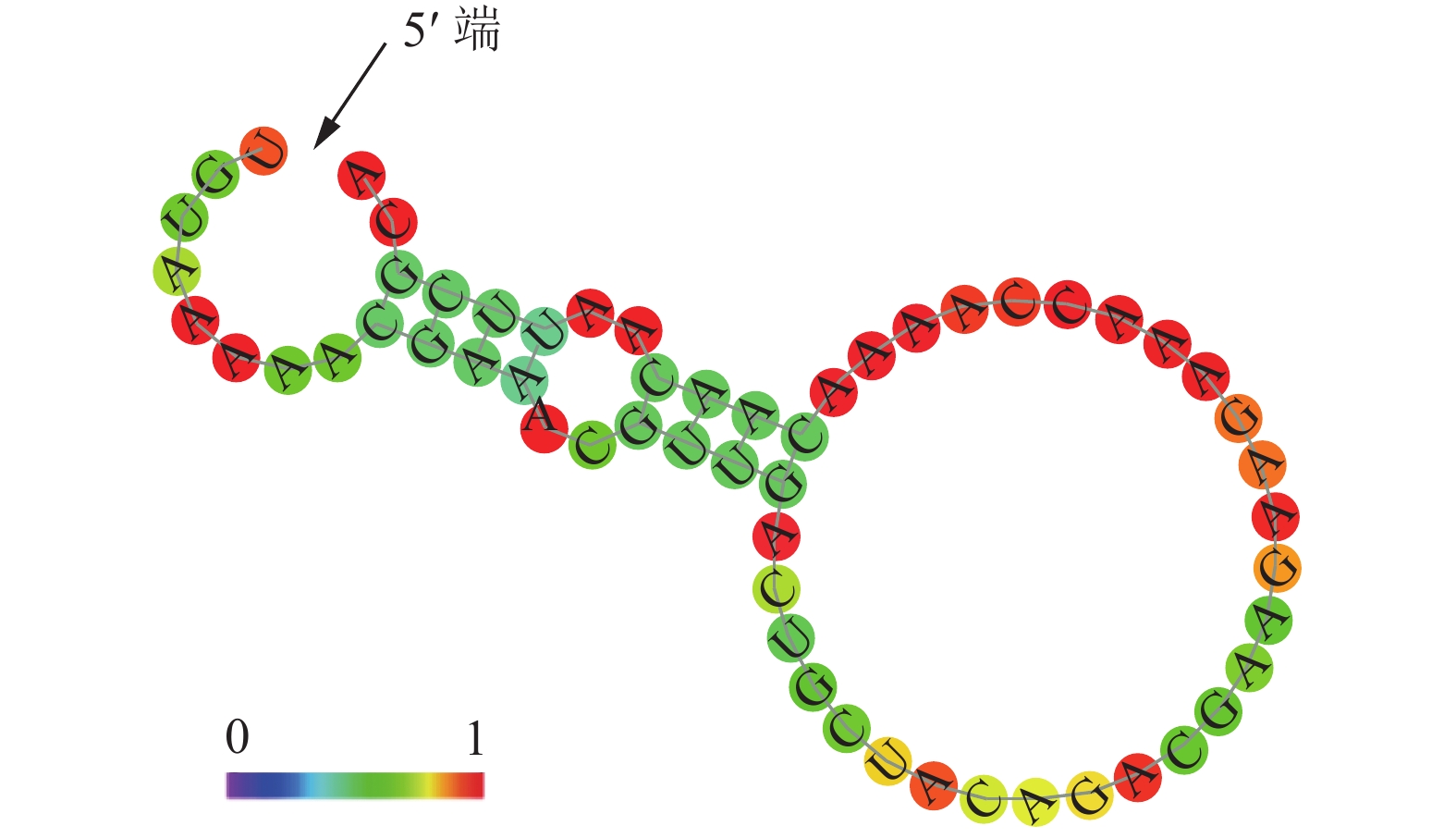
基于自由能最小的预测算法,通过在线软件RNAfold Web Sever分析重组病毒Leader RNA的二级结构。箭头所示为RNA的5'端,颜色越偏向红色,代表对应处碱基处于配对的概率越大;颜色越偏向紫色,代表对应处碱基处于配对的概率越小,结果如图4所示。由图4可见,rHep-Flury与Hep-dG具有完全相同的Leader RNA序列和二级结构,其最小自由能为–13.02 kJ·mol-1。说明2株RABV毒株的Leader RNA在结构上没有差异,IFN-β表达量的增加是由于Leader RNA的转录水平提高而引起。
2.5 G、M蛋白真核表达对NA细胞中IFN-β转录的影响
为了研究G蛋白和M蛋白本身的过表达对IFN-β转录的影响,首先摸索pH-G和pH-M质粒转染细胞后表达的最佳时间点。分别将5.0 μg的pH-G和pH-M重组表达质粒转染NA细胞,转染相同质量的pH-GFP质粒作为阳性对照,设空白细胞作为阴性对照。结果显示,在转染后24、48和72 h,pH-G、pH-M和pH-GFP质粒在NA细胞上均有效表达,48 h的荧光强度最大(图5a)。依据试验摸索的最佳时间,将pH-G和pH-M质粒以5.0和2.5 μg分别转染NA细胞,设空白细胞为阴性对照、空载体转染的细胞为阳性对照,在转染后48 h,提取细胞RNA,用荧光定量PCR检测IFN-β的转录水平。结果显示,与空白细胞比较,5 μg的pH-G质粒转染能刺激IFN-β的转录(P<0.05);而转染pH-M质粒均不能刺激IFN-β的转录(图5b)。说明G蛋白真核表达能在一定程度上刺激IFN-β的转录,这种刺激不依赖于RIG-I的识别,可能对Hep-dG激活干扰素通路起到一定的作用。
2.6 阻断IFN-β对重组狂犬病病毒复制的影响
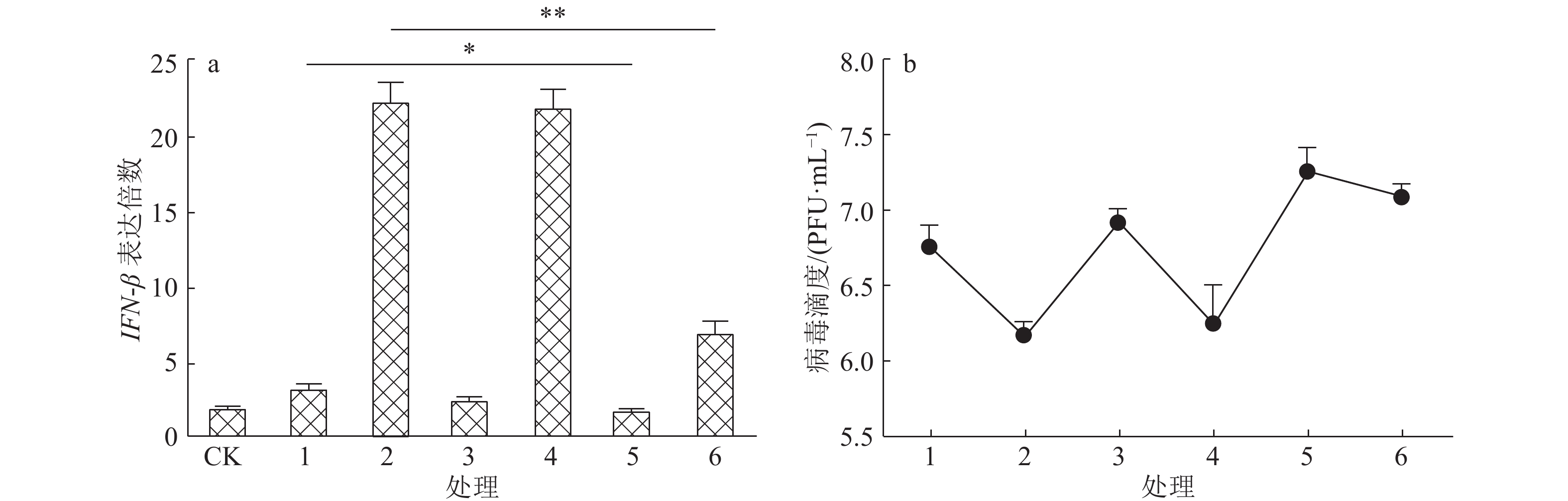
为了明确IFN-β对重组狂犬病毒复制的影响,我们用IFN-β特异性中和抗体(质量浓度为10 mg·L–1)处理NA细胞以阻断IFN信号通路,同时将正常鼠源IgG阴性血清处理的细胞和不做处理的细胞作为对照。结果(图6a)显示,IFN-β特异性中和抗体能阻断大部分β干扰素的转录。在阻断抗体处理后,rHep-Flury与Hep-dG感染的NA细胞中IFN-β基因的表达均显著下调,其中rHep-Flury阻断组比正常感染组下调了1.8倍(P<0.05),而Hep-dG阻断组比正常感染组下调了3.2倍(P<0.01)。在接毒后48 h,收集细胞上清液,检测抗体处理组和未处理组2株RABV的病毒滴度。结果(图6b)表明,IFN-β抗体处理对NA细胞培养上清液中rHep-Flury的子代病毒并无显著性影响,而Hep-dG的病毒滴度显著上调(P<0.01),约为阻断前的7.9倍。阻断后,Hep-dG的病毒滴度与亲代毒株rHep-Flury没有显著性差异。正常鼠源IgG阴性血清处理对2个感染组的病毒滴度均没有显著影响。说明NA细胞中IFN通路的激活可能是Hep-dG滴度下降的主要原因之一。
![]() 图 6 阻断IFN-β通路对狂犬病病毒复制的影响a.抗体阻断IFN-β的表达,CK:空白对照,1:rHep-Flury,2:Hep-dG,3:rHep-Flury+IgG,4:Hep-dG+IgG,5:rHep-Flury+Anti,6:Hep-dG+Anti,图中“*”和“**”分别表示直线两端所对应的样本达0.05和0.01的显著水平(t检验);b.病毒滴度的检测,1:rHep-Flury,2:Hep-dG,3:rHep-Flury+IgG,4:Hep-dG+IgG,5:rHep-Flury+Anti,6:Hep-dG+AntiFigure 6. Effect of blocking IFN-β pathway on rabies virus replication
图 6 阻断IFN-β通路对狂犬病病毒复制的影响a.抗体阻断IFN-β的表达,CK:空白对照,1:rHep-Flury,2:Hep-dG,3:rHep-Flury+IgG,4:Hep-dG+IgG,5:rHep-Flury+Anti,6:Hep-dG+Anti,图中“*”和“**”分别表示直线两端所对应的样本达0.05和0.01的显著水平(t检验);b.病毒滴度的检测,1:rHep-Flury,2:Hep-dG,3:rHep-Flury+IgG,4:Hep-dG+IgG,5:rHep-Flury+Anti,6:Hep-dG+AntiFigure 6. Effect of blocking IFN-β pathway on rabies virus replication3. 讨论与结论
本实验室前期的研究发现携带双G基因的重组狂犬病病毒(RABV)Hep-dG感染NA细胞后滴度明显低于亲代毒株rHep-Flury,而在BHK-21细胞中没有相同现象。前期的蛋白质组研究表明,RABV弱毒病毒G蛋白的过表达与干扰素的诱导呈高度的正相关[11]。本研究检测了不同时间点IFN-β mRNA的表达和干扰素下游因子STAT1的表达和磷酸化水平,结果发现,Hep-dG在感染NA细胞后,能显著上调IFN-β基因的表达,并激活下游因子STAT1的表达和磷酸化。由于BHK-21是RIG-I介导IFN通路的缺陷型细胞[12],推测2株重组RABV在BHK-21细胞和NA细胞中的病毒滴度差异可能与干扰素通路的激活有关。
RABV感染细胞后,为了逃逸干扰素的作用,通常会采取2种策略:一是通过聚合酶“加帽”[15],或在入侵细胞后基因组RNA始终被N蛋白缠绕,致使病毒核酸无法释放,从而逃逸PRRs的识别和结合[16-17];二是通过病毒P蛋白抑制IRF-3的磷酸化,拮抗干扰素的产生[18-20],或阻止STAT1的入核,从而抑制干扰素刺激基因的表达[21-22]。然而,RABV转录第一步生成的Leader RNA为5'端三磷酸化、不加帽、不加尾的58 nt单链RNA,能被RIG-I特异性识别并诱导IFN-β的释放[23-25]。Yang等[13]证明,抑制G蛋白表达量可降低RABV的入侵能力,因其减少了RABV Leader RNA的复制量,从而避免了DCs的激活而逃逸了宿主的免疫识别。本研究检测了不同感染复数(MOI)下RABV的吸附和入侵能力,结果发现,当MOI为0.10和1.00时,Hep-dG对NA细胞的吸附能力和入侵能力均明显高于亲代毒株rHep-Flury,说明G蛋白的过表达显著提高了RABV对神经细胞的吸附和穿入能力。然而,与Yang等[13]的推测不同,当MOI为0.01时,Hep-dG进入NA细胞的能力与亲代毒株rHep-Flury无显著性差异,Leader RNA表达量却显著高于rHep-Flury,说明G蛋白的过表达通过其他机制促进了RABV Leader RNA的复制量。此外,检测病毒基因组vRNA和RIG-I基因的表达水平发现,在感染36 h前,细胞内Leader RNA与病毒基因组vRNA的含量趋势一致;在感染36 h后,NA细胞内vRNA的含量继续升高,而Leader RNA的表达水平开始下降。在接毒后36和48 h,Hep-dG感染的NA细胞中RIG-I mRNA的含量显著高于亲代毒株rHep-Flury。细胞内RIG-I的变化趋势与Leader RNA一致,且都与IFN-β mRNA的表达高度一致。由于,rHep-Flury与Hep-dG具有完全相同的Leader RNA序列和二级结构,说明过表达G蛋白通过促进RABV Leader RNA的转录水平,从而激活了RIG-I介导的I型干扰素的激活。
Yang等[11]前期研究发现,G蛋白的过表达也促进了M蛋白的表达。为了证明病毒蛋白本身过表达对干扰素通路的影响,本研究将pH-G和pH-M质粒以5.0和2.5 μg分别转染6孔板NA细胞。结果发现,5 μg的pH-G质粒转染能刺激IFN-β的转录,而5.0和2.5 μg的pH-M质粒均不能刺激IFN-β的转录。说明G蛋白的真核表达在一定程度上也刺激了IFN-β的转录,可能对Hep-dG激活干扰素通路起到一定的作用,而具体作用的机制需要进一步研究。IFN-β特异性抗体处理NA细胞后,rHep-Flury的繁殖无显著影响,而Hep-dG的病毒滴度显著上升,且滴度与亲代毒株rHep-Flury无显著差异。说明NA细胞中干扰素通路的激活是Hep-dG滴度下降的主要原因之一。
RABV G蛋白的过表达通过促进RABV Leader RNA的转录,激活了RIG-I介导的IFN-β通路,进而抑制了Hep-dG在NA细胞的繁殖。此外,G蛋白本身的过表达也对干扰素通路起到一定的作用。本研究初步揭示了G蛋白激活先天性免疫应答的原因和作用,为RABV致病机制的研究奠定了基础。
-
图 6 阻断IFN-β通路对狂犬病病毒复制的影响
a.抗体阻断IFN-β的表达,CK:空白对照,1:rHep-Flury,2:Hep-dG,3:rHep-Flury+IgG,4:Hep-dG+IgG,5:rHep-Flury+Anti,6:Hep-dG+Anti,图中“*”和“**”分别表示直线两端所对应的样本达0.05和0.01的显著水平(t检验);b.病毒滴度的检测,1:rHep-Flury,2:Hep-dG,3:rHep-Flury+IgG,4:Hep-dG+IgG,5:rHep-Flury+Anti,6:Hep-dG+Anti
Figure 6. Effect of blocking IFN-β pathway on rabies virus replication
表 1 荧光定量PCR检测引物
Table 1 Primers used for fluorescence quantitative PCR detection
被检基因 引物序列 引物名称 产物长度/bp Leader RNA Leader-F 5'-CCAGATGCTTGGCGTCCT-3' 67 Leader-R 5'-ACGCTTAACAACAAAACC-3' vRNA vRNA-F 5'-GGAAAAGGGACATTTGAAAGAA-3' 130 vRNA-R 5'-AGTCCTCGTCATCGGAGTTGAC-3' RIG-I RIGI-F 5'-GCAAGTGCTTCCTCCTGACC-3' 142 RIGI-R 5'-ATGCGGTGAACCGTCTTTCC-3' GAPDH GAPDH-F 5'-AGAGTGTTTCCTCGTCCCGT-3' 199 GAPDH-R 5'-CTGTGCCGTTGAATTTGCCG-3' -
[1] 王朝, 周明, 傅振芳, 等. 狂犬病病毒逃逸宿主天然免疫反应的研究进展[J]. 生命科学, 2017(3): 237-244. [2] 张旭, 纪森林, 赵雯, 等. 狂犬病病毒致病机制的最新研究进展[J]. 中国兽医科学, 2018, 48(3): 295-304. [3] MEBATSION T, KONIG M, CONZELMANN K K. Budding of rabies virus particles in the absence of the spike glycoprotein[J]. Cell, 1996, 84(6): 941-951.
[4] MEBATSION T, WEILAND F, CONZELMANN K K. Matrix protein of rabies virus is responsible for the assembly and budding of bullet-shaped particles and interacts with the transmembrane spike glycoprotein G[J]. J Virol, 1999, 73(1): 242-250.
[5] MACFARLAN R I, DIETZSCHOLD B, WIKTOR T J, et al. T cell responses to cleaved rabies virus glycoprotein and to synthetic peptides[J]. J Immunol, 1984, 133(5): 2748-2752.
[6] 汪孟航, 朱洪伟, 何民辉, 等. 狂犬病病毒糖蛋白生物学功能研究进展[J]. 中国畜牧兽医, 2016(12): 3349-3355. [7] FISHER C R, STREICKER D G, SCHNELL M J. The spread and evolution of rabies virus: Conquering new frontiers[J]. Nat Rev Microbiol, 2018, 16(4): 241-255.
[8] LIU X, YANG Y, SUN Z, et al. A recombinant rabies virus encoding two copies of the glycoprotein gene confers protection in dogs against a virulent challenge[J]. PLoS One, 2014, 9(2): e87105.
[9] PREHAUD C, LAY S, DIETZSCHOLD B, et al. Glycoprotein of nonpathogenic rabies viruses is a key determinant of human cell apoptosis[J]. J Virol, 2003, 77(19): 10537-10547.
[10] FABER M, PULMANAUSAHAKUL R, HODAWADEKAR S S, et al. Overexpression of the rabies virus glycoprotein results in enhancement of apoptosis and antiviral immune response[J]. J Virol, 2002, 76(7): 3374-3381.
[11] YANG Y, LIU W, YAN G, et al. iTRAQ protein profile analysis of neuroblastoma (NA) cells infected with the rabies viruses rHep-Flury and Hep-dG[J/OL]. Front Microbiol, 2015, 6: 691. doi: 10.3389/fmicb.2015.00691.
[12] HABJAN M, PENSKI N, SPIEGEL M, et al. T7 RNA polymerase-dependent and -independent systems for cDNA-based rescue of Rift Valley fever virus[J]. J Gen Virol, 2008, 89(9): 2157-2166.
[13] YANG Y, HUANG Y, GNANADURAI C W, et al. The inability of wild-type rabies virus to activate dendritic cells is dependent on the glycoprotein and correlates with its low level of the de novo-synthesized leader RNA[J]. J Virol, 2015, 89(4): 2157-2169.
[14] WU J, MA C, WANG H, et al. A MyD88-JAK1-STAT1 complex directly induces SOCS-1 expression in macrophages infected with Group A Streptococcus[J]. Cell Mol Immunol, 2015, 12(3): 373-383.
[15] OGINO T, YADAV S P, BANERJEE A K. Histidine-mediated RNA transfer to GDP for unique mRNA capping by vesicular stomatitis virus RNA polymerase[J]. Proc Natl Acad Sci USA, 2010, 107(8): 3463-3468.
[16] GERLIER D, LYLES D S. Interplay between innate immunity and negative-strand RNA viruses: Towards a rational model[J]. Microbiol Mol Biol Rev, 2011, 75(3): 468-490.
[17] MOYER S A, ABRAHAM G, ADLER R, et al. Methylated and blocked 5' termini in vesicular stomatitis virus in vivo mRNAs[J]. Cell, 1975, 5(1): 59-67.
[18] BRZOZKA K, FINKE S, CONZELMANN K K. Identification of the rabies virus alpha/beta interferon antagonist: Phosphoprotein P interferes with phosphorylation of interferon regulatory factor 3[J]. J Virol, 2005, 79(12): 7673-7681.
[19] RIEDER M, CONZELMANN K K. Interferon in rabies virus infection[J]. Adv Virus Res, 2011, 79: 91-114.
[20] 王林栋, 张守峰, 刘晔, 等. 基于RIG-I的狂犬病病毒免疫逃逸机制[J]. 中国生物制品学杂志, 2013(10): 1517-1521. [21] VIDY A, CHELBI-ALIX M, BLONDEL D. Rabies virus P protein interacts with STAT1 and inhibits interferon signal transduction pathways[J]. J Virol, 2005, 79(22): 14411-14420.
[22] VIDY A, EL B J, CHELBI-ALIX M K, et al. The nucleocytoplasmic rabies virus P protein counteracts interferon signaling by inhibiting both nuclear accumulation and DNA binding of STAT1[J]. J Virol, 2007, 81(8): 4255-4263.
[23] PLUMET S, HERSCHKE F, BOURHIS J M, et al. Cytosolic 5'-triphosphate ended viral leader transcript of measles virus as activator of the RIG I-mediated interferon response[J]. PLoS One, 2007, 2(3): e279.
[24] FAUL E J, WANJALLA C N, SUTHAR M S, et al. Rabies virus infection induces type I interferon production in an IPS-1 dependent manner while dendritic cell activation relies on IFNAR signaling[J]. PLoS Pathog, 2010, 6(7): e1001016.
[25] KATZ I, GUEDES F, FERNANDES E R, et al. Immunological aspects of rabies: A literature review[J]. Arch Virol, 2017, 162(11): 3251-3268.



 下载:
下载: